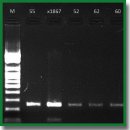
Секвенирование участка VP1–2A генома для субтипирования вируса гепатита A
Цель исследования — совершенствование методики секвенирования участка VP1–2A генома для молекулярно-генетического типирования вируса гепатита А.
Материалы и методы. Экстракцию нуклеиновых кислот проводили с помощью коммерческих наборов выделения нуклеиновых кислот и обратной транскрипции в ручном и автоматическом вариантах. Подбор последовательностей праймеров осуществляли с помощью программного обеспечения DNASTAR. Олигонуклеотиды синтезировали амидофосфитным методом с последующей очисткой электрофорезом в полиакриламидном геле. Искомые фрагменты генома получены методом ПЦР. Для приготовления реакционной смеси использовали коммерческие компоненты. Учет результатов проводили методом электрофореза в агарозном геле с окрашиванием ДНК бромидом этидия. Фрагменты ДНК после амплификации очищали методом адсорбции на колонках с диоксидом кремния. Определение последовательности очищенных фрагментов выполняли методом секвенирования по Сэнгеру. Полученные последовательности фрагмента VP1–2A анализировали в программном пакете MEGA 6. Оптимизированная методика секвенирования участка VP1–2A генома возбудителя апробирована на 171 образце клинического материала и проб внешней среды, содержащих РНК вируса гепатита А.
Результаты. Методика секвенирования оптимизирована на этапе амплификации вариабельного участка VP1–2A структурной части генома возбудителя, для чего были созданы авторские последовательности праймеров и подобраны условия полимеразной реакции (длина, локализация и температура отжига, концентрация праймеров и матрицы). Искомый фрагмент длиной 249 пар нуклеотидов находился в регионе 2988—3237 пар нуклеотидов согласно референсному штамму HM175. С помощью оптимизированной методики секвенирования фрагмента VP1–2A генома возбудителя в Н. Новгороде за период 2000–2017 гг. в 76 пробах из 171 образца, содержащего РНК возбудителя, установлен генотип вируса гепатита А. Выявлено преобладание возбудителя субтипа IA — 97% (74 из 76 образцов), и только 2 образца принадлежали субтипу IB.
Заключение. Оптимизированная методика секвенирования на этапе амплификации участка VP1–2A генома вируса гепатита А позволяет определять генотипическую структуру циркулирующих штаммов вируса, идентифицировать завозные штаммы возбудителя и устанавливать эпидемиологическую связь между различными случаями заболевания.
- Vaz J., Floyd C., Mason B., Shankar A.G., Lewis H. Control of a community outbreak of hepatitis A in an area of low endemicity, Wales, 2016. Hum Vaccin Immunother 2017; 13(10): 2352–2356, https://doi.org/10.1080/21645515.2017.1347242.
- Michaelis K., Wenzel J.J., Stark K., Faber M. Hepatitis A virus infections and outbreaks in asylum seekers arriving to Germany, September 2015 to March 2016. Emerg Microbes Infect 2017; 6(4): e26, https://doi.org/10.1038/emi.2017.11.
- Gallone M.F., Desiante F., Gallone M.S., Barbuti G., Tafuri S., Germinario C. Serosurveillance of hepatitis A in a region which adopted the universal mass vaccination. Medicine (Baltimore) 2017; 96(9): e5884, https://doi.org/10.1097/md.0000000000005884.
- Гепатит А: этиология, эпидемиология, диагностика, профилактика. Под ред. В.В. Шкарина. Н. Новгород: НижГМА; 2015.
- Вирусные гепатиты в Российской Федерации: аналитический обзор. Под ред. Покровского В.И., Тотоляна А.А. СПб: ФБУН НИИЭМ им. Пастера; 2016.
- Кареткина Г.Н. Вирусный гепатит А в прошлом, настоящем и будущем. Инфекционные болезни: новости, мнения, обучение 2014; 3(8): 38–48.
- Мукомолов С.Л., Михайлов М.И., Семененко Т.А., Селькова Е.П., Герасимова И.Е. Бремя гепатита А в Российской Федерации: научный обзор. Эпидемиология и вакцинопрофилактика 2014; 6(79): 24–34.
- Чернов А.В., Брусенская Т.Ю., Корчагин В.В. Анализ заболеваемости острыми вирусными гепатитами по Воронежской области. Территория инноваций 2017; 4(8): 117–120.
- WHO position paper on hepatitis A vaccines. Wkly Epidemiol Rec 2012; 87(28/29): 261–276. URL: http://www.who.int/wer/2012/wer8728_29/en/.
- Карандашова И.В., Пименов Н.Н., Неверов А.Д., Михайловская Г.В., Долгин В.А., Браславская С.И., Комарова С.В., Лыткина И.Н., Шулакова Н.И., Шипулин Г.А., Чуланов В.П. Молекулярно-эпидемиологические особенности вспышки гепатита А в Москве. Эпидемиология и инфекционные болезни. Актуальные вопросы 2012; 3: 9–14.
- Bondarenko T.Y., Ternovoi V.A., Netesov S.V. Hepatitis a virus: structure-functional features of genome, molecular diagnostics, and cultivation. Molecular Genetics, Microbiology and Virology 2013; 28(3): 99–109, https://doi.org/10.3103/s0891416813030038.
- Мукомолов C.Л., Калинина О.В. Молекулярная эпидемиология вирусных гепатитов. Мир вирусных гепатитов 2003; 11: 2–7.
- Чуланов В.П., Карандашова И.В., Пименов Н.Н., Молочный В.П., Томилка Г.С., Слепцова С.С., Семенова В.К., Малый В.П., Бойко В.В., Камолов Б.Д., Волчкова Е.В. Клиническое значение генетического разнообразия вируса гепатита А. Эпидемиология и инфекционные болезни 2014; 19(4): 12–17.
- Nainan O.V., Xia G., Vaughan G., Margolis H.S. Diagnosis of hepatitis A virus infection: a molecular approach. Clin Microbiol Rev 2006; 9(1): 63–79, https://doi.org/10.1128/cmr.19.1.63-79.2006.
- Vaughan G., Goncalves Rossi L.M., Forbi J.C., de Paula V.S., Purdy M.A., Xia G., Khudyakov Y.E. Hepatitis A virus: host interactions, molecular epidemiology and evolution. Infect Genet Evol 2014; 21: 227–243, https://doi.org/10.1016/j.meegid.2013.10.023.
- Costa-Mattioli M., Di Napoli A., Ferré V., Billaudel S., Perez-Bercoff R., Cristina J. Genetic variability of hepatitis A virus. J Gen Virol 2003; 84(Pt 12): 3191–3201, https://doi.org/10.1099/vir.0.19532-0.
- Sánchez G., Bosch A., Pintó R.M. Genome variability and capsid structural constraints of hepatitis A virus. J Virol 2003; 77(1): 452–459, https://doi.org/10.1128/jvi.77.1.452-459.2003.
- Robertson B.H., Jansen R.W., Khanna B. Genetic relatedness of hepatitis A virus strains recovered from different geographical regions. J Gen Virol 1992; 73 (Pt 6): 1365–1677, https://doi.org/10.1099/0022-1317-73-6-1365.
- Mukomolov S., Kontio M., Zheleznova N., Jokinen S., Sinayskaya E., Stalevskaya A., Davidkin I. Increased circulation of hepatitis A virus genotype IIIA over the last decade in St. Petersburg, Russia. J Med Virol 2012; 84(10): 1528–1534, https://doi.org/10.1002/jmv.23378.
- Bruni R., Taffon S., Equestre M., Cella E., Lo Presti A., Costantino A., Chionne P., Madonna E., Golkocheva-Markova E., Bankova D., Ciccozzi M., Teoharov P., Ciccaglione A.R. Hepatitis A virus genotypes and strains from an endemic area of Europe, Bulgaria 2012–2014. BMC Infect Dis 2017; 17(1): 497, https://doi.org/10.1186/s12879-017-2596-1.










