Компьютерная программа для выявления и анализа поринзависимой антибиотикорезистентности бактерий
Цель исследования — разработать новую компьютерную программу для выявления мутаций в генах, детерминирующих неплазмидную антибиотикорезистентность у грамотрицательных бактерий, и оценить ее возможности на примере выявления поринзависимой устойчивости к карбапенемам клинических изолятов Pseudomonas aeruginosa.
Материалы и методы. Алгоритм работы программы включает поиск соответствия между референсными и изучаемыми генами. При обнаружении в анализируемом геноме искомой нуклеотидной последовательности она сравнивается с референсной и анализируется. Полученные данные подтверждаются сравнением аминокислотных последовательностей, кодируемых референсными и изучаемыми генами. В качестве референсных нуклеотидных последовательностей генов поринов, обеспечивающих транспорт карбапенемов внутрь бактериальной клетки (OprD, OpdD и OpdP), были использованы гены чувствительного штамма P. aeruginosa ATCC 27853, а для проверки возможностей предлагаемой программы — полные геномы клинических изолятов P. aeruginosa из базы данных PATRIC 3.6.9 и собственной коллекции ДНК клинических штаммов. Проанализированные штаммы были охарактеризованы фенотипически согласно критериям стандарта CLSI. Поиск генов карбапенемаз в исследованных геномах P. aeruginosa проведен с использованием ресурса ResFinder 4.1.
Результаты. Разработанная компьютерная программа для определения генетических детерминант неплазмидной антибиотикорезистентности позволила выявить в генах поринов клинических изолятов P. aeruginosa различные по типу и значимости мутации, приводящие к модификациям пептидной структуры продукта. В поринах OpdD и OpdP карбапенем-чувствительных и карбапенем-нечувствительных изолятов преобладали единичные аминокислотные замены. Ген порина OprD карбапенем-нечувствительных штаммов характеризовался большим разнообразием модификаций, включая инсерции и/или делеции, приводящие к преждевременной остановке синтеза порина. У нескольких нечувствительных к меропенему изолятов не выявлено мутаций генов в OprD, что может быть связано с наличием альтернативных механизмов устойчивости.
Заключение. Предлагаемый программный продукт способен стать эффективным инструментом расшифровки молекулярно-генетических механизмов хромосомной устойчивости бактерий к антибиотикам. Результаты апробации программы показали различия между распространенностью значимых для возникновения карбапенемрезистентности мутаций в генах oprD, opdD и opdP.
Введение
Глобально распространившаяся устойчивость к антимикробным препаратам, или антимикробная резистентность (АМР), стала одной из важнейших проблем современного здравоохранения [1]. Ситуация усугубляется тем, что в ближайшее время ожидается резкий скачок распространения АМР вследствие масштабного увеличения употребления антибиотиков, связанного с пандемией COVID-19 [2, 3]. Это ставит перед медицинской наукой задачу совершенствования методов оценки антибиотикорезистентности и расшифровки ее механизмов. На современном этапе развития медицины фенотипическое определение АМР является необходимым, но недостаточным для успешного противодействия резистентности, фенотипический профиль резистентных бактерий должен быть дополнен описанием механизмов ее формирования [4].
К числу наиболее информативных способов определения механизмов АМР относятся методы выявления генетических детерминант резистентности. Основные детерминанты АМР традиционно подразделяются на плазмидные и хромосомные (хромосомальные). Плазмидные гены резистентности — это гены, которые могут передаваться от клетки к клетке горизонтальным путем, они могут быть локализованы как в плазмиде, так и в хромосоме [5–7]. Хромосомная резистентность связана с мутациями (точечные мутации, небольшие инсерции/делеции, обширные инсерции/делеции, в том числе вставки мобильных генетических элементов) в хромосомных генах, являющихся атрибутами данного вида бактерий [8–10].
Для поиска плазмидных генов АМР созданы и успешно применяются программные онлайн-инструменты или программы, требующие локально установленной (standalone) версии. К их числу принадлежат ResFinder, CARD, ARDB, ARG-ANNOT и другие инструменты [11–14]. Они в первую очередь нацелены на поиск генов, передающихся в составе мобильных генетических элементов, например генов бета-лактамаз (устойчивость к бета-лактамным антибиотикам), аминогликозид-трансфераз (устойчивость к аминогликозидам и фторхинолонам), хлорамфеникол-ацетилтрансфераз (устойчивость к хлорамфениколу), глутатион-S-трансфераз (устойчивость к фосфомицину), генов белков рибосомальной защиты (устойчивость к тетрациклину) и других детерминант резистентности. Некоторые из существующих инструментов реализуют функцию поиска хромосомных детерминант резистентности (PointFinder, CARD).
К сожалению, функциональные возможности перечисленных инструментов не всегда позволяют выявить и проанализировать важные генетические детерминанты АМР, связанные с изменением поринов, отвечающих за проникновение антибиотиков в клетку, регуляторных генов глобальных эффлюкс-систем и генов, кодирующих мишени антибиотиков. В частности, это относится к расшифровке молекулярных механизмов устойчивости к антибиотикам последних поколений, включая карбапенемы.
Цель настоящей работы — разработать новый программный инструмент для определения мутаций генов, детерминирующих поринзависимую антимикробную резистентность у грамотрицательных бактерий, и продемонстрировать возможности этой программы на примере выявления поринзависимой устойчивости к карбапенемам клинических изолятов P. aeruginosa.
Материалы и методы
Общий дизайн исследования включал:
1) создание базы референсных генов поринов, обеспечивающих транспорт антибиотиков через наружную мембрану внутрь периплазматического пространства;
2) получение нуклеотидных последовательностей генов поринов и аминокислотных последовательностей соответствующих им поринов изучаемых клинических изолятов (по данным об их полных геномах);
3) использование разработанного программного продукта для определения генетических детерминант неплазмидной антибиотикорезистентности [15], работающего по оригинальному алгоритму и выдающего заключение об отличиях анализируемой последовательности гена и его продукта (белка) от референсных последовательностей из базы данных; апробация работы программы на модели поринзависимой резистентности бактерий к меропенему и имипенему.
Программа включает базу референсных генов поринов, участвующих в формировании АМР у клинически значимых видов грамотрицательных бактерий, включая Pseudomonas aeruginosa, Klebsiella pneumoniae, Escherichia coli, Enterobacter spp., Salmonella enterica и др. Последовательности референсных генов были получены из базы данных NCBI (https://www.ncbi.nlm.nih.gov/nuccore/) путем выбора последовательностей генов поринов у штаммов, которые рекомендуются международными стандартами EUCAST (European Committee on Antimicrobial Susceptibility Testing) и CLSI (Clinical and Laboratory Standards Institute, США) в качестве стандартов чувствительности к антибиотикам. В настоящем исследовании были использованы чувствительный референс-штамм P. aeruginosa ATCC 27853 и нуклеотидные последовательности генов его поринов OprD/ОссD1, OpdD/OccK7 и OpdP/OccD3, участие которых в формировании резистентности к карбапенемам было подтверждено ранее (табл. 1).

|
Таблица 1. Порины Pseudomonas aeruginosa, участвующие в транспорте карбапенемов |
В качестве отрицательного контроля (штаммы, не имеющие видоспецифичных для P. aeruginosa поринов) были проанализированы 10 геномов изолятов из базы данных PATRIC 3.6.9 (https://www.patricbrc.org/) пяти разных видов: Acinetobacter baumannii (SP5515, GML-KP48-AB-TR), Klebsiella pneumoniae (AR0361, AR438), Escherichia coli (AR435, AR0450), Staphylococcus aureus (AR464, AR0216), Enterococcus faecium (VREN1530, VREN2775).
Для проверки возможностей предлагаемой программы были использованы полные геномы (полные сборки либо контиги) P. aeruginosa из двух источников. Первым источником стала база данных PATRIC 3.6.9, из которой были получены 15 геномов клинических штаммов P. aeruginosa и АМР-свойства этих изолятов. Вторым источником геномов (10 образцов) послужила собственная коллекция ДНК клинических штаммов, АМР-спектры (чувствительность к меропенему и имипинему) которых были предварительно охарактеризованы фенотипически согласно критериям стандарта CLSI, 2020 г. (https://clsi.org/standards/).
Поиск адаптивных генов карбапенемрезистентности (генов карбапенемаз) в исследованных геномах P. aeruginosa был проведен при помощи ресурса ResFinder 4.1 (https://cge.cbs.dtu.dk/services/ResFinder/).
Работа программы строится по следующему алгоритму (см. рисунок). На первом этапе выполняется поиск соответствия между референсными и изучаемыми генами с помощью программы BLAST на основе выбора best-matching [21]. При обнаружении в анализируемом геноме искомой нуклеотидной последовательности она сравнивается с референсной. В случае их различия данные анализируются программой и результат анализа автоматически выдается в текстовом либо табличном формате, отображающем обнаруженные изменения нуклеотидной последовательности гена. Значимость обнаруженных изменений нуклеотидной последовательности для возникновения АМР проверяется путем анализа синонимичности замен в аминокислотной последовательности порина. Анализ синонимичности выполняется автоматически, его алгоритм заложен в программном коде.
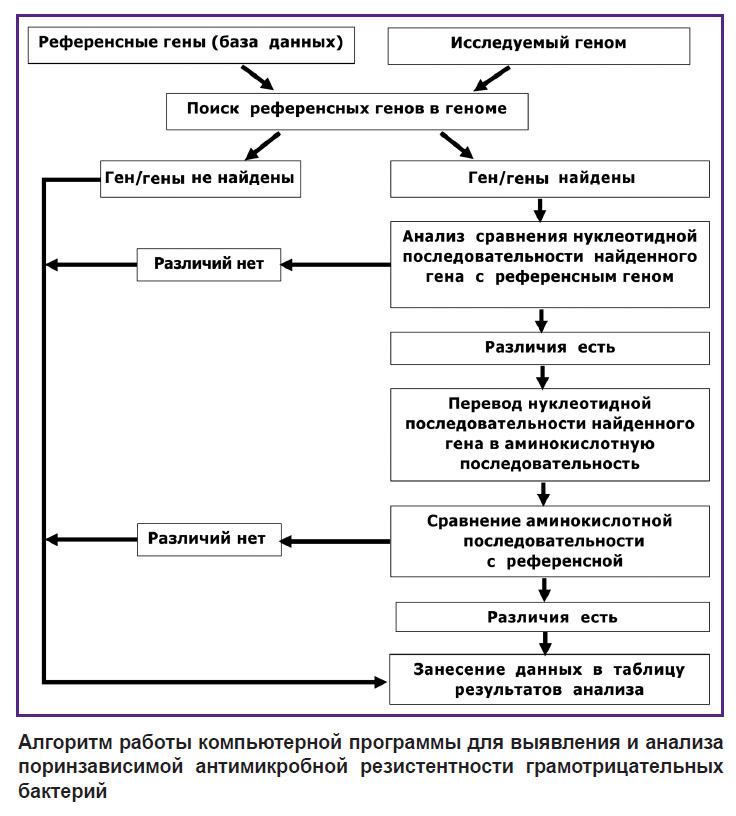
|
Алгоритм работы компьютерной программы для выявления и анализа поринзависимой антимикробной резистентности грамотрицательных бактерий |
Результаты
Во всех проанализированных геномах изолятов P. aeruginosa (n=25) были обнаружены гены поринов OprD, OpdD и OpdP. В геномах всех изолятов, не относящихся к виду P. aeruginosa (n=10) и использованных в качестве негативного контроля, гены выбранных поринов, специфичных для P. aeruginosa, не обнаружены.
В геномах исследованных 25 изолятов выявлены различные по типу и значимости мутации. В гене oprD детектированы мутации у 15 изолятов, в гене opdD — у 22 изолятов, в гене opdP — у 13 изолятов (табл. 2). Всего обнаружено 4 группы мутаций, приводящих к модификациям пептидной структуры порина.
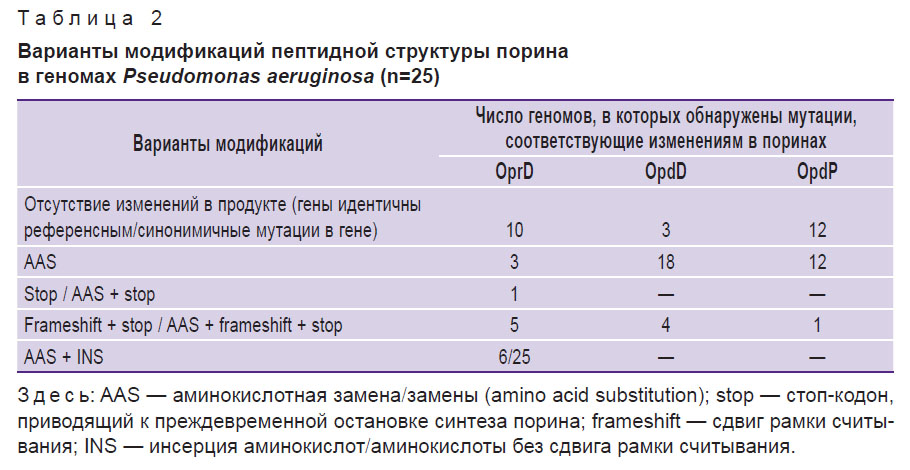
|
Таблица 2. Варианты модификаций пептидной структуры порина в геномах Pseudomonas aeruginosa (n=25) |
Наиболее частыми модификациями являлись аминокислотные замены в поринах OpdD и OpdP. Для поринов OprD и OpdD было характерно большее разнообразие модификаций, включая инсерции и/или делеции, сопровождающиеся появлением стоп-кодона.
Соответствие наличия мутаций в генах поринов либо наличия генов карбапенемаз с фенотипическим проявлением АМР (соотношение генотип/фенотип) оценивали исходя из участия поринов в транспорте антибиотиков через внешнюю мембрану (см. табл. 1; табл. 3). У 7 из 8 чувствительных к меропенему изолятов в поринах либо не наблюдалось аминокислотных замен (OprD и OpdP дикого типа), либо были выявлены от одной до четырех аминокислотных замен в OpdD и OpdP. Один чувствительный к меропенему изолят обладал мутантным порином OprD (16 аминокислотных замен). Наиболее частыми структурными мутациями в оprD у нечувствительных (резистентных и с промежуточным уровнем резистентности) к карбапенемам изолятов были замены аминокислот в сочетании с вставкой двух аминокислот (6/22) и мутацией сдвига рамки считывания, приводящей к преждевременной остановке синтеза порина (5/22). У 3 нечувствительных к меропенему изолятов мутаций в оprD не выявлено.
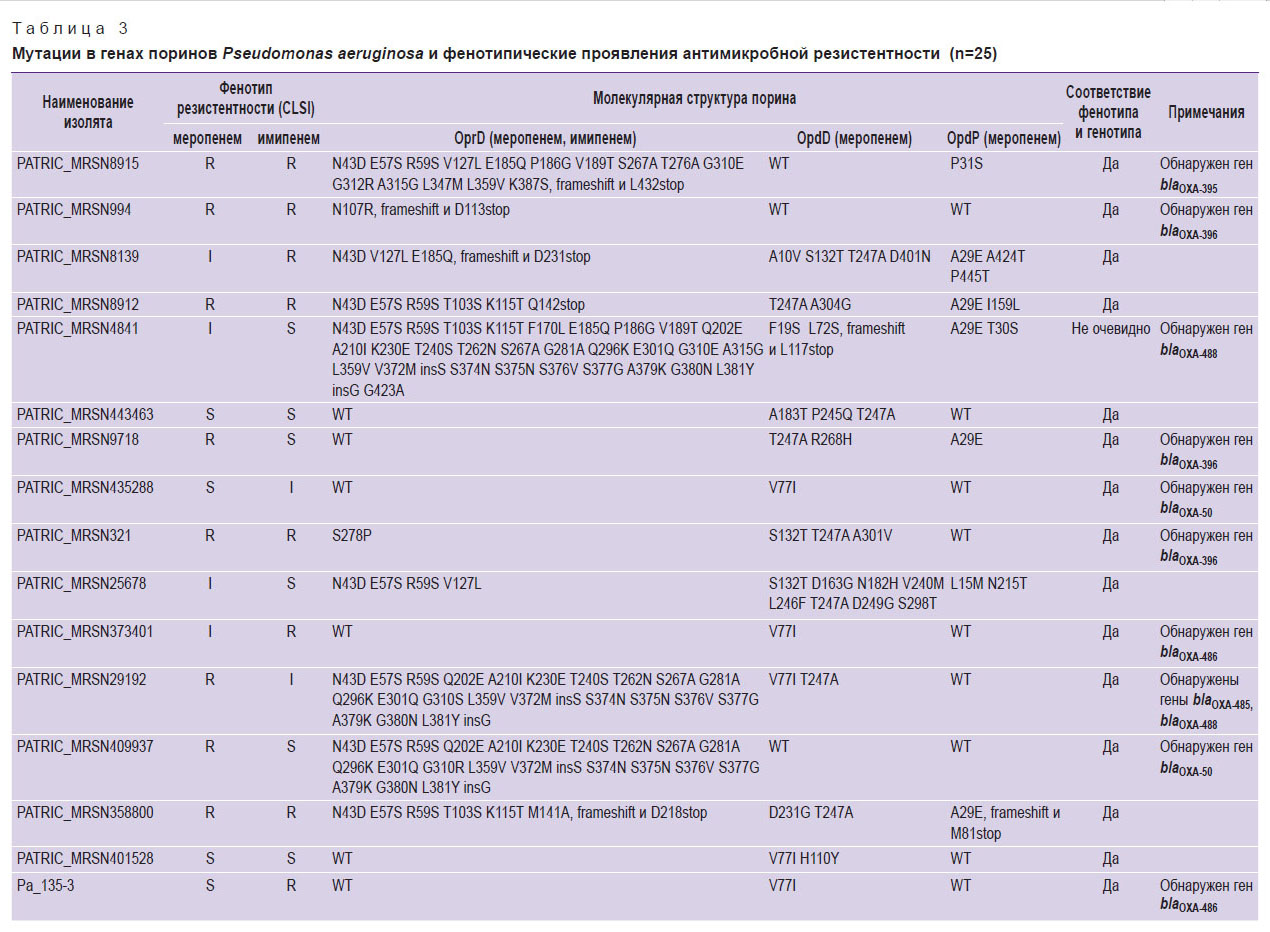
|
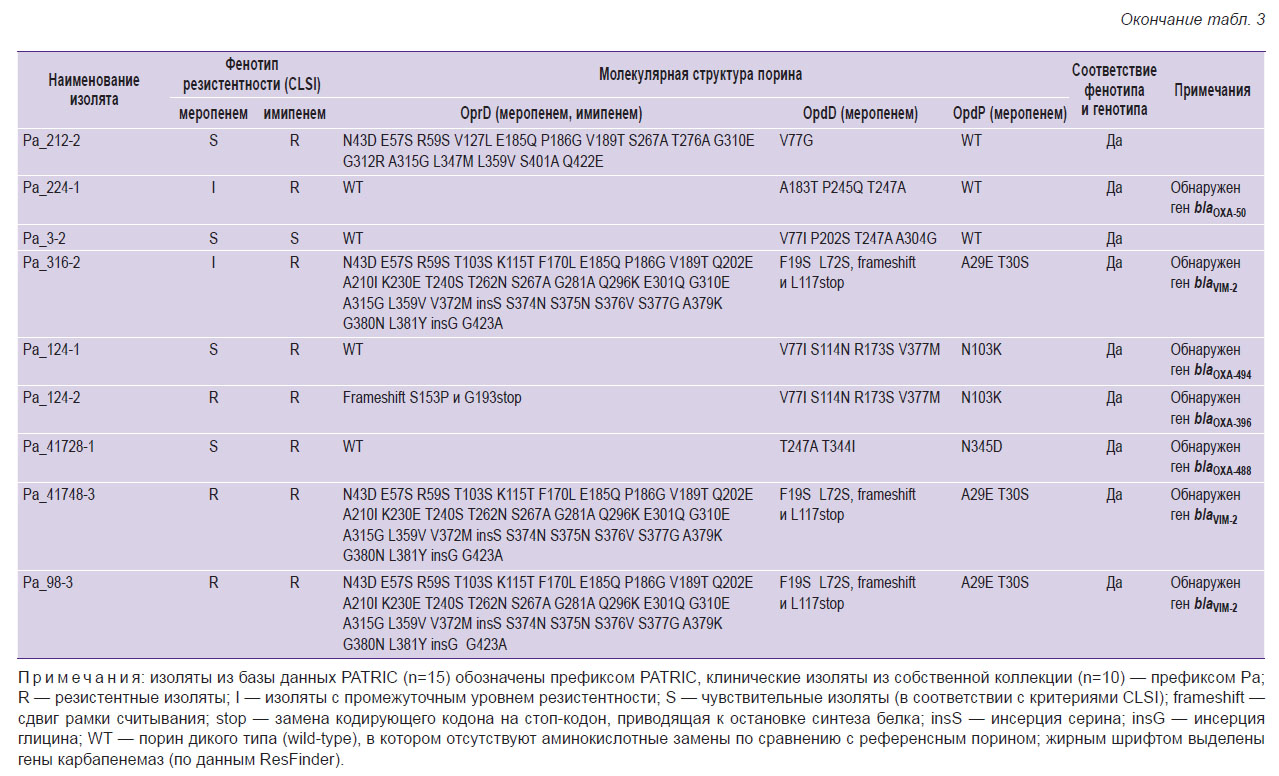
|
Таблица 3. Мутации в генах поринов Pseudomonas aeruginosa и фенотипические проявления антимикробной резистентности (n=25) |
В поринах OpdD и OpdP преобладали единичные замены аминокислот (1–8 AAS), причем такие паттерны наблюдались как у карбапенем-чувствительных, так и у карбапенем-нечувствительных изолятов. Делеция одного нуклеотида в гене opdD, приводящая к преждевременной остановке синтеза порина, присутствовала у 4 карбапенем-нечувствительных изолятов (у 3 — резистентных к меропенему и имипенему, у 1 — с промежуточным уровнем резистентности к меропенему). Потеря функционального порина OpdP в результате возникновения стоп-кодона в позиции, соответствующей 81-й аминокислоте белка, наблюдалась у 1 резистентного к меропенему и имипенему изолята.
Обсуждение
В большинстве случаев у госпитальных изолятов P. aeruginosa наблюдается сочетание механизмов резистентности к бета-лактамным антибиотикам, включая карбапенемы [22, 23]. Методы детекции резистентности, опосредованной бета-лактамазами, достаточно просты и хорошо реализуются при помощи рутинных лабораторных методов [24, 25]. В то же время анализ хромосомных детерминант резистентности связан с определенными трудностями с точки зрения биоинформатической обработки. В настоящей статье представлены результаты применения разработанной нами программы для определения генетических детерминант неплазмидной антибиотикорезистентности с целью поиска мутаций в генах поринов, транспортирующих карбапенемы внутрь бактериальной клетки (oprD, opdD, opdP), и анализа изменения аминокислотных последовательностей соответствующих протеинов как основы формирования карбапенемрезистентности.
Программный анализ 35 геномов (25 геномов P. aeruginosa и 10 геномов пяти других видов бактерий) позволил сделать несколько важных наблюдений относительно эффективности программы в поиске генов oprD, opdD и opdP и их мутаций. Выявление генов указанных поринов в геномах всех изолятов P. aeruginosa и их отсутствие в геномах A. baumannii, K. pneumoniae, E. coli, S. aureus, E. faecium свидетельствует о специфичности программного поиска целевых нуклеотидных последовательностей. Среди изолятов P. aeruginosa наиболее разнообразные изменения были обнаружены в гене оprD, что соответствует литературным данным о частой вовлеченности этого гена в формирование карбапенемрезистентного фенотипа у P. aeruginosa [26].
Программа позволила на практике выявить весь спектр теоретически возможных типов мутаций, включая мутации, ведущие к несинонимичным заменам аминокислот, нонсенс-мутации (ведущие к возникновению стоп-кодонов), инсерции и делеции, сдвиги рамки считывания.
Обнаружение резистентных к карбапенемам изолятов с OprD дикого типа можно объяснить с позиции альтернативных механизмов устойчивости и прежде всего — карбапенемаз групп OXA (см. табл. 3). Не исключена возможность гиперэкспрессии эффлюкс-систем и мутаций генов пенициллинсвязывающих белков [23]. Для более полной расшифровки генетических основ резистентного фенотипа необходим дополнительный анализ молекулярных механизмов устойчивости.
Интересно, что в генах oprD, opdD и opdP были обнаружены разные варианты мутаций, а также их разное число. Этот интригующий статистический результат трудно объяснить на современном этапе. Корректное объяснение данного феномена требует знаний о механизмах репарации ДНК в геномах P. aeruginosa, которые еще не получены.
Несколько штаммов сохраняли чувствительность к меропенему при наличии резистентности к имипенему. Для объяснения этого факта нужно обратить внимание на то, что меропенем имеет несколько альтернативных путей поступления в бактериальную клетку: порины OprD, OpdD и OpdP. В случае наиболее часто встречающихся мутаций гена oprD меропенем может транспортироваться через более устойчивые к мутациям порины OpdD и OpdP. Имипенем имеет только одни «входные ворота» — порин OprD, поэтому несоответствие между профилями карбапенемрезистентности штаммов P. aeruginosa выглядит весьма логичным.
Предлагаемый программный продукт может иметь ограничения, связанные с ошибками секвенирования и/или сборки генома, в этом случае программа выдает результат «Ген не найден».
В целом разработанный алгоритм позволяет осуществлять одновременный поиск детерминант резистентности в полногеномных последовательностях, количество которых лимитируется лишь возможностями используемого компьютера.
Заключение
Результаты апробации представленной новой компьютерной программы позволяют утверждать, что она может стать эффективным инструментом расшифровки молекулярно-генетических механизмов хромосомной устойчивости бактерий к антибиотикам. Ее использование позволило обнаружить различия между распространенностью значимых для возникновения карбапенемрезистентности мутаций в генах oprD, opdD и opdP. Гены oprD были более подвержены мутациям, гены opdP оказались консервативными. Из-за шунтированности поринзависимого транспорта меропенема выводы о резистентности штамма к этому антибиотику должны основываться не только на обнаружении поломок порина OprD, но и на наличии значимых мутаций в генах opdP и opdD. Меропенем, имея несколько путей для поступления в бактериальную клетку, является более перспективным антибиотиком в борьбе со штаммами P. aeruginosa, карбапенемрезистентность которых обусловлена нарушением пориновой проницаемости.
Благодарности. Авторы благодарят Центр высокоточного редактирования и генетических технологий для биомедицины Российского национального исследовательского медицинского университета им. Н.И. Пирогова за поддержку работы с методической частью исследования.
Финансирование исследования. Работа выполнена при поддержке гранта Российского научного фонда (проект №20-15-00235).
Конфликт интересов. Авторы заявляют об отсутствии конфликта интересов.
Литература
- Talebi Bezmin Abadi A., Rizvanov A.A., Haertlé T., Blatt N.L. World Health Organization report: current crisis of antibiotic resistance. Bionanoscience 2019; 9: 778–788, https://doi.org/10.1007/s12668-019-00658-4.
- Devi S. No time to lower the guard on AMR. Lancet Microbe 2020; 1(5): e198, https://doi.org/10.1016/s2666-5247(20)30129-4.
- Knight G.M., Glover R.E., McQuaid C.F., Olaru I.D., Gallandat K., Leclerc Q.J., Fuller N.M., Willcocks S.J., Hasan R., van Kleef E., Chandler C.I. Antimicrobial resistance and COVID-19: intersections and implications. Elife 2021; 10: e64139, https://doi.org/10.7554/elife.64139.
- World Health Organization. Global action plan on antimicrobial resistance. WHO, Library Cataloguing-in-Publication Data; 2015. URL: https://www.who.int/publications/i/item/9789241509763.
- Bennett P.M. Plasmid encoded antibiotic resistance: acquisition and transfer of antibiotic resistance genes in bacteria. Br J Pharmacol 2008; 153 Suppl 1(Suppl 1): S347–S357, https://doi.org/10.1038/sj.bjp.0707607.
- Partridge S.R. Analysis of antibiotic resistance regions in Gram-negative bacteria. FEMS Microbiol Rev 2011; 35(5): 820–855, https://doi.org/10.1111/j.1574-6976.2011.00277.x.
- van Hoek H.A.M., Mevius D., Guerra B., Mullany P., Roberts A.P., Aarts H.J. Acquired antibiotic resistance genes: an overview. Front Microbiol 2011; 2: 203, https://doi.org/10.3389/fmicb.2011.00203.
- Woodford N., Ellington M.J. The emergence of antibiotic resistance by mutation. Clin Microbiol Infect 2007; 13(1): 5–18, https://doi.org/10.1111/j.1469-0691.2006.01492.x.
- Sultan I., Rahman S., Jan A.T., Siddiqui M.T., Mondal A.H., Haq Q.M.R. Antibiotics, resistome and resistance mechanisms: a bacterial perspective. Front Microbiol 2018; 9: 2066, https://doi.org/10.3389/fmicb.2018.02066.
- López-Causapé C., Sommer L.M., Cabot G., Rubio R., Ocampo-Sosa A.A., Johansen H.K., Figuerola J., Cantón R., Kidd T.J., Molin S., Oliver A. Evolution of the Pseudomonas aeruginosa mutational resistome in an international cystic fibrosis clone. Sci Rep 2017; 7(1): 5555, https://doi.org/10.1038/s41598-017-05621-5.
- Bortolaia V., Kaas R.S., Ruppe E., Roberts M.C., Schwarz S., Cattoir V., Philippon A., Allesoe R.L., Rebelo A.R., Florensa A.F., Fagelhauer L., Chakraborty T., Neumann B., Werner G., Bender J.K., Stingl K., Nguyen M., Coppens J., Xavier B.B., Malhotra-Kumar S., Westh H., Pinholt M., Anjum M.F., Duggett N.A., Kempf I., Nykäsenoja S., Olkkola S., Wieczorek K., Amaro A., Clemente L., Mossong J., Losch S., Ragimbeau C., Lund O., Aarestrup F.M. ResFinder 4.0 for predictions of phenotypes from genotypes. J Antimicrob Chemother 2020; 75(12): 3491–3500, https://doi.org/10.1093/jac/dkaa345.
- Alcock B.P., Raphenya A.R., Lau T.T.Y., Tsang K.K., Bouchard M., Edalatmand A., Huynh W., Nguyen A.V., Cheng A.A., Liu S., Min S.Y., Miroshnichenko A., Tran H.K., Werfalli R.E., Nasir J.A., Oloni M., Speicher D.J., Florescu A., Singh B., Faltyn M., Hernandez-Koutoucheva A., Sharma A.N., Bordeleau E., Pawlowski A.C., Zubyk H.L., Dooley D., Griffiths E., Maguire F., Winsor G.L., Beiko R.G., Brinkman F.S.L., Hsiao W.W.L., Domselaar G.V., McArthur A.G. CARD 2020: antibiotic resistome surveillance with the comprehensive antibiotic resistance database. Nucleic Acids Res 2020; 48(D1): D517–D525, https://doi.org/10.1093/nar/gkz935.
- Liu B., Pop M. ARDB — Antibiotic Resistance Genes Database. Nucleic Acids Res 2009; 37(Database issue): D443–D447, https://doi.org/10.1093/nar/gkn656.
- Gupta S.K., Padmanabhan B.R., Diene S.M., Lopez-Rojas R., Kempf M., Landraud L., Rolain J.M. ARG-ANNOT, a new bioinformatic tool to discover antibiotic resistance genes in bacterial genomes. Antimicrob Agents Chemother 2014; 58(1): 212–220, https://doi.org/10.1128/aac.01310-13.
- Программа для ЭВМ «Компьютерная программа для определения генетических детерминант неплазмидной антибиотикорезистентности». Свидетельство Федеральной службы по интеллектуальной собственности (Роспатент) о государственной регистрации программы для ЭВМ №2021665744 от 01.10.2021 г.
- Trias J., Nikaido H. Outer membrane protein D2 catalyzes facilitated diffusion of carbapenems and penems through the outer membrane of Pseudomonas aeruginosa. Antimicrob Agents Chemother 1990; 34(1): 52–57, https://doi.org/10.1128/aac.34.1.52.
- Quinn J.P., Darzins A., Miyashiro D., Ripp S., Miller R.V. Imipenem resistance in Pseudomonas aeruginosa PAO: mapping of the OprD2 gene. Antimicrob Agents Chemother 1991; 35(4): 753–755, https://doi.org/10.1128/aac.35.4.753.
- Chevalier S., Bouffartigues E., Bodilis J., Maillot O., Lesouhaitier O., Feuilloley M.G.J., Orange N., Dufour A., Cornelis P. Structure, function and regulation of Pseudomonas aeruginosa porins. FEMS Microbiol Rev 2017; 41(5): 698–722, https://doi.org/10.1093/femsre/fux020.
- Sonnleitner E., Pusic P., Wolfinger M.T., Bläsi U. Distinctive regulation of carbapenem susceptibility in Pseudomonas aeruginosa by Hfq. Front Microbiol 2020; 11: 1001, https://doi.org/10.3389/fmicb.2020.01001.
- Soundararajan G., Bhamidimarri S.P., Winterhalter M. Understanding carbapenem translocation through OccD3 (OpdP) of Pseudomonas aeruginosa. ACS Chem Biol 2017; 12(6): 1656–1664, https://doi.org/10.1021/acschembio.6b01150.
- Camacho C., Coulouris G., Avagyan V., Ma N., Papadopoulos J., Bealer K., Madden T.L. BLAST+: architecture and applications. BMC Bioinformatics 2009; 10: 421, https://doi.org/10.1186/1471-2105-10-421.
- Pang Z., Raudonis R., Glick B.R., Lin T.J., Cheng Z. Antibiotic resistance in Pseudomonas aeruginosa: mechanisms and alternative therapeutic strategies. Biotechnol Adv 2019; 37(1): 177–192, https://doi.org/10.1016/j.biotechadv.2018.11.013.
- Чеботарь И.В., Бочарова Ю.А., Маянский Н.А. Механизмы резистентности Pseudomonas aeruginosa к антибиотикам и их регуляция. Клиническая микробиология и антимикробная химиотерапия 2017; 19(4): 308–319.
- Saito R., Koyano S., Dorin M., Higurashi Y., Misawa Y., Nagano N., Kaneko T., Moriya K. Evaluation of a simple phenotypic method for the detection of carbapenemase-producing Enterobacteriaceae. J Microbiol Methods 2015; 108: 45–48, https://doi.org/10.1016/j.mimet.2014.11.008.
- Dallenne C., Da Costa A., Decré D., Favier C., Arlet G. Development of a set of multiplex PCR assays for the detection of genes encoding important beta-lactamases in Enterobacteriaceae. J Antimicrob Chemother 2010; 65(3): 490–495, https://doi.org/10.1093/jac/dkp498.
- Suresh M., Skariyachan S., Narayanan N., Pullampara Rajamma J., Panickassery Ramakrishnan M.K. Mutational variation analysis of oprD porin gene in multidrug-resistant clinical isolates of Pseudomonas aeruginosa. Microb Drug Resist 2020; 26(8): 869–879, https://doi.org/10.1089/mdr.2019.0147.










