Диагностика колоректального рака и аденоматозного полипа толстой кишки по уровню экспрессии микроРНК в слизистой оболочке (пилотное клиническое исследование)
«Золотой стандарт» диагностики колоректального рака (КРР) — колоноскопия с биопсией — является инвазивным методом и имеет ограничения, а известные неинвазивные методы не обладают достаточной чувствительностью и специфичностью. Использование микроРНК в качестве диагностического и прогностического биомаркера КРР потенциально может компенсировать ограничения колоноскопии. Однако в литературе отсутствуют данные о существовании реальных тест-систем на основе определения экспрессии микроРНК. Наше пилотное исследование является первым шагом к созданию тест-системы для диагностики КРР на основе анализа экспрессии микроРНК в ткани интактной толстой кишки.
Цель работы — оценить перспективность использования уровня экспрессии микроРНК в качестве дополнительного метода диагностики колоректального рака и аденоматозных полипов.
Материалы и методы. Исследование проведено с участием пациентов с КРР (n=5) — 1-я группа, полипами толстой кишки (n=4) — 2-я группа и пациентов без онкологической патологии толстой кишки, которые находились на лечении по поводу геморроидальной болезни вне обострения (n=5) — 3-я группа.
Всем пациентам проводили забор образца ткани интактной кишки. Пациентам 1-й и 2-й групп биопсию выполняли в ходе лапароскопической резекции правых отделов толстой кишки с опухолью. У пациентов 3-й группы забор образца слизистой оболочки дистального отдела прямой кишки также осуществляли интраоперационно; они были прооперированы по методу Миллигана–Моргана. В 1-й и 2-й группах кроме участка интактной кишки для анализа забирали образец КРР и полипа соответственно.
Исследовательская панель включала в себя следующие микроРНК: hsa-miR-10b-5p, hsa-miR-20a-5p, hsa-miR-141-3p, hsa-miR-181b-5p. Уровни микроРНК эталонных генов анализировали с помощью полимеразной цепной реакции в режиме реального времени с использованием интеркалирующего красителя SYBR Green.
Результаты. Экспрессия hsa-miR-141-3p в слизистой оболочке толстой кишки у пациентов 1-й и 2-й групп (с КРР и полипами соответственно) была статистически значимо выше, чем у пациентов без опухолевых заболеваний кишечника. При этом уровень экспрессии hsa-miR-10b-5p был статистически значимо ниже в опухолевой ткани (рака и полипа) по сравнению с пациентами 3-й группы.
Обнаружены более низкие показатели экспрессии всех исследованных микроРНК в ткани КРР по сравнению с интактной слизистой тех же пациентов. Аналогичная тенденция наблюдалась и у больных с аденоматозными полипами.
Заключение. Результаты исследования показали, что из четырех микроРНК, включенных в исследовательскую панель, диагностическую ценность для выявления опухолевого поражения толстой кишки имеют hsa-miR-141-3p и hsa-miR-10b-5p. Таким образом, полученные нами данные подтверждают перспективность дополнения эндоскопического исследования толстой кишки эпигенетическим анализом слизистой оболочки при скрининге опухолевого поражения.
Введение
По прогнозам ВОЗ, к 2040 г. количество впервые выявленного колоректального рака (КРР) возрастет до 3,2 млн новых случаев в год, что на 63% больше, чем в 2023 г. [1]. Ранняя диагностика является приоритетным направлением в снижении летальности от КРР, а разработка и внедрение диагностических систем с высокой чувствительностью и специфичностью для своевременной верификации данного диагноза — по-настоящему актуальная задача.
Перечень основных неинвазивных методов ранней диагностики КРР внесен в клинические рекомендации многих стран и включает анализ кала на скрытую кровь (FOBT — fecal occult blood test; чувствительность метода — 50–80%, специфичность — 85–97%) [2], иммунохимический анализ кала (FIT — fecal immunochemical test; чувствительность метода — 61–91% и специфичность — 91–98%) [3] и ДНК-тест кала (mt-sDNA, Cologuard®; чувствительность метода — 92%, специфичность — 94%) [4]. Эти тесты основаны на выявлении крови или фрагментов клеток опухолей в кишечном содержимом. К их ключевым недостаткам прежде всего относятся сравнительно низкие показатели выявляемости КРР, особенно при правосторонней локализации опухоли, что вызывает необходимость частого скрининга [5].
Основные инвазивные методы прямой визуализации, применяемые в качестве методов скрининга КРР, — сигмоидоскопия, колоноскопия, КТ-колонография и капсульная эндоскопия [6]. Колоноскопия и сигмоидоскопия являются высокоспецифичными и чувствительными методами и по этой причине признаются «золотым стандартом» в диагностике КРР [7]. Однако колоноскопия требует тщательной и длительной специальной подготовки толстой кишки к обследованию, седации пациента, а чувствительность и специфичность диагностики варьируют в зависимости от характеристик оборудования и компетенций врача-эндоскописта. Существенная часть пациентов отказывается от проведения колоноскопии из-за боязни; в некоторых случаях, которые также составляют значимую долю, ее проведение противопоказано из-за наличия спаек брюшной полости [8, 9]. Эти факторы сдерживают массовое и повсеместное распространение колоноскопии как безальтернативного, исчерпывающего метода скрининга опухолей толстой кишки. В результате частота существующего, но не диагностированного (интервального) КРР достигает 4–9% [10, 11]. По этим причинам необходимость разработки и внедрения дополнительных методов скрининга для ранней диагностики опухолей толстой кишки не вызывает сомнений.
С развитием омиксных технологий появилась и новая перспектива в повышении эффективности скрининга КРР. Исследования изменений активности экспрессии микроРНК в тканях кишки при КРР в настоящее время проводятся достаточно интенсивно [12–14], однако литературных данных о разработанных и внедренных в клинику тест-системах для верификации КРР на основе экспрессии микроРНК нами не обнаружено. Мы предполагаем, что сочетание колоноскопии с определением уровня экспрессии микроРНК в слизистой оболочке толстой кишки позволит разработать более информативную методику ранней диагностики КРР. Это в свою очередь поможет значительно снизить количество интервального КРР. Для проверки этой гипотезы необходимо прежде всего ответить на вопрос: существуют ли различия в экспрессии микроРНК в слизистой оболочке толстой кишки у пациентов с КРР, полипами толстой кишки и у пациентов без онкологических заболеваний толстой кишки. Решению этого вопроса посвящено наше пилотное клиническое исследование.
Цель работы — оценить перспективность использования уровня экспрессии микроРНК в качестве дополнительного метода диагностики колоректального рака и аденоматозных полипов.
Материалы и методы
Исследованы образцы тканей толстой кишки и биологических жидкостей 14 пациентов, среди которых 8 мужчин (57%) и 6 женщин (43%); медиана возраста — 67 [61; 74] лет. 5 пациентов проходили лечение по поводу КРР — 1-я группа; у 4 пациентов были найдены доброкачественные полипы толстой кишки — 2-я группа; у 5 пациентов не было онкологической патологии толстой кишки, они находились на лечении по поводу геморроидальной болезни вне обострения — 3-я группа (контроль).
Исследован 21 образец ткани толстой кишки: в 1-й группе — 5 образцов интактной ткани и 4 образца опухолевой ткани; во 2-й группе — 4 образца интактной ткани и 3 образца ткани полипа; в 3-й группе — 5 образцов интактной ткани слизистой оболочки толстой кишки. Сравнивали уровни экспрессии исследуемых микроРНК в слизистой оболочке толстой кишки между группами. Кроме того, отдельно сравнивали экспрессию микроРНК в тканях КРР (1-я группа) и аденоматозного полипа (2-я группа) с тканью интактной слизистой оболочки пациентов 3-й группы.
В исследование не включали пациентов с осложненным течением заболеваний — с кишечной непроходимостью, кровотечением, перфорацией опухоли и другими возможными осложнениями.
Все пациенты дали информированное согласие на участие в исследовании, в том числе на биопсию слизистой оболочки толстой кишки. Исследование проведено в соответствии с Хельсинкской декларацией и одобрено Этическим комитетом Приволжского исследовательского медицинского университета (код протокола №9, дата утверждения — 10.06.2022).
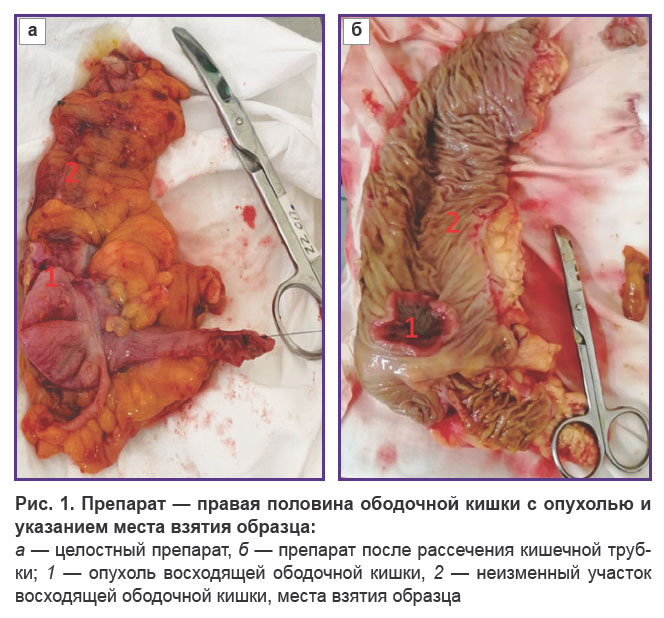
Всем пациентам проводили забор образца ткани интактной кишки. Пациентам 1-й и 2-й групп биопсию выполняли в ходе лапароскопической резекции правых отделов толстой кишки с опухолью. В ходе лапароскопической правосторонней гемиколэктомии и извлечения препарата из брюшной полости (рис. 1) через среднесрединную минилапаротомию вскрывали просвет толстой кишки и под визуальным контролем осуществляли забор образца слизистой оболочки размером 0,5×0,5 см, локализованного на расстоянии не менее 10 см от опухоли. Размер участка ткани соответствовал стандартным размерам биоптата, получаемым с помощью биопсийных щипцов при колоноскопии.
Лица, включенные в 3-ю группу, находились на лечении по поводу хронического геморроя II–III стадии и были прооперированы по методу Миллигана–Моргана. У них забор образца слизистой оболочки дистального отдела прямой кишки размерами 0,5×0,5 см осуществляли также интраоперационно. В 1-й и 2-й группах кроме участка интактной кишки для анализа забирали образец КРР и полипа соответственно.
Помимо рутинных лабораторных и инструментальных методов исследования (общий анализ крови, общий анализ мочи, биохимический анализ крови, коагулограмма, электрокардиография и т.д.) пациентам выполняли колоноскопию с гистологической верификацией диагноза, гастроскопию, КТ органов брюшной полости и органов грудной клетки с внутривенным контрастированием и МРТ органов малого таза; также выявляли наличие биомаркеров КРР (раковый эмбриональный антиген и СА 19-9).
Перечень исследованных микроРНК определили, основываясь на данных литературы [15]: hsa-miR-10b-5p, hsa-miR-20a-5p, hsa-miR-141-3p, hsa-miR-181b-5p. МикроРНК hsa-miR-10b-5p регулирует транскрипты CDKN1A, CDKN2A, BCL2L11, PTCH1, TP53; микроРНК hsa-miR-20a-5p — TP53INP1, CDKN1A, E2F1, PPP2R2A, TGFBR2, BCL2; микроРНК hsa-miR-141-3p — MAP4K4, PTEN, WDR37, CDC25A, KLF5; микроРНК hsa-miR-181b-5p — MCL1, BCL2, TCL1a, XIAP.
Выделение микроРНК из клинических образцов осуществляли с помощью набора HiPure Universal miRNA Kit (Magen Biotechnology Co., Ltd, Китай). Анализ уровня экспрессии микроРНК проводили с использованием наборов ALMIR («Алгимед Техно», Беларусь) в соответствии с рекомендациями производителя методом ПЦР в режиме реального времени на приборе Real-time CFX96 Touch (Bio-Rad, США). Для образцов тканей экспрессию микроРНК нормализовали относительно набора референсных генов (EIF2B1, IPO8, ABL1), определенного алгоритмом geNorm [16, 17]. Уровни мРНК эталонных генов анализировали с помощью ПЦР в режиме реального времени с использованием интеркалирующего красителя SYBR Green.
Статистический анализ выполняли при помощи пакета прикладных программ IBM SPSS Statistics 20. Проверку нормальности распределения количественных признаков проводили с использованием критерия Колмогорова–Смирнова. Все исследованные показатели не имели нормального распределения, поэтому оценку статистической значимости различий при сравнении групп по количественным признакам выполняли непараметрическими методами. Для сравнения показателей в группах применяли критерий Краскела–Уоллиса. Выборочные параметры, приводимые далее, имеют следующие обозначения: Ме — медиана, Q1 — верхний квартиль, Q3 — нижний квартиль, минимум (min) и максимум (max) — минимальное и максимальное значения переменной, n — объем анализируемой подгруппы, р — величина статистической значимости различий. Критическое значение уровня значимости принимали равным 5% (р≤0,05). При использовании множественных сравнений уровень значимости различий указан в виде скорректированного значения (padjusted=p·m, где p — полученное по результатам сравнения значение, m — число сравнений).
Результаты
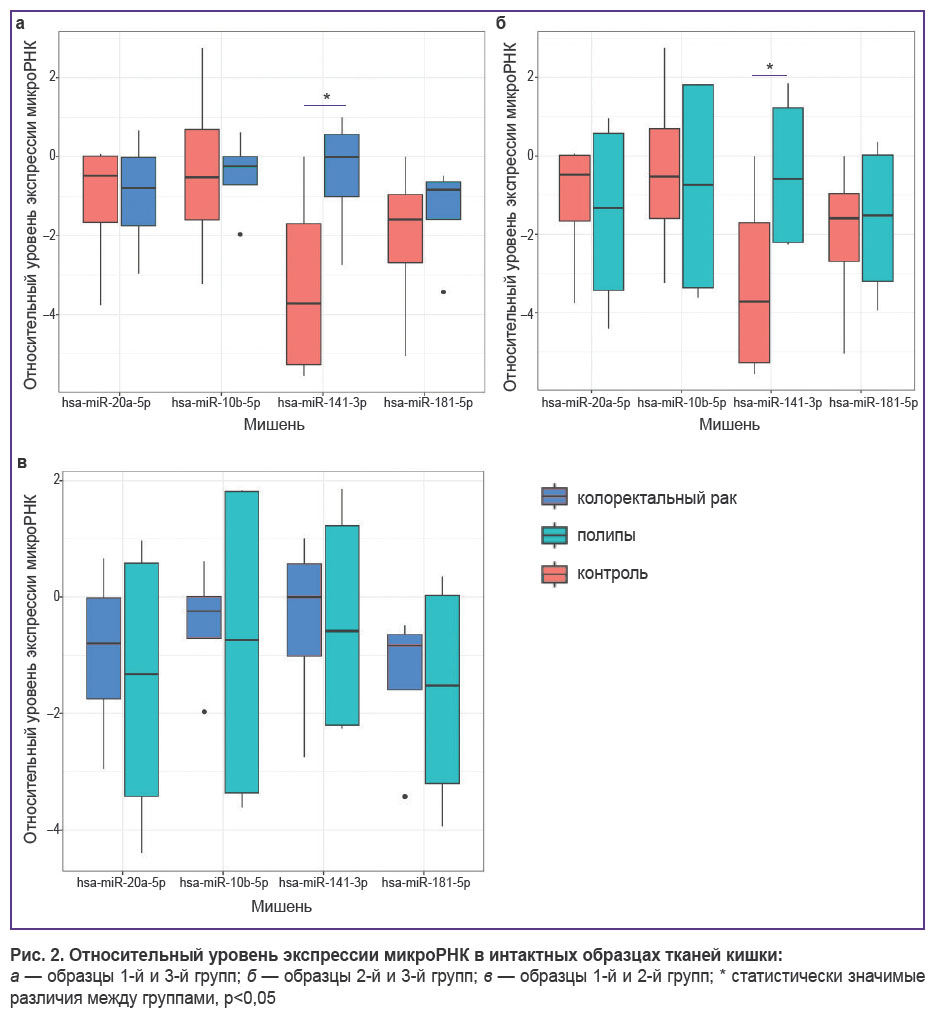
Экспрессия микроРНК в интактной слизистой оболочке кишки у пациентов всех трех групп. При множественном сравнении 1, 2 и 3-й групп (пациенты с КРР, полипами и геморроидальной болезнью) установлены статистически значимые различия уровня экспрессии hsa-miR-141-3p (критерий Краскела–Уоллиса, р=0,032) в ткани интактной кишки. При попарном сравнении обнаружено, что экспрессия hsa-miR-141-3p в 1-й и 2-й группах статистически значимо выше, чем в 3-й группе (рис. 2, а, б) (p=0,032). При этом статистически значимых различий данного показателя при сравнении 1-й и 2-й групп (биоптатов пациентов с КРР и полипами) не выявлено (рис. 2, в).
Статистически значимых различий между группами в уровне экспрессии hsa-miR-10b-5p, hsa-miR-20a-5p, hsa-miR-181b-5p в образцах интактной ткани кишки также не выявлено (см. рис. 2).
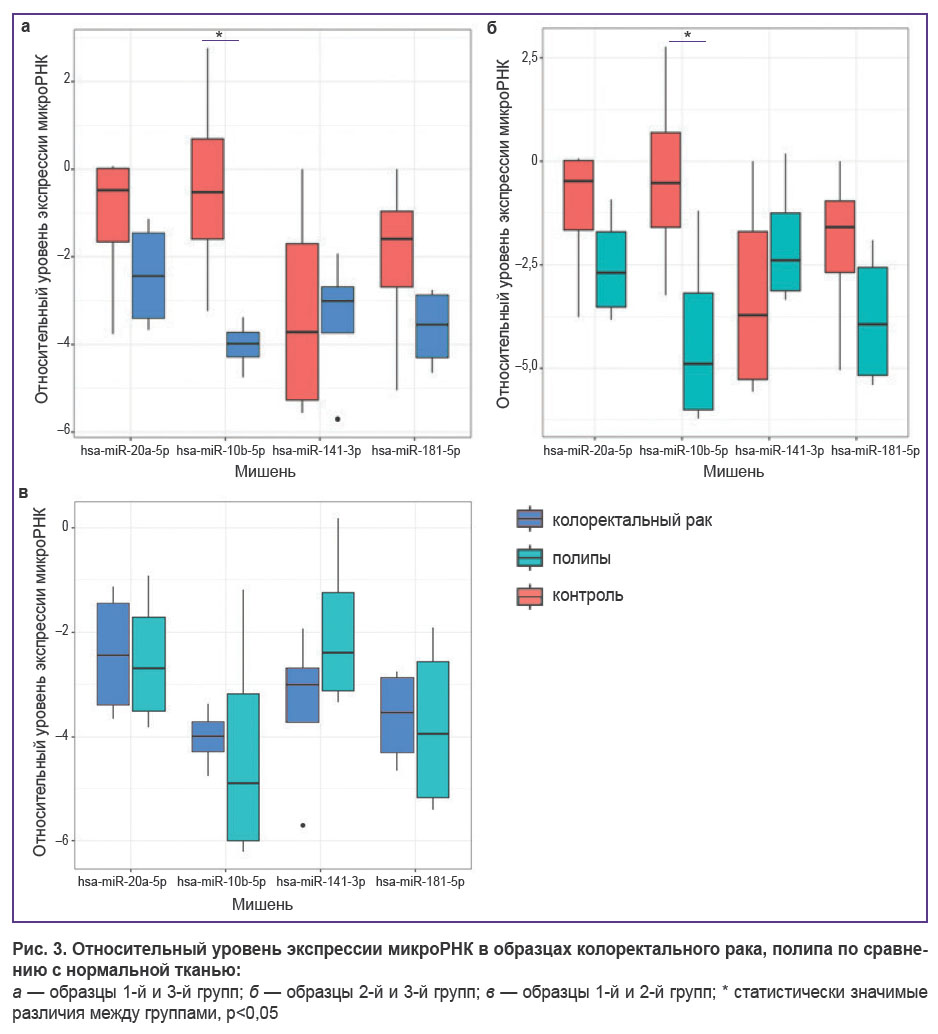
Экспрессия микроРНК в опухолевой ткани 1-й и 2-й групп (КРР и полипы) и ткани нормальной слизистой оболочки кишки 3-й группы. При сравнении показателей экспрессии микроРНК наблюдалась однонаправленная тенденция: уровень экспрессии hsa-miR-10b-5p, hsa-miR-20a-5p, hsa-miR-141-3p, hsa-miR-181b-5p в тканях пациентов 3-й группы был выше, чем в тканях КРР или полипа (рис. 3, а, б). Статистически значимые различия выявлены только в уровне экспрессии hsa-miR-10b-5p как при сравнении 1-й и 3-й групп (рadjusted=0,018; см. рис. 3, а), так и при сравнении 2-й и 3-й групп (рadjusted=0,011; см. рис. 3, б). При этом в ходе сравнения уровня экспрессии в злокачественной опухоли и в полипе статистически значимых различий не обнаружено (рис. 3, в).
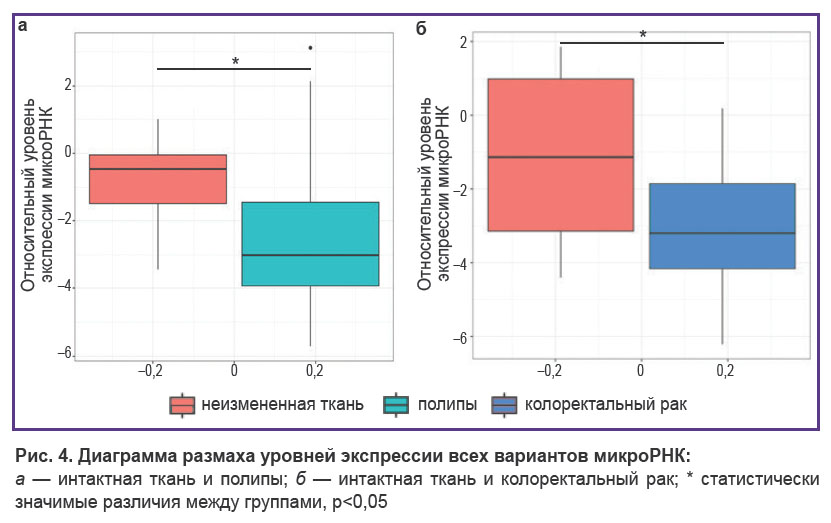
Экспрессия микроРНК в опухолевой и интактной ткани 1-й и 2-й групп. Обнаружены более низкие показатели экспрессии четырех исследованных микроРНК в ткани КРР (рис. 4) по сравнению с интактной слизистой тех же пациентов (рadjusted=0,033). Аналогичная тенденция наблюдалась и у больных с аденоматозными полипами (рadjusted=0,045).
|
Рис. 4. Диаграмма размаха уровней экспрессии всех вариантов микроРНК: а — интактная ткань и полипы; б — интактная ткань и колоректальный рак; * статистически значимые различия между группами, р<0,05 |
Таким образом, наблюдался общий эффект снижения экспрессии микроРНК в опухолевой ткани (КРР и полипы) по сравнению с интактной слизистой оболочкой. Соответственно, вышеописанное снижение has-miR-10b-5p в опухолевой ткани может быть следствием этого эффекта.
Обсуждение
К 2024 г. в мире зарегистрированы только два эпигенетических диагностических коммерческих набора, которые применяются на практике для анализа экспрессии генов с помощью метилированной ДНК: SEPT9 и VIM [18, 19]. Нужно отметить, что один из них — тест для обнаружения метилированного гена SEPT9 (Epi procolon®) — имеет сравнительно низкую чувствительность (48%) при специфичности 92%. В диагностике подозрительных аденом чувствительность данного метода еще меньше [20]. Таким образом, исследования в области разработки новых диагностических тестов на основе анализа экспрессии микроРНК в диагностике КРР в настоящее время имеют высокую актуальность [15, 21–24].
Повышенный уровень экспрессии hsa-miR-141-3p может означать благоприятный прогноз течения КРР, поскольку снижение уровня экспрессии hsa-miR-141-3p способствует развитию более агрессивной опухоли, ускоренному росту и метастазированию. Уровень экспрессии hsa-miR-10b-5p связан с активностью и пролиферацией опухолевых клеток. По данным литературы, экспрессия коррелирует со стадией развития опухоли [25, 26], что может служить подтверждением высокого риска их злокачественного перерождения. Примечательно, что интактная ткань, полученная от пациентов с полипами, имеет высокую гетерогенность и четкую кластеризацию на образцы с повышенной и пониженной экспрессией относительно нормальной ткани, что может свидетельствовать о риске малигнизации аденоматозного полипа.
Заключение
Из четырех микроРНК (hsa-miR-10b-5p, hsa-miR-20a-5p, hsa-miR-141-3p, hsa-miR-181b-5p), включенных в исследовательскую панель, диагностическую ценность для выявления опухолевого поражения толстой кишки имеют hsa-miR-141-3p и hsa-miR-10b-5p. Экспрессия hsa-miR-141-3p в слизистой оболочке толстой кишки у пациентов с колоректальным раком и у пациентов с полипами статистически значимо выше, чем у пациентов без онкологических заболеваний кишечника. При этом сравнение уровня экспрессии hsa-miR-10b-5p в опухолевой ткани (рака и полипа) с тканью слизистой оболочки кишки пациентов без опухолей кишечника показало обратный результат: в ткани колоректального рака и аденоматозного полипа этот показатель достоверно ниже. Все исследованные микроРНК в интактной ткани характеризуются значительно более высоким уровнем экспрессии по сравнению с опухолевой тканью. Таким образом, полученные в пилотном исследовании данные подтверждают перспективность дополнения эндоскопического исследования толстой кишки эпигенетическим анализом слизистой оболочки при скрининге опухолевого поражения.
Финансирование исследования. Работа финансировалась из средств гранта «Приоритет 2030».
Конфликт интересов отсутствует.
Литература
- Morgan E., Arnold M., Gini A., Lorenzoni V., Cabasag C.J., Laversanne M., Vignat J., Ferlay J., Murphy N., Bray F. Global burden of colorectal cancer in 2020 and 2040: incidence and mortality estimates from GLOBOCAN. Gut 2023; 72(2): 338–344, https://doi.org/10.1136/gutjnl-2022-327736.
- Knudsen A.B., Zauber A.G., Rutter C.M., Naber S.K., Doria-Rose V.P., Pabiniak C., Johanson C., Fischer S.E., Lansdorp-Vogelaar I., Kuntz K.M. Estimation of benefits, burden, and harms of colorectal cancer screening strategies: modeling study for the us preventive services task force. JAMA 2016; 315(23): 2595–2609, https://doi.org/10.1001/jama.2016.6828.
- van Dam L., Kuipers E.J., van Leerdam M.E. Performance improvements of stool-based screening tests. Best Pract Res Clin Gastroenterol 2010; 24(4): 479–492, https://doi.org/10.1016/j.bpg.2010.03.009.
- Imperiale T.F., Ransohoff D.F., Itzkowitz S.H., Levin T.R., Lavin P., Lidgard G.P., Ahlquist D.A., Berger B.M. Multitarget stool DNA testing for colorectal-cancer screening. N Engl J Med 2014; 370(14): 1287–1297, https://doi.org/10.1056/NEJMoa1311194.
- Robertson D.J., Lee J.K., Boland C.R., Dominitz J.A., Giardiello F.M., Johnson D.A., Kaltenbach T., Lieberman D., Levin T.R., Rex D.K. Recommendations on fecal immunochemical testing to screen for colorectal neoplasia: a consensus statement by the US Multi-Society Task Force on Colorectal Cancer. Gastroenterology 2017; 152(5): 1217–1237.e3, https://doi.org/10.1053/j.gastro.2016.08.053.
- Wu Z., Li Y., Zhang Y., Hu H., Wu T., Liu S., Chen W., Xie S., Lu Z. Colorectal cancer screening methods and molecular markers for early detection. Technol Cancer Res Treat 2020; 19: 1533033820980426, https://doi.org/10.1177/1533033820980426.
- Issa I.A., Noureddine M. Colorectal cancer screening: an updated review of the available options. World J Gastroenterol 2017; 23(28): 5086–5096, https://doi.org/10.3748/wjg.v23.i28.5086.
- Jain S., Maque J., Galoosian A., Osuna-Garcia A., May F.P. Optimal strategies for colorectal cancer screening. Curr Treat Options Oncol 2022; 23(4): 474–493, https://doi.org/10.1007/s11864-022-00962-4.
- Полянская Е.А., Федянин М.Ю., Трякин А.А., Тюляндин С.А. Скрининг рака толстой кишки: достижения и перспективы. Онкологическая колопроктология 2018;8(4):11–29.
- Teixeira C., Martins C., Dantas E., Trabulo D., Mangualde J., Freire R., Alves A.L., Cremers I., Oliveira A.P. Interval colorectal cancer after colonoscopy. Rev Gastroenterol Mex (Engl Ed) 2019; 84(3): 284–289, https://doi.org/10.1016/j.rgmx.2018.04.006.
- Lee J.Y., Lee J.H. Post-colonoscopy colorectal cancer: causes and prevention of interval colorectal cancer. Korean J Gastroenterol 2020; 75(6): 314–321, https://doi.org/10.4166/kjg.2020.75.6.314.
- Giannopoulou N., Constantinou C. Recent developments in diagnostic and prognostic biomarkers for colorectal cancer: a narrative review. Oncology 2023; 101(10): 675–684, https://doi.org/10.1159/000531474.
- Gmerek L., Martyniak K., Horbacka K., Krokowicz P., Scierski W., Golusinski P., Golusinski W., Schneider A., Masternak M.M. MicroRNA regulation in colorectal cancer tissue and serum. PLoS One 2019; 14(8): e0222013, https://doi.org/10.1371/journal.pone.0222013.
- Moghadamnia F., Ghoraeian P., Minaeian S., Talebi A., Farsi F., Akbari A. MicroRNA expression and correlation with mRNA levels of colorectal cancer-related genes. J Gastrointest Cancer 2020; 51(1): 271–279, https://doi.org/10.1007/s12029-019-00249-2.
- Багрянцев М.В., Самойленко В.М., Рябков М.Г., Базаев А.В., Дезорцев И.Л., Бунова С.С., Батанов М.А., Киселева Е.Б. Эпигенетические маркеры колоректального рака: анализ данных о клиническом применении. Вестник экспериментальной и клинической хирургии 2021; 14(4): 316–324, https://doi.org/10.18499/2070-478X-2021-14-4-316-324.
- geNorm. URL: https://genorm.cmgg.be/.
- URL: https://www.gene-quantification.de/hkg.html.
- Gopal P., Ahmed Z., Venkata Ravi Kant V., Rao G.V., Rebala P. Circulating tumor DNA for monitoring colorectal cancer: a prospective observational study to assess the presence of methylated SEPT9 and VIM promoter genes and its role as a biomarker in colorectal cancer management. Turk J Surg 2023; 39(2): 107–114, https://doi.org/10.47717/turkjsurg.2023.6038.
- Deris Zayeri Z., Parsi A., Shahrabi S., Kargar M., Davari N., Saki N. Epigenetic and metabolic reprogramming in inflammatory bowel diseases: diagnostic and prognostic biomarkers in colorectal cancer. Cancer Cell Int 2023; 23(1): 264, https://doi.org/10.1186/s12935-023-03117-z.
- Church T.R., Wandell M., Lofton-Day C., Mongin S.J., Burger M., Payne S.R., Castaños-Vélez E., Blumenstein B.A., Rösch T., Osborn N., Snover D., Day R.W., Ransohoff D.F.; PRESEPT Clinical Study Steering Committee, Investigators and Study Team. Prospective evaluation of methylated SEPT9 in plasma for detection of asymptomatic colorectal cancer. Gut 2014; 63(2): 317–325, https://doi.org/10.1136/gutjnl-2012-304149.
- Huang Q., Song Q., Zhong W., Chen Y., Liang L. MicroRNA-10b and the clinical outcomes of various cancers: a systematic review and meta-analysis. Clin Chim Acta 2017; 474: 14–22, https://doi.org/10.1016/j.cca.2017.08.034.
- Wang Y.H., Ji J., Weng H., Wang B.C., Wang F.B. MiR-139 in digestive system tumor diagnosis and detection: Bioinformatics and meta-analysis. Clin Chim Acta 2018; 485: 33–41, https://doi.org/10.1016/j.cca.2018.06.006.
- Gu X., Jin R., Mao X., Wang J., Yuan J., Zhao G. Prognostic value of miRNA-181a/b in colorectal cancer: a meta-analysis. Biomark Med 2018; 12(3): 299–308, https://doi.org/10.2217/bmm-2016-0222.
- Zhang Q., Wang Q., Sun W., Gao F., Liu L., Cheng L., Li Z. Change of circulating and tissue-based miR-20a in human cancers and associated prognostic implication: a systematic review and meta-analysis. Biomed Res Int 2018; 2018: 6124927, https://doi.org/10.1155/2018/6124927.
- Niu X., Sun H., Qiu F., Liu J., Yang T., Han W. miR-10b-5p Suppresses the proliferation and invasion of primary hepatic carcinoma cells by downregulating epha2 [retracted in: Biomed Res Int 2024; 2024: 9834789, https://doi.org/10.1155/2024/9834789]. Biomed Res Int 2021; 2021: 1382061, https://doi.org/10.1155/2021/1382061.
- Yoshida K., Yokoi A., Kitagawa M., Sugiyama M., Yamamoto T., Nakayama J., Yoshida H., Kato T., Kajiyama H., Yamamoto Y. Downregulation of miR‑10b‑5p facilitates the proliferation of uterine leiomyosarcoma cells: a microRNA sequencing‑based approach. Oncol Rep 2023; 49(5): 86, https://doi.org/10.3892/or.2023.8523.