Генетические факторы риска нетуберкулезных микобактериальных заболеваний легких (систематический обзор)
Данная работа представляет собой систематический обзор литературных источников, описывающих генетические факторы риска развития пульмонологических заболеваний, вызванных нетуберкулезными микобактериями, — нетуберкулезных микобактериозов легких (НТМЛ).
Цель исследования — составление конкретного списка генетических маркеров, связанных с риском развития НТМЛ.
Данный литературный обзор подготовлен по критериям PRISMA, а также зарегистрирован в международном проспективном перечне систематических обзоров PROSPERO (регистрационный номер — CRD42019128569).
В ходе работы изучено множество статей из баз данных PubMed, Google Scholar и ScienceDirect. В результате тщательного отбора и анализа составлен список из 14 генетических вариантов, ассоциированных с повышенным риском развития НТМЛ. К ним относятся варианты в генах SLC11A1, NLRP3, TLR2, CFTR, IFNGR1, PDCD1, а также варианты в межгенных областях, влияющие на экспрессию генов STK17A, IFNL3, TNF, IL10. Продукты данных генов выполняют разнообразные функции в регуляции ответа на патогенные факторы, причем некоторые из них мало изучены. Для более точной оценки влияния этих генетических вариантов на риск развития НТМЛ требуется дальнейшее изучение данной проблемы в группах пациентов различных популяций с учетом комбинаций генетических вариантов и межгенных взаимодействий.
Введение
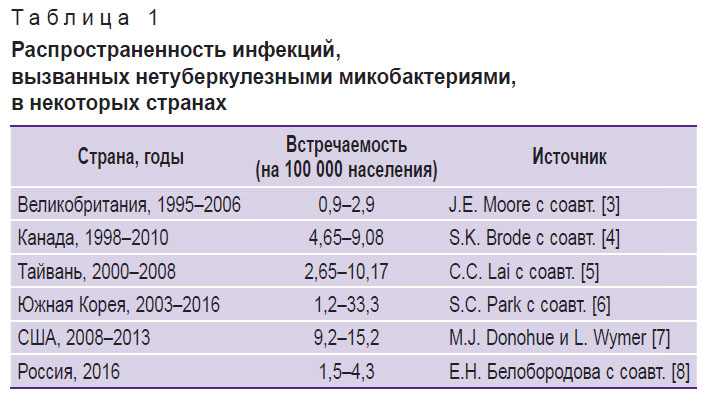
Род Mycobacterium — это большая гетерогенная группа микроорганизмов, включающая сапрофитные и условно-патогенные виды, некоторые из которых способны вызывать такие болезни, как туберкулез, лепра, а также микобактериозы [1, 2]. Повсеместная распространенность инфекций, ассоциированных с нетуберкулезными микобактериями — НТМБ (табл. 1) [3–8], обусловливает повышенный интерес медицинского сообщества к заболеваниям, вызванным этими патогенами [1, 9, 10].
|
Таблица 1. Распространенность инфекций, вызванных нетуберкулезными микобактериями, в некоторых странах |
Увеличение случаев выявления инфекций, вызванных НТМБ, может быть связано как с совершенствованием методов диагностики, так и с возросшей осведомленностью медицинского сообщества о данных микобактериях [3, 5]. Однако заболевания, относящиеся к НТМБ-инфекциям, включая наиболее распространенную форму — нетуберкулезные микобактериозы легких (НТМЛ), не входят в список для обязательной регистрации [9]. Эта особенность затрудняет актуальный подсчет заболеваемости.
Наибольшему риску развития НТМЛ подвержены иммунокомпрометированные лица, страдающие от ВИЧ-инфекций; пациенты, получающие иммуносупрессивную терапию в связи с трансплантацией органов; больные с хроническими заболеваниями легких, такими как муковисцидоз, хроническая обструктивная болезнь легких, бронхоэктатическая болезнь и другие. Пожилые люди также склонны к повышенному риску развития НТМЛ [11, 12].
Таким образом, выявление генетически-обусловленных факторов предрасположенности к НТМЛ может помочь в разработке персонализированных подходов для предотвращения развития осложнений у лиц в группе риска.
Цель систематического обзора — составление списка генетических маркеров, обусловливающих повышенный риск развития нетуберкулезных микобактериозов легких, на основе анализа литературных источников и сведений геномных баз данных.
Материалы и методы
Критерии включения и исключения представлены в табл. 2. Для анализа решили отбирать исследования случай–контроль, а также направленные на поиск полногеномных ассоциаций (GWAS). Исследуемые группы должны были состоять из лиц 18 лет и старше, исключая ВИЧ-инфицированных, онкологических больных или пациентов, страдающих от системных заболеваний и получающих иммуносупрессивную терапию. Описанный генетический вариант считали протективным при отношении шансов меньше единицы (OR<1). В случае OR=1 корреляции не выявлено. При OR>1 установлена взаимосвязь между генетическим вариантом и повышенным риском развития НТМЛ. Ассоциация считалась статистически значимой только при p≤0,05. Таким образом, мы исключали литературные источники, в которых корреляция не была выявлена или при OR≤1.

|
Таблица 2. Критерии включения и исключения литературных источников |
Стратегия поиска литературных источников. Для нашей работы мы обратились к трем глобальным базам данных: PubMed, Google Scholar и ScienceDirect. Поиск не ограничивали по времени публикации материала.
Поисковый запрос в PubMed включал фразы “nontuberculous mycobacterial lung disease” или “nontuberculous mycobacterial pulmonary disease”, а также слова “gene”, “polymorphism” и их производные. Для предварительной фильтрации результатов поиска выводная информация не должна была содержать слово “children” и его производные. В соответствии с вышеизложенными условиями запрос для поискового алгоритма выглядел следующим образом:
(((“nontuberculous”[All Fields] AND (“mycobacterial”[All Fields] OR “mycobacterials”[All Fields]) AND (“lung diseases”[MeSH Terms] OR (“lung”[All Fields] AND “diseases”[All Fields]) OR “lung diseases”[All Fields] OR (“lung”[All Fields] AND “disease”[All Fields]) OR “lung disease”[All Fields])) OR (“nontuberculous”[All Fields] AND (“mycobacterial”[All Fields] OR “mycobacterials”[All Fields]) AND (“lung diseases”[MeSH Terms] OR (“lung”[All Fields] AND “diseases”[All Fields]) OR “lung diseases”[All Fields] OR (“pulmonary”[All Fields] AND “disease”[All Fields]) OR “pulmonary disease”[All Fields]))) AND (“genes”[MeSH Terms] OR “genes”[All Fields] OR “gene”[All Fields]) AND (“polymorphic”[All Fields] OR “polymorphics”[All Fields] OR “polymorphism s”[All Fields] OR “polymorphism, genetic”[MeSH Terms] OR (“polymorphism”[All Fields] AND “genetic”[All Fields]) OR “genetic polymorphism”[All Fields] OR “polymorphism”[All Fields] OR “polymorphisms”[All Fields])) NOT (“child”[MeSH Terms] OR “child”[All Fields] OR “children”[All Fields] OR “child s”[All Fields] OR “children s”[All Fields] OR “childrens”[All Fields] OR “childs”[All Fields]).
В результате поиска отобрано 28 публикаций. В связи с малым количеством источников провели дополнительные поиски в агрегаторе Google Scholar и базе данных ScienceDirect. Для запроса в Google Scholar использовали фразу “genetic factors of nontuberculous mycobacterial lung disease” и получили 25 700 записей. В поле поиска ScienceDirect вводили “nontuberculous mycobacterial pulmonary disease genetics”, что привело к выводу 348 записей. Далее проводили скрининг работ по заголовкам и аннотациям, в ходе которого исключали статьи, не подходящие к теме нашей работы. К таким статьям мы относили исследования микобактериальных культур, клинические наблюдения и дублирующиеся записи. В итоге перечень литературных источников составил 42 статьи.
На следующем этапе тщательно анализировали полные тексты отобранных статей и их сопроводительные материалы (при наличии), опираясь на критерии включения и исключения. Мы исключили статьи, в которых исследуемая группа состояла из несовершеннолетних (до 18 лет), пациентов с онкологическими заболеваниями или лиц с системными заболеваниями, получающих иммуносупрессивную терапию. Во внимание также не брали работы, в которых взаимосвязь между генетическими вариантами и риском развития НТМЛ была статистически незначимой (p>0,05).
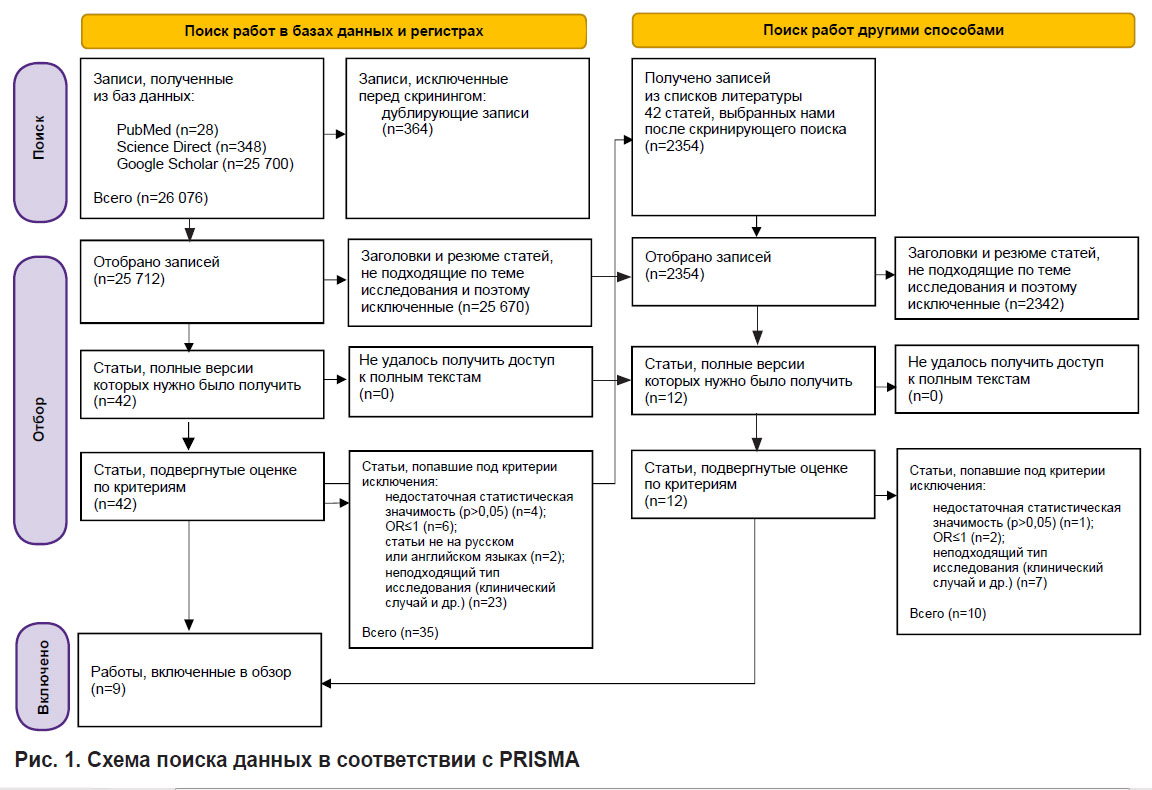
Далее мы изучили списки литературных источников избранных статей и проанализировали еще 12 публикаций. После исключения статей, неподходящих по нашим критериям, осталось 9 работ. Схема отбора источников представлена на рис. 1.
|
Рис. 1. Схема поиска данных в соответствии с PRISMA |
Данный литературный обзор подготовлен с учетом критериев PRISMA, а также зарегистрирован в международном проспективном перечне систематических обзоров PROSPERO (регистрационный номер — CRD42019128569).
Результаты
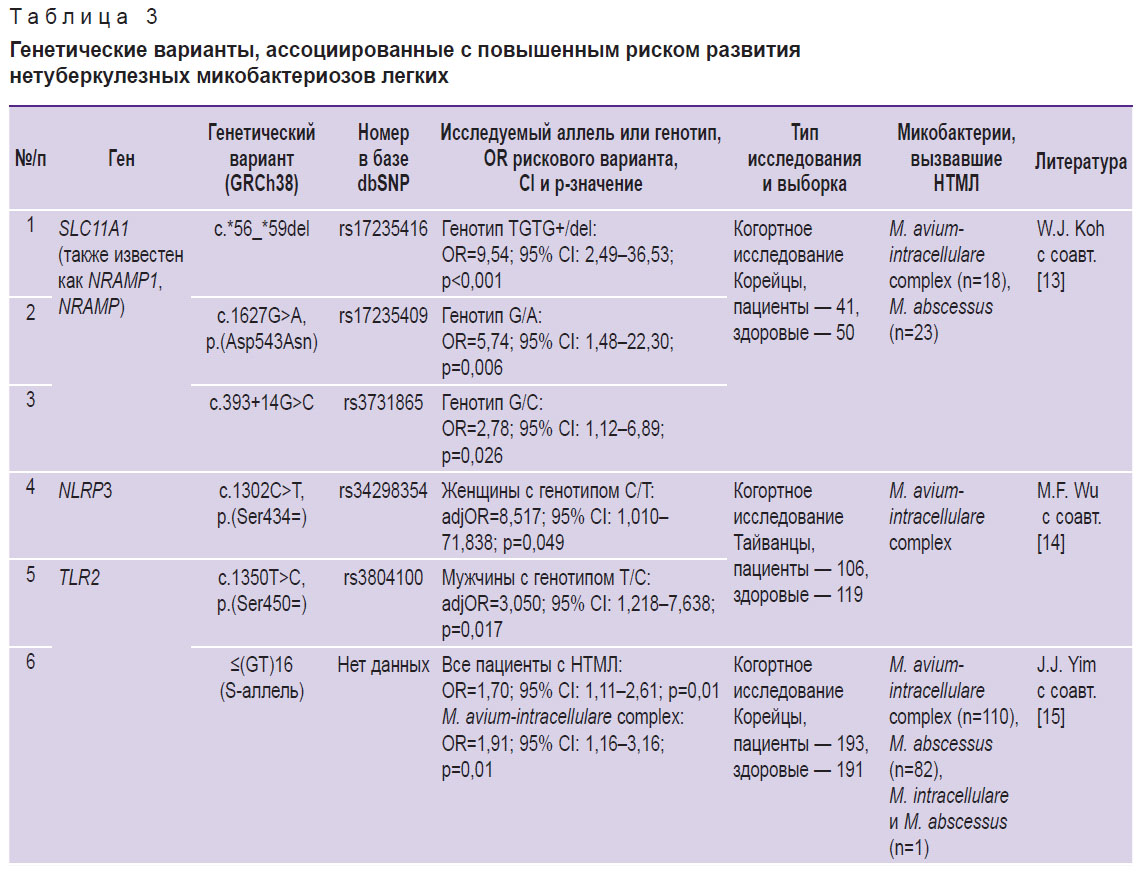
В результате тщательного отбора и систематического анализа множества публикаций составлен список генетических маркеров, наличие которых может свидетельствовать о повышенном риске развития НТМЛ. Этот список включает 14 генетических вариантов в 10 генах (табл. 3) [13–20]. Приведенные нами генетические варианты связаны с различными иммунологическими процессами, в том числе с ответом на микобактерии. Ниже представлена информация о роли каждого гена и его продукта.
|
Таблица 3. Генетические варианты, ассоциированные с повышенным риском развития нетуберкулезных микобактериозов легких |
Ген SLC11A1 (также NRAMP, NRAMP1), кодирующий одноименный белок, расположен на длинном плече второй хромосомы (2q35). Белок SLC11A1 является аналогом мышиного белка Slc11a1, который участвует в защите от микроорганизмов, в том числе от Mycobacterium tuberculosis, лейшманий и сальмонелл [21–23]. Основная функция этого белка заключается в переносе ионов металлов в любом направлении против протонного градиента. SLC11A1 локализуется на лизосомных мембранах и транспортирует дивалентные катионы из цитозоля внутрь лизосом для их прямого участия в антимикробной активности. Разделение ионов металлов, в частности Fe2+ и Mn2+, являющихся кофакторами как у прокариот, так и у эукариот, способствует защите макрофагов от активных форм кислорода и одновременно не позволяет патогенным микроорганизмам использовать железо и магний для синтеза защитных ферментов [24, 25].
Ген NLRP3, кодирующий белок NLRP3, расположен на длинном плече первой хромосомы (1q44). Данный белок, также известный как криопирин, в основном экспрессируется в хондроцитах и белых клетках крови, в частности макрофагах. Криопирин является частью NLRP3-инфламмасомы и отвечает за ее активацию в ответ на повреждение мембраны клетки и на присутствие инородных объектов [26]. Активацию NLRP3 стимулируют внеклеточный аденозинтрифосфат, нигерицин, активные формы кислорода, кристаллы уратов или холестерина, волокна бета-амилоида, а также частицы и наночастицы, такие как асбест, оксид кремния, угольная пыль и другие [27–29].
Следует отметить, что в работе M.F. Wu и соавт. [14] была выявлена взаимосвязь варианта rs34298354 с развитием вызванного Mycobacterium avium complex НТМЛ у женщин при генотипе С/Т (adjOR=8,517; p=0,049) и варианта rs3806268 — при генотипе G/A (adjOR=1,945; p=0,085). Мы не включили вариант rs3806268 в наш список в связи с тем, что p-значение было выше 0,05.
Ген TLR2 кодирует одноименный белок, который является членом семейства толл-подобных рецепторов (TLR), играющих фундаментальную роль в распознавании патогенов и активации врожденного иммунитета. TLR2 находится на поверхности клеток и может образовывать гетеродимеры с другими белками семейства TLR для распознавания патоген-ассоциированных молекулярных паттернов (PAMP). Активация с помощью PAMP приводит к запуску сигнальных путей и последующему воспалительному ответу. Cчитается также, что этот белок способствует апоптозу в ответ на бактериальные липопротеины, в том числе и содержащиеся в M. tuberculosis [30–32]. TLR2 связан с патогенезом нескольких аутоиммунных заболеваний.
Ген CFTR, кодирующий белок CFTR, находится на длинном плече седьмой хромосомы (7q31.2). CFTR является трансмембранным регулятором муковисцидоза и экспрессируется в различных органах, включая поджелудочную железу и легкие, а также в протоках потовых желез и других тканях [33–35]. Данный белок относится к семейству ABC-транспортеров и состоит из двух трансмембранных доменов, соединенных с цитоплазматическими доменами. Встраиваясь в апикальную мембрану эпителиальных клеток, CFTR регулирует транспорт ионов хлора [36]. Повреждение хлорного канала приводит к изменению электролитного состава клетки и, как следствие, нарушению секретируемого продукта [37].
Ген IFNGR1 расположен на длинном плече шестой хромосомы (6q23.3) и кодирует белок IFNGR1. Данный белок наряду с IFNGR2 является частью рецептора интерферона-гамма, который играет важную роль в антимикробном и противоопухолевом ответе, активируя эффекторные иммунные клетки и усиливая антиген-презентацию [38, 39].
Ген STK17A (также DRAK1) расположен на коротком плече седьмой хромосомы (7p13) и кодирует белок STK17A, который относится к серин/треонин-киназам. Этот белок участвует в регуляции апоптоза и метаболизме активных форм кислорода [40, 41].
Ген PDCD1 расположен на длинном плече второй хромосомы (2q37.3) и кодирует мембранный белок PD1, относящийся к надсемейству иммуноглобулинов. PD1 имеет два лиганда — PD-L1 и PD-L2 — и участвует в отрицательной регуляции иммунной системы, предотвращая активацию Т-лимфоцитов, тем самым снижая аутоиммунность и одновременно повышая аутотолерантность [42]. PDCD1-опосредованный путь может использоваться раковыми клетками для изменения противоопухолевого ответа и избегания уничтожения иммунной системой [43].
IFNL3 (также IL28B) расположен на длинном плече девятнадцатой хромосомы (19q13.2) и кодирует белок интерферон лямбда 3, который представляет собой цитокин с противовирусной, противоопухолевой и иммуномодуляторной активностью. Этот белок играет важную роль в противовирусном ответе (в основном в эпителиальных тканях), являясь лигандом к гетеродимерному рецептору, состоящему из IL10RB и IFNLR1. Взаимодействие с рецептором приводит к активации пути JAK/STAT и экспрессии интерферон-стимулируемых генов [44, 45].
Ген TNF, кодирующий фактор некроза опухоли альфа (TNF-α), находится на коротком плече шестой хромосомы (6p21.33). TNF-α — цитокин, секретируемый макрофагами, который способствует клеточной смерти некоторых опухолевых клеток. Этот цитокин также является важным пирогеном, вызывающим лихорадку, воздействуя напрямую или стимулируя секрецию интерлейкина-1. При определенных условиях TNF-α может способствовать клеточной пролиферации и индуцировать дифференциацию клеток [46].
Ген IL10 расположен на длинном плече первой хромосомы (1q32.1) и кодирует интерлейкин-10 — цитокин, который участвует в ограничении иммунного ответа и воспаления, что помогает уменьшить повреждение клеток организма. Мутации в данном гене ассоциируют с восприимчивостью к ВИЧ-инфекции и ревматоидному артриту [47–49].
Обсуждение
По данным проанализированных нами статей, повышенный риск развития НТМЛ ассоциирован с женским полом, хронической патологией органов дыхания и снижением иммунологической реактивности [50]. Генетический фактор является тем нюансом, который при сочетании даже с одним из названных выше факторов может сыграть решающую роль в развитии активного патологического процесса и обусловить характер его течения. Учитывая связь НТМЛ с состояниями, снижающими защиту организма от проникновения чужеродных антигенов, можно предположить, что у людей с обозначенными факторами риска имеются генетически детерминированные особенности врожденного иммунитета, что и было продемонстрировано в нашем обзоре.
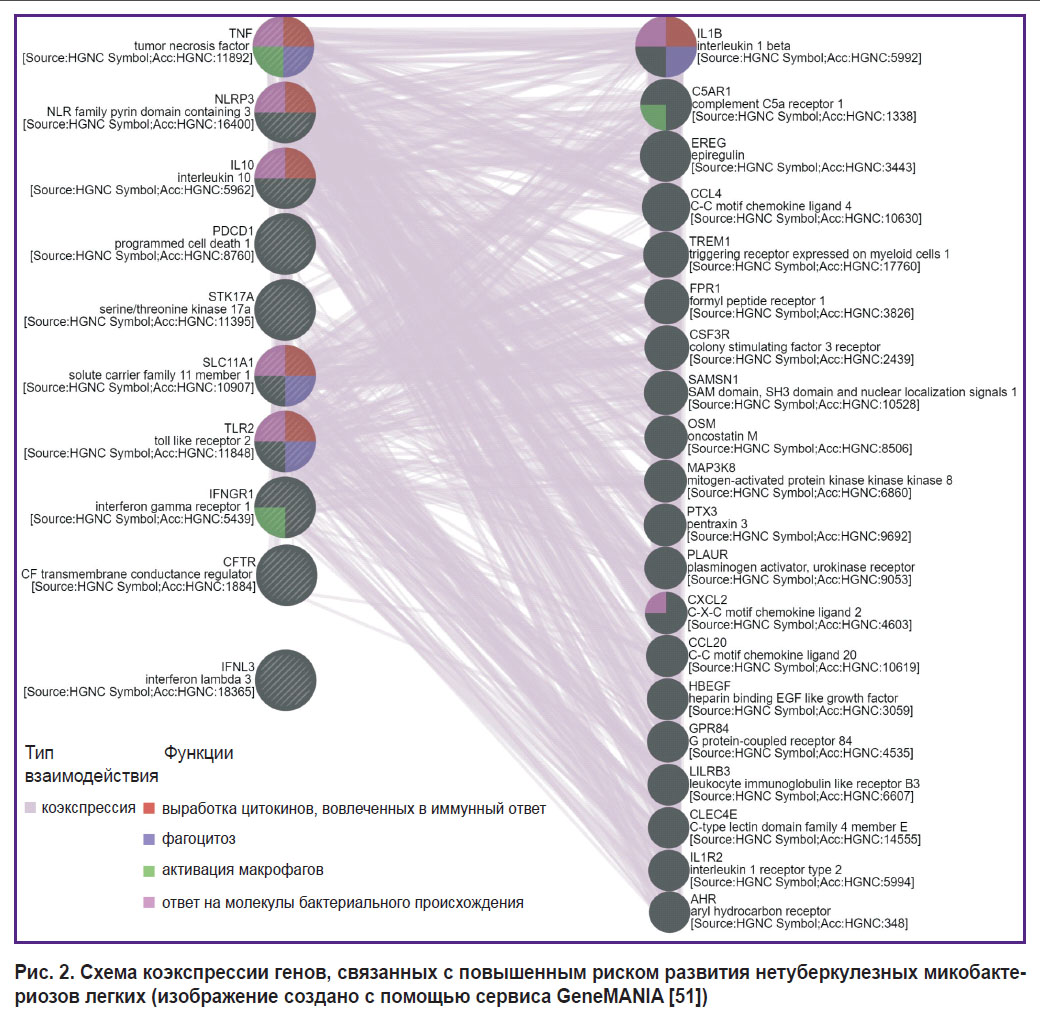
Исходя из представленной выше информации, предполагается связь по меньшей мере 10 генов и полиморфизмов в них с развитием НТМЛ. Приведенный нами в табл. 3 перечень генов можно разделить по функциям кодируемых ими белков. Продукты генов IFNL3, TLR2, IFNGR1, PDCD1, IL10 — регуляторные белки, участвующие в усилении или ингибировании тех или иных иммунных реакций; белки, кодируемые генами SLC11A1 и CFTR, можно отнести к транспортным белкам, обеспечивающим перемещение определенных молекул в зависимости от потребностей клетки; ген NLRP3 кодирует белок воспалительного ответа, являющийся частью инфламмасомного комплекса; продукты генов STK17A, TNF — белки, участвующие в запуске контролируемой клеточной смерти. Среди указанных в этой статье генов, варианты в которых ассоциированы с повышенным риском развития НТМЛ, преобладают гены, вовлекаемые в формирование ответа на молекулы бактериальной природы (рис. 2; создано с помощью GeneMANIA [51]).
|
Рис. 2. Схема коэкспрессии генов, связанных с повышенным риском развития нетуберкулезных микобактериозов легких (изображение создано с помощью сервиса GeneMANIA [51]) |
Несмотря на тщательный отбор и анализ литературных источников, в перечне оказались гены, отвечающие за синтез многофункциональных, часто неспецифических факторов, например цитокинов. Это значительно понижает уровень достоверности интерпретации их роли как предикторов развития НТМЛ и требует дальнейшего, более детального изучения данной проблемы.
Кроме того, каждый из представленных полиморфизмов не оказывает свое действие изолированно, а вносит вклад в риск развития заболевания в контексте общей генетической конституции пациента. В связи с этим особое внимание должно быть уделено изучению взаимодействия генетических факторов между собой и исследованию роли сочетания полиморфизмов генов рецепторов и опсонизирующих белков, участвующих в реакциях врожденного иммунитета. Кроме того, немалый интерес представляет анализ взаимосвязи вариантов генов, коэкспрессирующихся с теми, которые показали ассоциацию с развитием НТМЛ в нашем исследовании (см. рис. 2).
Ассоциация генных вариантов с развитием НТМЛ продемонстрирована в разных популяциях. В связи с этим частота и вклад каждого из упомянутых генетических маркеров могут отличаться среди представителей той или иной популяции. Последнее подчеркивает актуальность изучения взаимосвязи указанных нами полиморфизмов с риском развития НТМЛ в отечественной популяции.
При анализе литературных источников мы не нашли данных о генетических особенностях, способствующих преимущественному развитию инфекционного процесса, обусловленного конкретным видом микобактерий. В опубликованных исследованиях описаны в основном наиболее часто диагностируемые комплексы микобактерий (в первую очередь M. avium-intracellulare complex) — около 50% всех НТМЛ [50, 52]. Возможно, это связано с тем, что заболевания, вызванные отдельными штаммами НТМБ, встречаются в небольшом проценте случаев, что затрудняет получение статистически значимых результатов [53].
Таким образом, вопрос влияния генетических факторов на заболеваемость и течение НТМЛ в зависимости от вызвавшего его возбудителя требует дальнейшего изучения.
Заключение
В ходе анализа литературных источников мы определили 14 генетических вариантов в 10 генах человека, наиболее подходящих на роль факторов повышенного риска развития НТМЛ. В дальнейшем необходимо изучить влияние описанных вариантов на заболеваемость НТМЛ в группах пациентов различных популяций, а также исследовать потенциальное взаимодействие генов между собой. В связи с этим требуется разработка молекулярно-генетической системы для выявления конкретных генетических вариантов. Кроме того, немаловажным шагом является определение частоты рисковых аллелей в российской популяции. Следует также изучить молекулярные эффекты вариантов на широкой выборке пациентов с легочными заболеваниями, вызванными нетуберкулезными микобактериями, с целью установления истинного влияния генетических вариантов на риск повышения заболеваемости НТМЛ.
Вклад авторов. Ф.С. Свиридов — сбор материала, написание текста, ответственность за целостность всех частей статьи; М.М. Литвинова — сбор материала, разработка концепции и дизайна исследования, написание текста, утверждение окончательного варианта статьи, ответственность за целостность всех частей статьи; М.А. Карнаушкина — участие в разработке концепции и дизайна исследования, написание текста, редактирование; Н.Н. Макарьянц — сбор и систематизация материала; М.В. Горбунова — сбор материала, участие в написание текста, подготовке статьи к публикации.
Источник финансирования. Авторы заявляют об отсутствии внешнего финансирования при проведении исследования.
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Литература
- Makarova M.V., Guntupova L.D. Nontuberculous mycobacteria. Bioprep Prev Diagn Treat 2020; 20(2): 97–102, https://doi.org/10.30895/2221-996x-2020-20-2-97-102.
- Sharma S.K., Upadhyay V. Epidemiology, diagnosis & treatment of non-tuberculous mycobacterial diseases. Indian J Med Res 2020; 152(3): 185–226, https://doi.org/10.4103/ijmr.IJMR_902_20.
- Moore J.E., Kruijshaar M.E., Ormerod L.P., Drobniewski F., Abubakar I. Increasing reports of non-tuberculous mycobacteria in England, Wales and Northern Ireland, 1995–2006. BMC Public Health 2010; 10: 612, https://doi.org/10.1186/1471-2458-10-612.
- Brode S.K., Marchand-Austin A., Jamieson F.B., Marras T.K. Pulmonary versus nonpulmonary nontuberculous mycobacteria, Ontario, Canada. Emerg Infect Dis 2017; 23(11): 1898–1901, https://doi.org/10.3201/eid2311.170959.
- Lai C.C., Tan C.K., Chou C.H., Hsu H.L., Liao C.H., Huang Y.T., Yang P.C., Luh K.T., Hsueh P.R. Increasing incidence of nontuberculous mycobacteria, Taiwan, 2000–2008. Emerg Infect Dis 2010; 16(2): 294–296, https://doi.org/10.3201/eid1602.090675.
- Park S.C., Kang M.J., Han C.H., Lee S.M., Kim C.J., Lee J.M., Kang Y.A. Prevalence, incidence, and mortality of nontuberculous mycobacterial infection in Korea: a nationwide population-based study. BMC Pulm Med 2019; 19(1): 140, https://doi.org/10.1186/s12890-019-0901-z.
- Donohue M.J., Wymer L. Increasing prevalence rate of nontuberculous mycobacteria infections in five states, 2008–2013. Ann Am Thorac Soc 2016; 13(12): 2143–2150, https://doi.org/10.1513/AnnalsATS.201605-353OC.
- Белобородова Е.Н., Зимина В.Н., Дегтярева С.Ю., Гизатуллина Ф.Б., Волченков Г.В., Фесенко О.В. Клинико-эпидемиологическая характеристика микобактериоза в двух субъектах Российской Федерации. Инфекционные болезни 2020; 18(1): 35–42.
- Владимирова Е.Б., Шмелев Е.И., Зайцева А.С., Ковалевская М.Н., Касимцева С.А., Амансахедов Р.Б., Черноусова Л.Н., Эргешов А.Э., Шмелева Н.М. Нетуберкулезный микобактериоз легких — возможности диагностики в практике пульмонолога. Терапевтический архив 2019; 91(11): 26–31, https://doi.org/10.26442/00403660.2019.11.000306.
- Zhestkov A.V., Lyamin A.V., Ismatullin D.D., Martinovich A.A., Haykina E.V. Review of the international clinical practice guidelines for the treatment of lung infections caused by nontuberculous mycobacteria. Clin Microbiol Antimicrob Chemother 2021; 23(1): 66–91, https://doi.org/10.36488/cmac.2021.1.66-91.
- Cowman S., van Ingen J., Griffith D.E., Loebinger M.R. Non-tuberculous mycobacterial pulmonary disease. Eur Respir J 2019; 54(1): 1900250, https://doi.org/10.1183/13993003.00250-2019.
- Henkle E., Winthrop K.L. Nontuberculous mycobacteria infections in immunosuppressed hosts. Clin Chest Med 2015; 36(1): 91–99, https://doi.org/10.1016/j.ccm.2014.11.002.
- Koh W.J., Kwon O.J., Kim E.J., Lee K.S., Ki C.S., Kim J.W. NRAMP1 gene polymorphism and susceptibility to nontuberculous mycobacterial lung diseases. Chest 2005; 128(1): 94–101, https://doi.org/10.1378/chest.128.1.94.
- Wu M.F., Shu C.C., Wang J.Y., Yan B.S., Lai H.C., Chiang B.L., Wu L.S., Yu C.J. NLRP3 inflammasome is attenuated in patients with Mycobacterium avium complex lung disease and correlated with decreased interleukin-1β response and host susceptibility. Sci Rep 2019; 9(1): 12534, https://doi.org/10.1038/s41598-019-47609-3.
- Yim J.J., Kim H.J., Kwon O.J., Koh W.J. Association between microsatellite polymorphisms in intron II of the human toll-like receptor 2 gene and nontuberculous mycobacterial lung disease in a Korean population. Hum Immunol 2008; 69(9): 572–576, https://doi.org/10.1016/j.humimm.2008.06.003.
- Jang M.A., Kim S.Y., Jeong B.H., Park H.Y., Jeon K., Kim J.W., Ki C.S., Koh W.J. Association of CFTR gene variants with nontuberculous mycobacterial lung disease in a Korean population with a low prevalence of cystic fibrosis. J Hum Genet 2013; 58(5): 298–303, https://doi.org/10.1038/jhg.2013.19.
- Farnia P., Ghanavi J., Saif S., Farnia P., Velayati A.A. Association of interferon-γ receptor-1 gene polymorphism with nontuberculous mycobacterial lung infection among Iranian patients with pulmonary disease. Am J Trop Med Hyg 2017; 97(1): 57–61, https://doi.org/10.4269/ajtmh.16-0905.
- Cho J., Park K., Choi S.M., Lee J., Lee C.H., Lee J.K., Heo E.Y., Kim D.K., Lee Y.J., Park J.S., Cho Y.J., Yoon H.I., Lee J.H., Lee C.T., Kim N., Choi K.Y., Lee K.H., Sung J., Won S., Yim J.J. Genome-wide association study of non-tuberculous mycobacterial pulmonary disease. Thorax 2021; 76(2): 169–177, https://doi.org/10.1136/thoraxjnl-2019-214430.
- Pan S.W., Feng J.Y., Wu L.S., Shu C.C., Wang P.H., Wang J.Y., Wang H.C., Su W.J., Chen Y.M., Yu C.J. Sex-Specific associations between susceptibility to Mycobacterium avium complex lung disease and programmed cell death 1 gene polymorphisms. J Infect Dis 2023; 228(1): 18–27, https://doi.org/10.1093/infdis/jiac396.
- Affandi J.S., Hendry S., Waterer G., Thomson R., Wallace H., Burrows S., Price P. Searching for an immunogenetic factor that will illuminate susceptibility to non-tuberculous mycobacterial disease. Hum Immunol 2013; 74(10): 1382–1385, https://doi.org/10.1016/j.humimm.2013.06.019.
- Buschman E., Skamene E. From Bcg/Lsh/Ity to Nramp1: three decades of search and research. Drug Metab Dispos 2001; 29(4 Pt 2): 471–473.
- Pauline R., Devaraj D.V., Sivasubramanian J., Velmurugan S., Stephen S.B., Yasam S.K., Kulanthaivel L., Subbaraj G.K. Systemic assessment of solute carrier family 11-member A1 (rs17235409) gene polymorphism and mycobacterium tuberculosis risk in Asian and Caucasian population: a comprehensive updated meta-analysis. Int J Mycobacteriol 2023; 12(4): 467–477, https://doi.org/10.4103/ijmy.ijmy_180_23.
- Barbosa Júnior W.L., Justo A.M., Dos Santos A.M.A., do Carmo R.F., de Melo F.L., Vasconcelos L.R.S., de Medeiros Z.M. SLC11A1 (rs3731865) polymorphism and susceptibility to visceral leishmaniasis in HIV-coinfected patients from Northeastern Brazil. Parasitol Res 2020; 119(2): 491–499, https://doi.org/10.1007/s00436-019-06596-0.
- Goswami T., Bhattacharjee A., Babal P., Searle S., Moore E., Li M., Blackwell J.M. Natural-resistance-associated macrophage protein 1 is an H+/bivalent cation antiporter. Biochem J 2001; 354(Pt 3): 511–519, https://doi.org/10.1042/0264-6021:3540511.
- Waworuntu W., Tanoerahardjo F.S., Mallongi A., Ahmad A., Amin M., Djaharuddin I., Bukhari A., Tabri N.A., Bahar B., Hidayah N., Halik H., Massi M.N. Serum iron levels in tuberculosis patients and household contacts and its association with natural resistance-associated macrophage protein 1 polymorphism and expression. Clin Respir J 2023; 17(9): 893–904, https://doi.org/10.1111/crj.13677.
- Chen Y., Ye X., Escames G., Lei W., Zhang X., Li M., Jing T., Yao Y., Qiu Z., Wang Z., Acuña-Castroviejo D., Yang Y. The NLRP3 inflammasome: contributions to inflammation-related diseases. Cell Mol Biol Lett 2023; 28(1): 51, https://doi.org/10.1186/s11658-023-00462-9.
- Martinon F., Pétrilli V., Mayor A., Tardivel A., Tschopp J. Gout-associated uric acid crystals activate the NALP3 inflammasome. Nature 2006; 440(7081): 237–241, https://doi.org/10.1038/nature04516.
- Dostert C., Pétrilli V., Van Bruggen R., Steele C., Mossman B.T., Tschopp J. Innate immune activation through Nalp3 inflammasome sensing of asbestos and silica. Science 2008; 320(5876): 674–677, https://doi.org/10.1126/science.1156995.
- Theobald S.J., Simonis A., Georgomanolis T., Kreer C., Zehner M., Eisfeld H.S., Albert M.C., Chhen J., Motameny S., Erger F., Fischer J., Malin J.J., Gräb J., Winter S., Pouikli A., David F., Böll B., Koehler P., Vanshylla K., Gruell H., Suárez I., Hallek M., Fätkenheuer G., Jung N., Cornely O.A., Lehmann C., Tessarz P., Altmüller J., Nürnberg P., Kashkar H., Klein F., Koch M., Rybniker J. Long-lived macrophage reprogramming drives spike protein-mediated inflammasome activation in COVID-19. EMBO Mol Med 2021; 13(8): e14150, https://doi.org/10.15252/emmm.202114150.
- Lancioni C.L., Li Q., Thomas J.J., Ding X., Thiel B., Drage M.G., Pecora N.D., Ziady A.G., Shank S., Harding C.V., Boom W.H., Rojas R.E. Mycobacterium tuberculosis lipoproteins directly regulate human memory CD4(+) T cell activation via toll-like receptors 1 and 2. Infect Immun 2011; 79(2): 663–673, https://doi.org/10.1128/IAI.00806-10.
- Jin M.S., Kim S.E., Heo J.Y., Lee M.E., Kim H.M., Paik S.G., Lee H., Lee J.O. Crystal structure of the TLR1-TLR2 heterodimer induced by binding of a tri-acylated lipopeptide. Cell 2007; 130(6): 1071–1082, https://doi.org/10.1016/j.cell.2007.09.008.
- Drage M.G., Pecora N.D., Hise A.G., Febbraio M., Silverstein R.L., Golenbock D.T., Boom W.H., Harding C.V. TLR2 and its co-receptors determine responses of macrophages and dendritic cells to lipoproteins of Mycobacterium tuberculosis. Cell Immunol 2009; 258(1): 29–37, https://doi.org/10.1016/j.cellimm.2009.03.008.
- Enuka Y., Hanukoglu I., Edelheit O., Vaknine H., Hanukoglu A. Epithelial sodium channels (ENaC) are uniformly distributed on motile cilia in the oviduct and the respiratory airways. Histochem Cell Biol 2012; 137(3): 339–353, https://doi.org/10.1007/s00418-011-0904-1.
- Kartner N., Augustinas O., Jensen T.J., Naismith A.L., Riordan J.R. Mislocalization of delta F508 CFTR in cystic fibrosis sweat gland. Nat Genet 1992; 1(5): 321–327, https://doi.org/10.1038/ng0892-321.
- Hanukoglu I., Boggula V.R., Vaknine H., Sharma S., Kleyman T., Hanukoglu A. Expression of epithelial sodium channel (ENaC) and CFTR in the human epidermis and epidermal appendages. Histochem Cell Biol 2017; 147(6): 733–748, https://doi.org/10.1007/s00418-016-1535-3.
- Shah V.S., Meyerholz D.K., Tang X.X., Reznikov L., Abou Alaiwa M., Ernst S.E., Karp P.H., Wohlford-Lenane C.L., Heilmann K.P., Leidinger M.R., Allen P.D., Zabner J., McCray P.B. Jr, Ostedgaard L.S., Stoltz D.A., Randak C.O., Welsh M.J. Airway acidification initiates host defense abnormalities in cystic fibrosis mice. Science 2016; 351(6272): 503–507, https://doi.org/10.1126/science.aad5589.
- Endres T.M., Konstan M.W. What is cystic fibrosis? JAMA 2022; 327(2): 191, https://doi.org/10.1001/jama.2021.23280.
- van de Wetering D., de Paus R.A., van Dissel J.T., van de Vosse E. Functional analysis of naturally occurring amino acid substitutions in human IFN-gammaR1. Mol Immunol 2010; 47(5): 1023–1030, https://doi.org/10.1016/j.molimm.2009.11.016.
- Londino J.D., Gulick D.L., Lear T.B., Suber T.L., Weathington N.M., Masa L.S., Chen B.B., Mallampalli R.K. Post-translational modification of the interferon-gamma receptor alters its stability and signaling. Biochem J 2017; 474(20): 3543–3557, https://doi.org/10.1042/BCJ20170548.
- Sanjo H., Kawai T., Akira S. DRAKs, novel serine/threonine kinases related to death-associated protein kinase that trigger apoptosis. J Biol Chem 1998; 273(44): 29066–29071, https://doi.org/10.1074/jbc.273.44.29066.
- Mao P., Hever M.P., Niemaszyk L.M., Haghkerdar J.M., Yanco E.G., Desai D., Beyrouthy M.J., Kerley-Hamilton J.S., Freemantle S.J., Spinella M.J. Serine/threonine kinase 17A is a novel p53 target gene and modulator of cisplatin toxicity and reactive oxygen species in testicular cancer cells. J Biol Chem 2011; 286(22): 19381–19391, https://doi.org/10.1074/jbc.M111.218040.
- Fife B.T., Pauken K.E. The role of the PD-1 pathway in autoimmunity and peripheral tolerance. Ann N Y Acad Sci 2011; 1217: 45–59, https://doi.org/10.1111/j.1749-6632.2010.05919.x.
- Topalian S.L., Hodi F.S., Brahmer J.R., Gettinger S.N., Smith D.C., McDermott D.F., Powderly J.D., Carvajal R.D., Sosman J.A., Atkins M.B., Leming P.D., Spigel D.R., Antonia S.J., Horn L., Drake C.G., Pardoll D.M., Chen L., Sharfman W.H., Anders R.A., Taube J.M., McMiller T.L., Xu H., Korman A.J., Jure-Kunkel M., Agrawal S., McDonald D., Kollia G.D., Gupta A., Wigginton J.M., Sznol M. Safety, activity, and immune correlates of anti-PD-1 antibody in cancer. N Engl J Med 2012; 366(26): 2443–2454, https://doi.org/10.1056/NEJMoa1200690.
- Sheppard P., Kindsvogel W., Xu W., Henderson K., Schlutsmeyer S., Whitmore T.E., Kuestner R., Garrigues U., Birks C., Roraback J., Ostrander C., Dong D., Shin J., Presnell S., Fox B., Haldeman B., Cooper E., Taft D., Gilbert T., Grant F.J., Tackett M., Krivan W., McKnight G., Clegg C., Foster D., Klucher K.M. IL-28, IL-29 and their class II cytokine receptor IL-28R. Nat Immunol 2003; 4(1): 63–68, https://doi.org/10.1038/ni873.
- Kotenko S.V., Gallagher G., Baurin V.V., Lewis-Antes A., Shen M., Shah N.K., Langer J.A., Sheikh F., Dickensheets H., Donnelly R.P. IFN-lambdas mediate antiviral protection through a distinct class II cytokine receptor complex. Nat Immunol 2003; 4(1): 69–77, https://doi.org/10.1038/ni875.
- Nie H., Zheng Y., Li R., Guo T.B., He D., Fang L., Liu X., Xiao L., Chen X., Wan B., Chin Y.E., Zhang J.Z. Phosphorylation of FOXP3 controls regulatory T cell function and is inhibited by TNF-α in rheumatoid arthritis. Nat Med 2013; 19(3): 322–328, https://doi.org/10.1038/nm.3085.
- Hashimoto S.I., Komuro I., Yamada M., Akagawa K.S. IL-10 inhibits granulocyte-macrophage colony-stimulating factor-dependent human monocyte survival at the early stage of the culture and inhibits the generation of macrophages. J Immunol 2001; 167(7): 3619–3625, https://doi.org/10.4049/jimmunol.167.7.3619.
- Yoon S.I., Logsdon N.J., Sheikh F., Donnelly R.P., Walter M.R. Conformational changes mediate interleukin-10 receptor 2 (IL-10R2) binding to IL-10 and assembly of the signaling complex. J Biol Chem 2006; 281(46): 35088–35096, https://doi.org/10.1074/jbc.M606791200.
- El Kasmi K.C., Smith A.M., Williams L., Neale G., Panopoulos A.D., Watowich S.S., Häcker H., Foxwell B.M., Murray P.J. Cutting edge: a transcriptional repressor and corepressor induced by the STAT3-regulated anti-inflammatory signaling pathway. J Immunol 2007; 179(11): 7215–7219, https://doi.org/10.4049/jimmunol.179.11.7215. Erratum in: J Immunol 2008; 180(5): 3612.
- Chin K.L., Sarmiento M.E., Alvarez-Cabrera N., Norazmi M.N., Acosta A. Pulmonary non-tuberculous mycobacterial infections: current state and future management. Eur J Clin Microbiol Infect Dis 2020; 39(5): 799–826, https://doi.org/10.1007/s10096-019-03771-0.
- Warde-Farley D., Donaldson S.L., Comes O., Zuberi K., Badrawi R., Chao P., Franz M., Grouios C., Kazi F., Lopes C.T., Maitland A., Mostafavi S., Montojo J., Shao Q., Wright G., Bader G.D., Morris Q. The GeneMANIA prediction server: biological network integration for gene prioritization and predicting gene function. Nucleic Acids Res 2010; 38(Suppl 2): W214–W220, https://doi.org/10.1093/nar/gkq537.
- Лепеха Л.Н., Красникова Е.В., Тарасов Р.В., Никитин С.С. Морфологическая диагностика нетуберкулезного микобактериоза в операционном материале пациентов с туберкулемой легкого. Вестник медицинского института «РЕАВИЗ». Реабилитация, врач и здоровье 2023; 13(2): 16–22, https://doi.org/10.20340/vmi-rvz.2023.2.MORPH.2.
- Микобактериозы органов дыхания. Клинические рекомендации. URL: https://spulmo.ru/upload/kr/Microbakterioz_2022.pdf.










