Разработка ДНК-биочипа для индикации вирусных возбудителей внебольничной пневмонии
Цель исследования — разработка ДНК-биочипа для индикации вирусных возбудителей внебольничной пневмонии.
Материалы и методы. Материалом исследования служили мазки слизистой оболочки носо- и ротоглотки пациентов в возрасте от 2 мес до 18 лет с подтвержденным диагнозом «пневмония». Подбор ДНК-зондов для детекции возбудителей выполняли с использованием нуклеотидных последовательностей из баз данных NCBI Nucleotide и GISAID и с помощью собственной программы disprose. Подобранные ДНК-зонды синтезировали на слайды компании CustomArray (США). На модельном пулированном образце, содержащем ДНК аденовируса и РНК коронавируса SARS-CoV-2, осуществляли подбор оптимальной температуры гибридизации, при которой наблюдали максимальный процент зондов со стандартизированными сигналами гибридизации (ССГ) ≥3 Z, а уровни ССГ специфичных зондов превышали уровни ССГ неспецифичных зондов. Далее в ходе гибридизации на биочипы исследуемых образцов при выбранных условиях проводили отбор эффективных ДНК-зондов для детекции вирусных возбудителей внебольничной пневмонии. С помощью ROC-анализа устанавливали пороговые значения уровня сигналов специфичных зондов, превышение которых свидетельствует о наличии возбудителя.
Результаты. Выбрано 544 ДНК-зонда для детекции аденовируса, бокавируса, респираторно-синцитиального вируса, метапневмовируса, вируса парагриппа человека, риновируса и коронавируса. ДНК-зонды были синтезированы на слайды. Установлена оптимальная температура гибридизации ДНК на биочипы (47°С). Составлен перечень эффективных зондов для специфической детекции аденовируса группы B, бокавируса, вируса парагриппа человека типа 3, респираторно-синцитиального вируса, риновируса и коронавируса SARS-CoV-2. Определены пороговые значения уровня ССГ специфичных зондов для детекции возбудителей внебольничной пневмонии в клинических образцах.
Заключение. Разработан ДНК-биочип для индикации вирусных возбудителей внебольничной пневмонии. Составлен перечень специфичных ДНК-зондов и определены пороговые значения уровня их сигналов, превышение которых свидетельствует о наличии соответствующего возбудителя в клиническом образце. Биочипы позволяют оптимизировать лабораторную диагностику вирусных возбудителей внебольничной пневмонии.
Введение
Внебольничная пневмония (ВП) — острое инфекционное воспаление легких, возникающее вне стационара или выявленное в первые двое суток после госпитализации [1]. ВП остается актуальной проблемой здравоохранения в связи с высоким уровнем заболеваемости. В 2023 г. в России заболеваемость составила 498,02 на 100 тыс. населения, среди детского населения — 803,6 на 100 тыс., а среди детей 1–2 лет — 1465,5 на 100 тыс. [2]. В качестве этиологических агентов ВП часто выявляют респираторные вирусы, такие как метапневмовирус (HMPV), респираторно-синцитиальный вирус (HRSV), риновирус (RV), аденовирус (HAdV), вирус парагриппа человека (HPIV), коронавирусы (HCoV) [3–6]. Этиологическая расшифровка ВП критически необходима для повышения эффективности терапии, однако возбудителей этой патологии часто выявляют у лиц без клинических признаков заболевания. Выявление нескольких потенциальных патогенов ВП может свидетельствовать о здоровом носительстве, бессимптомной инфекции или персистенции после заражения [7]. Количественный ПЦР-анализ является одним из надежных методов идентификации преобладающего патогена, который, предположительно, вызывает синдром ВП [8]. С учетом постоянно расширяющегося спектра возбудителей ВП постановка ПЦР-тестов для детекции каждого из них является трудоемкой и дорогостоящей процедурой.
Возможность мультиплексной детекции многих возбудителей различных патологий при оптимальном соотношении производительности и точности результатов, а также стоимости исследования дают ДНК-биочипы [9]. За рубежом были разработаны ДНК-биочипы для детекции широкого спектра бактерий и вирусов, в том числе возбудителей патологий дыхательных путей. Так, ДНК-биочип LLMDA (Lawrence Livermore Microbial Detection Array; Lawrence Livermore National Laboratory, США) способен детектировать 2200 видов вирусов и 900 видов бактерий [10, 11], а ДНК-микрочип Axiom Microbiome Array (Axiom, CША) — 6091 вид бактерий и 4770 видов вирусов [12]. Однако спектр возбудителей, выявляемых с помощью данных ДНК-биочипов, избыточен относительно определения клинически значимых патогенов. При этом их применение затрудняют также высокая стоимость и сложность пробоподготовки с использованием множества специфичных праймеров и нескольких реакционных смесей. Компанией Genomica (Испания) были разработаны ДНК-биочипы низкой плотности CLART для детекции наиболее распространенных вирусных возбудителей ВП (эти биочипы прошли клинические испытания). Они не способны различать носительство и активную инфекцию [13], поэтому является актуальной разработка ДНК-биочипов для детекции вирусных возбудителей ВП, преимуществом которых при возможности детекции множества возбудителей служило бы применение рандомных праймеров, удешевляющих и упрощающих процесс пробоподготовки, а также дифференциальной диагностики активной инфекции и носительства патогена.
Цель исследования — разработка ДНК-биочипа для индикации вирусных возбудителей внебольничной пневмонии.
Материалы и методы
Подбор и синтез ДНК-зондов. С помощью собственного алгоритма disprose [14] были подобраны ДНК-зонды для диагностики аденовирусов групп B, C, E, бокавируса (HBoV), респираторно-синцитиального вируса, метапневмовируса, вирусов парагриппа человека типов 1–4, риновируса, коронавирусов видов HKU-1, OC-43, NL-63, E-229, SARS-CoV-2. Использовали нуклеотидные последовательности возбудителей ВП из баз данных NCBI Nucleotide и GISAID [15, 16]. В результате отобрано 544 целевых ДНК-зонда длиной 25–30 н.о., которые далее были синтезированы на слайды CustomArray Blank Slide 12K (CustomArray, США). Слайд содержал 4 идентичных сектора, в каждом из которых находилось три блока по 544 целевых ДНК-зонда, три блока по 30 ДНК-зондов, специфичных к геному Rhizobium rubi и используемых в качестве отрицательного контроля, и три блока по 30 зондов контроля качества синтеза. Синтез зондов проводили по протоколу производителя с помощью аппарата B3 Synthesizer (CustomArray, США) и комплекта реагентов компаний Merck Sharp & Dohme (США), Sigma (США, Германия, Франция), Panreac (Испания), «Биохим» (Россия).
Материалы исследования. Материалом для исследования являлись мазки слизистой оболочки носо- и ротоглотки пациентов в возрасте от 2 мес до 18 лет с рентгенологически подтвержденным диагнозом «пневмония». В соответствии с Хельсинкской декларацией (2024) врачами медицинских учреждений получено добровольное информированное согласие у больных и представителей несовершеннолетних пациентов.
Отбор образцов для исследования. Диагностику возбудителей ВП проводили с помощью коммерческих наборов «АмплиСенс ОРВИ-скрин-FL» и «АмплиСенс CoV-Bat-FL» (ЦНИИЭ Роспотребнадзора, Россия). В работе использовали клинические образцы, включающие согласно результатам ПЦР-тестирования исключительно ДНК/РНК-содержащие возбудители: аденовируса (10 ед.), бокавируса (8 ед.), коронавируса SARS-CoV-2 (12 ед.), вируса парагриппа человека типа 3 (10 ед.), респираторно-синцитиального вируса (12 ед.) и риновируса (12 ед.). Отрицательным контролем служил пулированный образец мазков слизистой оболочки носо- и ротоглотки здоровых доноров с отрицательным результатом ПЦР-анализа на возбудителей ВП.
Протокол пробоподготовки нуклеиновой кислоты для гибридизации. С использованием коммерческого набора «РИБО-преп» (ЦНИИЭ Роспотребнадзора, Россия) из образцов выделяли тотальную нуклеиновую кислоту (НК) и дополнительно очищали ее переосаждением изопропанолом («Биохим»). Полученную НК разделяли на две части, одну из которых использовали для пробоподготовки ДНК-содержащих возбудителей (HAdV, HBoV), а другую — для РНК-содержащих возбудителей (HPIV, HRSV, RV, HMPV, SARS-CoV-2).
Процесс пробоподготовки образцов с ДНК-содержащими возбудителями ВП проходил в несколько этапов. Проводили фрагментацию тотальной НК с помощью набора NEBNext dsDNA Fragmentase (New England Biolabs, Великобритания). ДНК переосаждали изопропанолом в присутствии 3 M ацетата натрия (рН=7,0). Выполняли амплификацию ДНК с помощью рандомных праймеров Random (dN)10 primer («Евроген», Россия) и набора Encyclo Plus PCR kit («Евроген») на амплификаторе MaxyGene Gradient (Axygen, США). Температурный профиль реакции: 95°С — 2 мин; 25 циклов (95°С — 30 с, 30°С — 30 с, 72°С — 6 мин); 72°С — 8 мин. Полученную ДНК переосаждали изопропанолом в присутствии 3 M ацетата натрия (рН=7,0) и использовали для in vitro транскрипции с помощью рандомных праймеров Random (dN)10 primer («Евроген») и набора реагентов «ДНК-полимераза 1 E. coli (фрагмент Кленова)» (SibEnzyme, Россия). Половину количества дезоксиуридинтрифосфата (dUTP) заменяли на его биотинилированную модификацию Bio-12-dUTP («ДНК-синтез», Россия). Биотин-меченую ДНК переосаждали изопропанолом в присутствии 3 M ацетата натрия (рН=7,0).
В ходе пробоподготовки образцов тотальной НК с РНК-содержащими возбудителями ВП к стандартной процедуре добавляли стадию обратной транскрипции с помощью набора MINT («Евроген») и рандомных праймеров Random (dN)10 primer («Евроген»). Далее следовали амплификация ДНК, фрагментация и транскрипция ДНК с биотин-мечеными нуклеотидами по вышеописанным протоколам.
После прохождения стадий пробоподготовки исследуемого образца с ДНК- и РНК-содержащими возбудителями ВП полученную биотин-меченую ДНК объединяли в один образец и в соответствии с инструкциями производителя (CustomArray, США) гибридизировали на биочип с последующей отмывкой для повторного использования.
Математическая обработка и анализ сигналов гибридизации. С помощью программы Electra Sense Analysis 3.4.2 (CustomArray, США) первичные сигналы гибридизации в формате ECD конвертировали в формат CSV. Для статистической обработки сигналов гибридизации использовали среду программирования R 4.3.1 Beagle Scouts (лицензия GNU3 [17]) и оболочку RStudio 2024.04.2 Chocolate Cosmos (лицензия GNU3 [18]).
Стандартизация сигналов гибридизации. В результате стандартизации полученных первичных сигналов гибридизации получали стандартизированные сигналы гибридизации (ССГ) в Z-единицах. Их число характеризует вариабельность сигнала зонда относительно среднего сигнала пула зондов отрицательного контроля. Расчет проводили по формуле:
Z = (X – MNC)/SDNС,
где Z — стандартизированный сигнал гибридизации; Х — первичный сигнал гибридизации зонда; MNC — среднее арифметическое значение сигналов зондов отрицательного контроля; SDNC — стандартное отклонение сигналов зондов отрицательного контроля. ССГ >3 Z считали эффективными [19], ССГ >4 Z — высокими, ССГ >5 Z — очень высокими, а ССГ >10 Z исключали из анализа, расценивая как неспецифическое/частичное связывание.
Определение оптимальных параметров гибридизации. Оптимальные параметры гибридизации устанавливали в ходе серии экспериментов с гибридизацией модельного образца тотальной НК, пулированного из двух, в одном из которых методом ПЦР был выявлен ДНК-содержащий вирус HAdV, а в другом — РНК-содержащий вирус SARS-Cov-2. Пулированный образец разделяли на 3 аликвоты, каждая из которых проходила цикл пробоподготовки, с дальнейшим объединением в один образец. Таким образом было получено достаточное количество биотин-меченой ДНК для проведения эксперимента.
Ранее, в ходе разработки ДНК-биочипа для индикации бактериальных возбудителей ВП, нами были определены оптимальные параметры гибридизации ДНК на биочип: размер фрагмента целевой ДНК — 300 н.о. и количество гибридизируемой ДНК — 2 мкг [20]. Поскольку известна обратная зависимость специфичности и эффективности гибридизации ДНК от температуры гибридизации [21], с использованием выбранных параметров выполняли подбор температуры гибридизации: 45, 47, 49°С. Каждую комбинацию параметров гибридизации тестировали 6 раз, выполняя последовательные гибридизации ДНК на биочип с последующей отмывкой. Для каждой серии испытаний определяли эффективность гибридизации, отношение сигнал/шум и валидность гибридизации.
Эффективность гибридизации оценивали, рассчитывая процент эффективных зондов с ССГ ≥3 Z от всей совокупности зондов за исключением зондов отрицательного контроля, а также процент эффективных специфичных зондов с ССГ ≥3 Z от всей совокупности специфичных зондов. Под специфичными понимали зонды, предназначенные для детекции HAdV и SARS-Cov-2, содержащихся в модельном образце.
Отношение сигнал/шум рассчитывали как отношение среднего ССГ эффективных зондов (ССГ ≥3 Z) и порогового значения эффективного ССГ (ССГ=3 Z).
Валидность гибридизации оценивали как отношение среднего ССГ эффективных специфичных (ССГ ≥3 Z) и среднего ССГ эффективных неспецифичных (ССГ ≥3 Z) зондов, за исключением зондов отрицательного контроля. Под специфичными понимали зонды, предназначенные для детекции HAdV и SARS-Cov-2. Зонды, предназначенные для детекции других вирусов, рассматривали как неспецифичные.
Оценка воспроизводимости результатов гибридизации. Для ее проведения использовали биотин-меченую ДНК пулированного образца, содержащего ДНК HAdV и РНК SARS-CoV-2. Образец гибридизировали на три разных слайда биочипа и три раза — на один слайд биочипа с последующими отмывками.
Проводили расчет парных коэффициентов корреляции Спирмена (ρ) между сигналами гибридизации зондов, полученных в результате гибридизаций на одном слайде, и коэффициента вариации (Cv) сигналов гибридизации зондов на разных слайдах по формуле:
Cv = (SD/M)·100%,
где Cv — коэффициент вариации, SD — стандартное отклонение ССГ-зонда, M — среднее арифметическое значение ССГ-зонда.
Определение порога значимого сигнала для детекции вирусных возбудителей внебольничной пневмонии. Порогом значимого сигнала (ПЗС) является уровень ССГ зонда, превышение которого расценивается как наличие в образце соответствующего возбудителя ВП. ПЗС устанавливали таким образом, чтобы интерпретация результатов гибридизации ДНК-биочипа была сопоставима с результатами, полученными методом ПЦР-анализа. Нами были использованы клинические образцы пациентов с рентгенологически подтвержденным диагнозом «пневмония», в которых были выявлены возбудители ВП методом ПЦР с помощью тест-систем «АмплиСенс ОРВИ-скрин-FL» и «АмплиСенс CoV-Bat-FL» (ЦНИИЭ Роспотребнадзора). Всего для тестирования были выбраны образцы, содержащие ДНК/РНК вирусных возбудителей ВП — HAdV (10 ед.), HBoV (8 ед.), SARS-CoV-2 (12 ед.), HPIV3 (10 ед.), HRSV (12 ед.) и RV (12 ед.).
После прохождения протокола пробоподготовки ДНК/РНК каждый образец гибридизировали на ДНК-биочип согласно определенным на предыдущем этапе условиям. На основе полученных данных гибридизации проводили ROC-анализ, рассчитывая чувствительность и специфичность результата определения патогенов для набора из тестируемых ПЗС. При расчетах учитывали только эффективные зонды (ССГ ≥3 Z).
Для каждого образца строили ROC-кривую с использованием набора тестируемых пороговых значений ССГ от 3 до 6 Z с шагом 0,1. Для каждого порогового значения ССГ рассчитывали значения специфичности и чувствительности по формулам:
чувствительность = ИП / (ИП + ЛО);
специфичность = ИО / (ИО + ЛП),
где ИП — количество истинно-положительных зондов (предназначенных для детекции возбудителя с ССГ выше тестируемого порогового значения); ЛП — количество ложно-положительных зондов (не предназначенных для детекции возбудителя с ССГ выше тестируемого порогового значения); ИО — количество истинно-отрицательных зондов (не предназначенных для детекции возбудителя с ССГ, ниже или равным пороговому значению); ЛО — количество ложно-отрицательных зондов (предназначенных для детекции возбудителя с ССГ, ниже или равным пороговому значению).
Под построенными ROC-кривыми находили площадь AUC (area under curve) и определяли пороговые значения ССГ в точке с максимальным значением индекса Юдена (оптимального соотношения чувствительности и специфичности) и в точке с максимальной специфичностью теста. Найденные пороговые значения ССГ усредняли для набора образцов, содержащих один патоген. Полученное усредненное пороговое значение ССГ, соответствующее точке максимальной специфичности, считали ПЗС для детекции конкретного возбудителя.
Для всех протестированных образцов проверяли наличие неспецифичных зондов, ССГ которых превышал установленные ранее ПЗС. В качестве отрицательного контроля использовали образцы мазков слизистой оболочки носо- и ротоглотки практически здоровых доноров без клинических и лабораторных признаков ВП, ПЦР-тестирование которых показало отрицательный результат (n=6).
Выбор оптимальных зондов для специфической детекции вирусных возбудителей внебольничной пневмонии. Из всех зондов, составляющих дизайн ДНК-биочипа, для детекции каждого возбудителя отбирали оптимальные специфичные зонды. Для этого при гибридизации набора образцов, содержащих определенный патоген, выявляли эффективные специфичные зонды, ССГ которых превышал ПЗС гибридизации специфичных к патогену зондов, установленный ранее. Рассчитывали усредненное значение ССГ каждого из таких зондов и его активность — процент образцов, при гибридизации которых ССГ зонда превысил ПЗС. Оптимальный набор специфичных зондов для детекции каждого патогена определяли путем максимизации двух параметров.
С помощью программы BLASTN из пакета BLAST+ 2.10.0 [22] для каждого выбранного зонда устанавливали область его происхождения — фрагмент референсного генома возбудителя, на который с идентичностью 100% выравнивается данный зонд. Аннотацию фрагментов референсных геномов выполняли с помощью базы данных NCBI Nucleotide [15].
Статистическая обработка данных. Расчеты проводили в свободно распространяемой среде программирования R 4.3.1 Beagle Scouts (лицензия GNU3 [17]) с использованием оболочки RStudio 2024.04.2 Chocolate Cosmos (лицензия GNU3) [18]. Описательную статистику приводили с указанием медианы (Me), 1-го и 3-го квартилей (Q1 и Q3). Показатели воспроизводимости оценивали с помощью коэффициента корреляции Спирмена (ρ) и коэффициента вариации (Cv). Чувствительность и специфичность результатов гибридизации оценивали с помощью построения ROC-кривых с определением показателей AUC и индекса Юдена. Для сравнения случайных переменных двух групп использовали критерий Манна–Уитни, разницу приводили с указанием 95% доверительного интервала. При выполнении множественных сравнений рассчитанные уровни статистической значимости корректировали с применением поправки Бенджамини–Хохберга, различия считали статистически значимыми при значении скорректированного «р» менее 0,05.
Результаты
Разработка дизайна ДНК-биочипа для индикации вирусных возбудителей внебольничной пневмонии. В ходе разработки дизайна ДНК-биочипа с помощью собственной программы disprose отбирали нуклеотидные последовательности ДНК-зондов для детекции аденовируса, бокавируса, респираторно-синцитиального вируса, метапневмовируса, вируса парагриппа человека, риновируса и коронавируса. С помощью баз данных NCBI Nucleotide и GISAID формировали локальные банки целевых и неспецифичных нуклеотидных последовательностей. С целевыми последовательностями должна происходить эффективная гибридизация ДНК-зондов, а с нецелевыми их гибридизация нежелательна. Выбирали нуклеотидные последовательности референсных аннотированных геномов основных видов вирусных возбудителей ВП (материнские последовательности) и разделяли их на участки установленной длины, формируя пул кандидатных зондов. Для дальнейшего отбора зондов использовали следующие физико-химические параметры: длина — 24–32 н.о., содержание гуанина и цитозина — 40–60%, количество гомоповторов <5, минимальная энергия фолдинга ≥0 ккал/моль, температура плавления — 55–60°С. Проводили выравнивание отобранных зондов на целевую и неспецифическую базу последовательностей по алгоритму BLAST. Далее отбирали специфичные зонды с полным покрытием целевых последовательностей (100% без пробелов) при отсутствии перекрестной гибридизации с нецелевыми последовательностями (покрытие <50%). Дизайн биочипа составил 544 целевых ДНК-зонда длиной 25–30 н.о. Зонды были синтезированы на слайды.
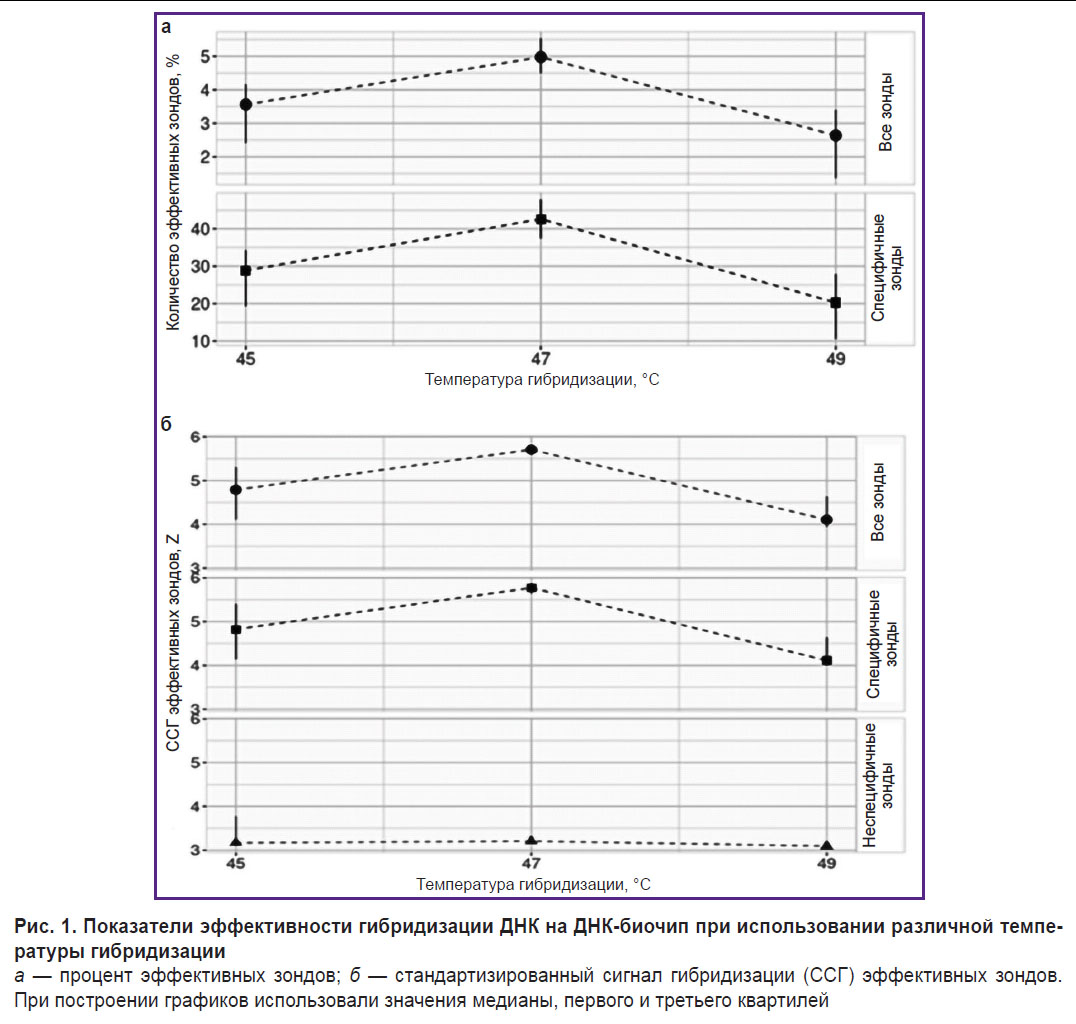
Подбор температуры гибридизации ДНК на биочипы. После прохождения пробоподготовки пулированный образец, содержащий, согласно ПЦР-тесту, ДНК HAdV и РНК SARS-CoV-2, гибридизировали на биочип и осуществляли подбор различных температурных режимов гибридизации (45, 47 или 49°С). Зонды для детекции HAdV выявляли только вариант HAdVB, поэтому далее под специфичными зондами понимали только зонды, предназначенные для детекции HAdVB и SARS-CoV-2. Оптимальная температура гибридизации, при которой регистрировали самый высокий процент эффективных зондов и самые высокие значения ССГ эффективных зондов, составила 47°С (рис. 1).
Оценка воспроизводимости результатов гибридизации. Разработанный ДНК-биочип показал высокую воспроизводимость сигналов гибридизации. Парные коэффициенты корреляции сигнала гибридизации между разными биочипами, а также при повторном использовании одного биочипа составляли более 0,90.
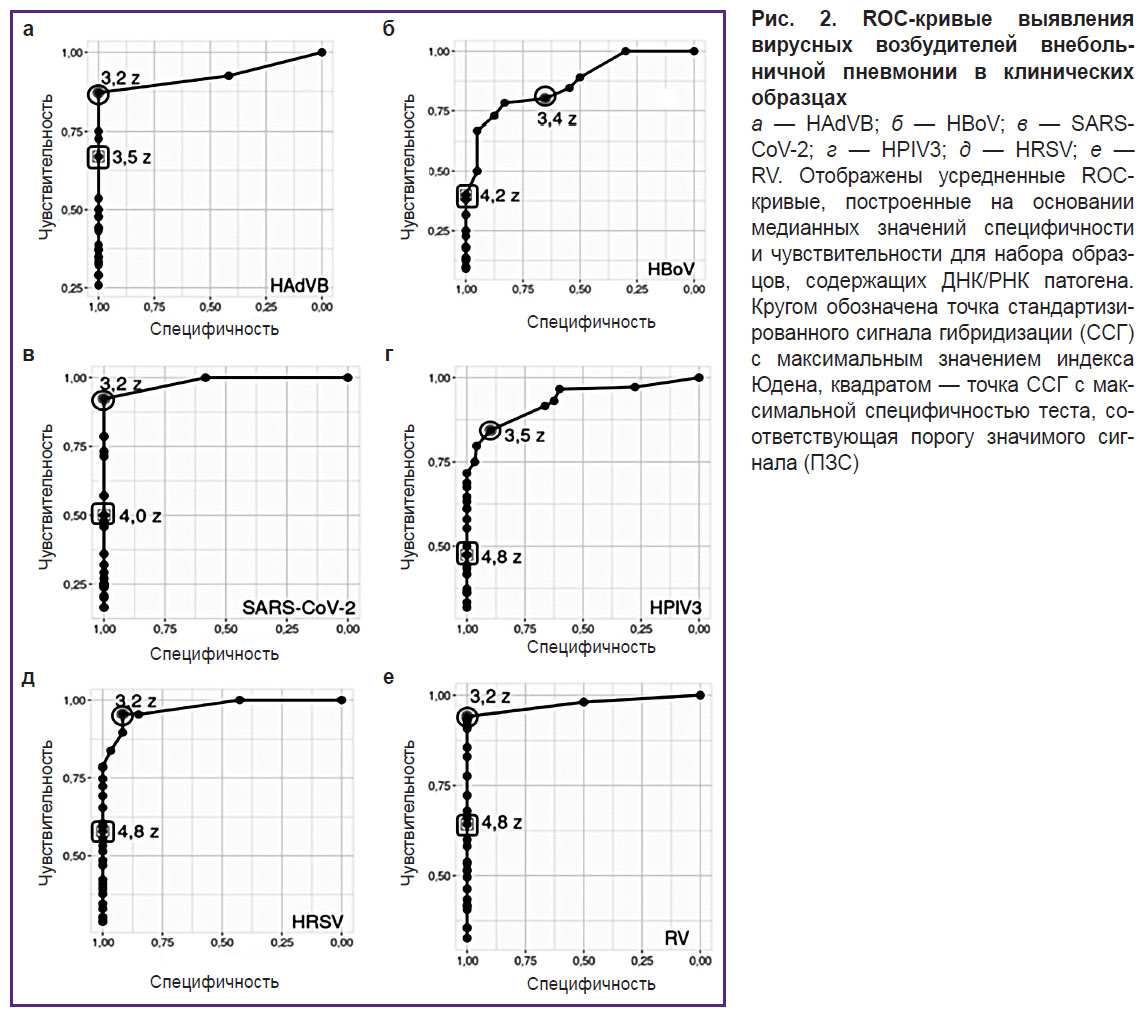
Определение порога значимого сигнала для детекции вирусных возбудителей внебольничной пневмонии. Для шести вирусных возбудителей ВП (HAdVB, HВoV, HPIV3, HRSV, RV и SARS-CoV-2) с помощью ROC-анализа исследовали диапазоны сигналов гибридизации специфичных зондов и определяли ПЗС зондов, превышение ССГ которых интерпретировали как наличие ДНК/РНК патогена в образце. Для каждого возбудителя найденные средние значения ПЗС превышали средние значения индекса Юдена (J): HAdVB — ПЗС=3,5 Z; J=3,2 Z; для HBoV — ПЗС=4,2 Z; J=3,4 Z; для SARS-CoV-2 — ПЗС=4,0 Z; J=3,2 Z; для HPIV3 — ПЗС=4,8 Z; J=3,5 Z; для HRSV — ПЗС=4,8 Z; J=3,2 Z; для RV — ПЗС=4,8 Z; J=3,2 Z (рис. 2).
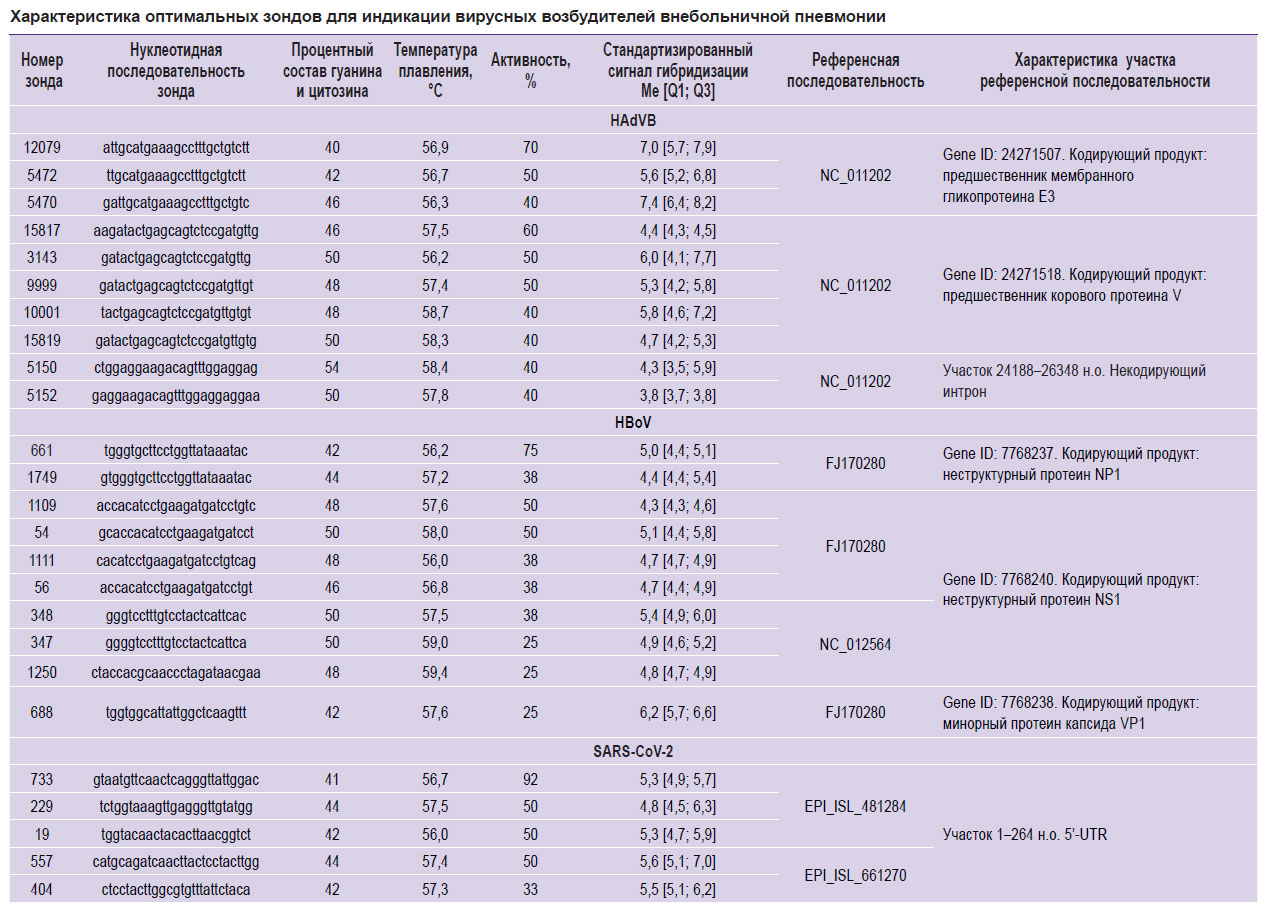
Выбор оптимальных зондов для специфической детекции вирусных возбудителей внебольничной пневмонии. В результате тестирования для каждого возбудителя (HAdVB, HВoV, HPIV3, HRSV, RV и SARS-CoV-2) были выбраны зонды с максимальным средним ССГ и максимальной активностью, которые мы определили как оптимальные для специфической детекции патогена с помощью ДНК-биочипов. Анализ областей происхождения выбранных зондов показал, что большинство из них происходят из кодирующих участков вирусных геномов либо некодирующей области 5’-UTR (см. таблицу).
|
Характеристика оптимальных зондов для индикации вирусных возбудителей внебольничной пневмонии |
Обсуждение
На первом этапе разработки ДНК-биочипа с использованием баз данных GenBank и GISAID и собственной программы disprose был проведен отбор ДНК-зондов для индикации основных циркулирующих в мире в настоящее время вирусных возбудителей ВП. Выбранные 544 ДНК-зонда синтезировали на слайды. Далее тестировали температуру гибридизации пулированного образца, содержащего ДНК HAdVB и РНК SARS-CoV-2, на разработанный биочип. При оптимальной температуре гибридизации 47°С регистрировали самые высокие проценты эффективных зондов и самые высокие значения ССГ эффективных зондов (см. рис. 1). Выбранная температура гибридизации совпадала с оптимальной температурой гибридизации для ранее разработанного биочипа, ориентированного на выявление бактериальных возбудителей ВП [20]. Это свидетельствует, что в дальнейшем возможна разработка универсального биочипа для детекции бактериальных и вирусных возбудителей ВП с унифицированными параметрами гибридизации ДНК. Отметим, что введение в протокол пробоподготовки дополнительной стадии обратной транскрипции для РНК-содержащих возбудителей ВП не оказывает влияния на эффективность гибридизации.
Парные коэффициенты корреляции сигнала гибридизации между разными биочипами и при повторном использовании одного биочипа соответствовали рекомендованному в литературе значению показателя — не менее 0,90 [23]. Это свидетельствует о возможности повторного использования разработанных ДНК-биочипов без потери качества гибридизации, что удешевляет стоимость исследования.
Для шести вирусных возбудителей ВП (HAdVB, HВoV, HPIV3, HRSV, RV и SARS-CoV-2) ROC-анализ при определении ПЗС показал, что на усредненных ROC-кривых (см. рис. 2) для всех возбудителей значение ССГ в точке ПЗС выше, чем в точке Юдена (оптимальное значение чувствительности и специфичности). Произвольное занижение чувствительности ДНК-биочипа было допущено нами с целью отсечения части сигналов эффективных специфичных зондов при возможном бессимптомном носительстве патогена. По данным литературы [7, 8], респираторные вирусы, в том числе возбудители ВП, детектируются с помощью молекулярно-генетических методов и у клинически здоровых лиц без симптомов заболевания, однако количество ДНК/РНК патогена при носительстве меньше, чем при активной инфекции. Вероятность специфической гибридизации НК патогена на ДНК-биочип при носительстве ниже, чем при активной инфекции, а эффективная гибридизация при носительстве будет регистрироваться только с единичными зондами спота (поверхности биочипа, на которой синтезированы молекулы одного зонда). Хотя концентрация молекулы-мишени не оказывает влияния на силу гибридизации одной молекулы зонда [24], она определяет долю прореагировавших молекул зондов спота, совокупный сигнал гибридизации которых интерпретируется нами как сигнал гибридизации соответствующего зонда [25]. В итоге низкая концентрация ДНК/РНК возбудителя в образце при носительстве приводит к неполной гибридизации зондов спота и снижению уровня сигнала гибридизации, что позволяет нам различать ССГ специфичных зондов при носительстве и активной инфекции.
Таким образом, завышение значения ПЗС позволяет исключить из трактовки результатов гибридизации ДНК-биочипа сигналы неспецифической гибридизации и сигналы связывания специфичных зондов в случае здорового носительства. Сопутствующие потери чувствительности можно считать допустимыми, поскольку итоговая трактовка результатов гибридизации носит качественный характер (выявлено–не выявлено) и не зависит от количества эффективных специфичных зондов, превысивших ПЗС. Различия значений ПЗС для разных патогенов подтверждают тот факт, что значение ПЗС не является универсальным для всех определяемых биочипом патогенов, и это требует индивидуального расчета ПЗС для каждого из тестируемых возбудителей ВП.
Для индикации каждого патогена (HAdVB, HВoV, HPIV3, HRSV, RV и SARS-CoV-2) нами был выбран набор зондов с максимальным средним ССГ и максимальной активностью (см. таблицу). Большинство выбранных зондов не обладали 100% активностью, и при гибридизации разных образцов, содержащих ДНК/РНК одного патогена, положительный сигнал регистрировали от разных наборов специфичных зондов. По данным литературы [24], различные ДНК-зонды, специфичные для одной молекулярной мишени и даже для одного участка мишени, различаются аффинностью и, соответственно, уровнем сигнала гибридизации. При гибридизации разных образцов уровень сигнала каждого из специфичных зондов является непостоянным и даже не всегда однозначно трактуется как положительный, что обосновывает использование пула специфичных зондов в дизайне ДНК-биочипа. Большинство выбранных нами ДНК-зондов происходили из кодирующих участков вирусного генома либо области 5’-UTR. Поскольку последовательности зондов подбирались таким образом, чтобы специфически взаимодействовать с генетическими последовательностями вирусов вне зависимости от их штамма, геноварианта и других индивидуальных особенностей [14], участки происхождения зондов могут рассматриваться как консервативные области вирусных геномов, пригодные для разработки молекулярно-диагностических тестов, но не подходящие для решения задач по типированию возбудителей ВП.
Таким образом, ДНК-биочип обеспечивает эффективную индикацию вирусных возбудителей ВП, где каждый патоген детектируется набором специфичных зондов. Применение порогов значимого сигнала зондов позволяет дифференцировать клинически значимую инфекцию и носительство возбудителей ВП. За счет применения рандомных праймеров обеспечиваются унификация пробоподготовки биоматериала и уменьшение трудозатрат. ДНК-биочип подходит для многоразового использования, что также снижает себестоимость анализа.
Заключение
В ходе разработки ДНК-биочипа для индикации вирусных возбудителей внебольничной пневмонии были подобраны и синтезированы на слайды ДНК-зонды для детекции аденовируса, бокавируса, метапневмовируса, вируса парагриппа человека, риновируса и коронавируса. Выбрана оптимальная температура гибридизации ДНК (47°С), при которой детектируются достаточно эффективные, специфические и воспроизводимые сигналы гибридизации. Рассчитаны пороговые значения значимых сигналов зондов для специфической детекции выбранных вирусов, позволяющие интерпретировать результаты гибридизации идентично результатам ПЦР-анализа. Определен перечень зондов для специфической детекции данных вирусов, характеризующихся эффективным сигналом гибридизации. Разработанный ДНК-биочип может использоваться в качестве современного инструмента для лабораторной диагностики и мониторинга актуальных вирусных возбудителей ВП.
Источники финансирования. Данное исследование профинансировано из средств государственного бюджета в рамках выполнения отраслевой научно-исследовательской программы Роспотребнадзора на период 2021–2025 гг. «Научное обеспечение эпидемиологического надзора и санитарной охраны территории Российской Федерации. Создание новых технологий, средств и методов контроля и профилактики инфекционных и паразитарных болезней».
Конфликт интересов. Авторы заявляют об отсутствии конфликта интересов.
Литература
- Watkins R.R., Lemonovich T.L. Diagnosis and management of community-acquired pneumonia in adults. Am Fam Physician 2011; 83(11): 1299–1306.
- О состоянии санитарно-эпидемиологического благополучия населения в Российской Федерации в 2023 году: государственный доклад. М: Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека; 2024. URL: https://rospotrebnadzor.ru/upload/iblock/fbc/sd3prfszlc9c2r4xbmsb7o3us38nrvpk/ Gosudarstvennyy-doklad-_O-sostoyanii-sanitarno_ epidemiologicheskogo-blagopoluchiya-naseleniya-v- Rossiyskoy-Federatsii-v-2023-godu_..pdf.
- Holter J.C., Müller F., Bjørang O., Samdal H.H., Marthinsen J.B., Jenum P.A., Ueland T., Frøland S.S., Aukrust P., Husebye E., Heggelund L. Etiology of community-acquired pneumonia and diagnostic yields of microbiological methods: a 3-year prospective study in Norway. BMC Infect Dis 2015; 15: 64, https://doi.org/10.1186/s12879-015-0803-5.
- Waterer G.W. Diagnosing viral and atypical pathogens in the setting of community-acquired pneumonia. Clin Chest Med 2017; 38(1): 21–28, https://doi.org/10.1016/j.ccm.2016.11.004.
- Wiemken T.L., Peyrani P., Ramirez J.A. Global changes in the epidemiology of community-acquired pneumonia. Semin Respir Crit Care Med 2012; 33(3): 213–219, https://doi.org/10.1055/s-0032-1315633.
- Shah B.A., Ahmed W., Dhobi G.N., Shah N.N., Khursheed S.Q., Haq I. Validity of pneumonia severity index and CURB-65 severity scoring systems in community acquired pneumonia in an Indian setting. Indian J Chest Dis Allied Sci 2010; 52(1): 9–17.
- Meyer Sauteur P.M. Childhood community-acquired pneumonia. Eur J Pediatr 2024; 183(3): 1129–1136, https://doi.org/10.1007/s00431-023-05366-6.
- Jansen R.R., Wieringa J., Koekkoek S.M., Visser C.E., Pajkrt D., Molenkamp R., de Jong M.D., Schinkel J. Frequent detection of respiratory viruses without symptoms: toward defining clinically relevant cutoff values. J Clin Microbiol 2011; 49(7): 2631–2636, https://doi.org/10.1128/JCM.02094-10.
- Eads B., Cash A., Bogart K., Costello J., Andrews J. Troubleshooting microarray hybridizations. Methods Enzymol 2006; 411: 34–49, https://doi.org/10.1016/S0076-6879(06)11003-4.
- Gardner S.N., Jaing C.J., McLoughlin K.S., Slezak T.R. A microbial detection array (MDA) for viral and bacterial detection. BMC Genomics 2010; 11: 668, https://doi.org/10.1186/1471-2164-11-668.
- Thissen J.B., McLoughlin K., Gardner S., Gu P., Mabery S., Slezak T., Jaing C. Analysis of sensitivity and rapid hybridization of a multiplexed microbial detection microarray. J Virol Methods 2014; 201: 73–78, https://doi.org/10.1016/j.jviromet.2014.01.024.
- Thissen J.B., Be N.A., McLoughlin K., Gardner S., Rack P.G., Shapero M.H., Rowland R.R.R., Slezak T., Jaing C.J. Axiom microbiome array, the next generation microarray for high-throughput pathogen and microbiome analysis. PLoS One 2019; 14(2): e0212045, https://doi.org/10.1371/journal.pone.0212045.
- Ferreira I.C.S., Alegretti A.P., De Paris F., Paiva R.M., Chakr V.C.B.G. Comparison of a direct immunofluorescence assay (Oxoid IMAGEN®) and a multiplex RT-PCR DNA microarray assay (CLART® PneumoVir) for the detection of respiratory viruses in hospitalized children. J Virol Methods 2020; 284: 113930, https://doi.org/10.1016/j.jviromet.2020.113930.
- Filatova E.N., Chaikina A.S., Brusnigina N.F., Makhova M.A., Utkin O.V. An Algorithm for the selection of probes for specific detection of human disease pathogens using the DNA microarray technology. Sovremennye tehnologii v medicine 2022; 14(1): 6–12, https://doi.org/10.17691/stm2022.14.1.01.
- National Center for Biotechnology Information. Nucleotide. URL: https://www.ncbi.nlm.nih.gov/nucleotide.
- The GISAID database. URL: https://www.gisaid.org.
- R Core Team. R: a language and environment for statistical computing. Vienna: R Foundation for Statistical Computing; 2022. URL: https://www.R-project.org.
- RStudio Team (2020). RStudio: integrated development for R. Boston: RStudio, PBC; 2020. URL: http://www.rstudio.com.
- Liu R.H., Dill K., Fuji H.S., McShea A. Integrated microfluidic biochips for DNA microarray analysis. Expert Rev Mol Diagn 2006; 6(2): 253–261, https://doi.org/10.1586/14737159.6.2.253.
- Sakharnov N.A., Filatova E.N., Popkova M.I., Slavin S.L., Utkin O.V. Development of DNA microarray for parallel detection of community-acquired pneumonia bacterial pathogens. Sovremennye tehnologii v medicine 2024; 16(2): 16–26, https://doi.org/10.17691/stm2024.16.2.02.
- You Y.H., Wang P., Wang Y.H., Wang H.B., Yu D.Z., Hai R., Zhang J.Z. Assessment of comparative genomic hybridization experiment by an in situ synthesized CombiMatrix microarray with Yersinia pestis vaccine strain EV76 DNA. Biomed Environ Sci 2010; 23(5): 384–390, https://doi.org/10.1016/S0895-3988(10)60080-3.
- Camacho C., Coulouris G., Avagyan V., Ma N., Papadopoulos J., Bealer K., Madden T.L. BLAST+: architecture and applications. BMC Bioinformatics 2009; 10:421, https://doi.org/10.1186/1471-2105-10-421.
- Raman T., O’Connor T.P., Hackett N.R., Wang W., Harvey B.G., Attiyeh M.A., Dang D.T., Teater M., Crystal R.G. Quality control in microarray assessment of gene expression in human airway epithelium. BMC Genomics 2009; 10: 493, https://doi.org/10.1186/1471-2164-10-493.
- Ghindilis A.L., Smith M.W., Schwarzkopf K.R., Roth K.M., Peyvan K., Munro S.B., Lodes M.J., Stöver A.G., Bernards K., Dill K., McShea A. CombiMatrix oligonucleotide arrays: genotyping and gene expression assays employing electrochemical detection. Biosens Bioelectron 2007; 22(9-10): 1853–1860, https://doi.org/10.1016/j.bios.2006.06.024.
- Ashwood B., Jones M.S., Radakovic A., Khanna S., Lee Y., Sachleben J.R., Szostak J.W., Ferguson A.L., Tokmakoff A. Thermodynamics and kinetics of DNA and RNA dinucleotide hybridization to gaps and overhangs. Biophys J 2023; 122(16): 3323–3339, https://doi.org/10.1016/j.bpj.2023.07.009.