Радиомика и машинное обучение в диагностике глиальных опухолей головного мозга: систематический обзор и метаанализ
Глиальные опухоли являются наиболее распространенными новообразованиями головного мозга нейроэпителиального ряда. В связи с этим целесообразным представляется поиск высокоинформативных неинвазивных методов типирования данных опухолей, в первую очередь с помощью современной мультимодальной нейровизуализации и технологий радиомики.
Настоящий систематический обзор научной литературы с метаанализом проведен с целью определить основные задачи применения радиомики и машинного обучения в диагностике глиальных опухолей по данным МРТ, а также оценить качество такой неинвазивной диагностики.
Проанализированы 42 публикации, в которых на основе методов радиомики и машинного обучения по данным МР-изображений глиальных опухолей прогнозировали статус молекулярных биомаркеров, включая мутации генов IDH, ATRX, BRAF, H3K27M, промотора TERT, коделецию 1p/19q, метилирование промотора гена MGMT, пролиферативную активность (индекс мечения Ki-67) и др. Обобщенная точность таких методов оказалась высокой и составила 0,86 [0,83; 0,89]. Тем не менее в работах выявлена существенная методологическая гетерогенность, в частности связанная с отсутствием единых стандартов выбора положения, размера и формы зоны интереса для извлечения лучевых биомаркеров, что является серьезным препятствием для воспроизведения полученных экспериментальных результатов в клинической практике. Следовательно, вопрос стандартизации методов радиомики остается актуальным для дальнейших исследований глиальных опухолей.
Введение
Глиальные опухоли (глиомы) являются наиболее распространенными новообразованиями головного мозга нейроэпителиального ряда [1]. В последние годы, благодаря достижениям молекулярной генетики, усложнились представления о генотипе глиом, их биологической неоднородности, а также биомаркерах, связанных с эффективностью лечения и продолжительностью жизни пациентов [2]. Современные стандарты диагностики и лечения глиом в большинстве случаев опираются на результаты биопсии опухоли, в том числе на данные молекулярно-генетического анализа.
В то же время актуальным остается поиск высокоинформативных неинвазивных методов типирования глиальных опухолей, в первую очередь с помощью современной мультимодальной нейровизуализации. В основе исследований с помощью данного метода лежит базовая гипотеза: лучевой фенотип отражает гистологические и молекулярные характеристики биологической ткани. Таким образом, количественные свойства форм и текстур медицинских изображений можно рассматривать как потенциальные биомаркеры. Технологии компьютерных наук, с помощью которых проводится поиск лучевых маркеров — коррелятов биологических характеристик тканей, получили название «радиомика» и (в контексте предсказания генетических признаков патологии) «радиогеномика» [3].
Моделирование сложной взаимосвязи между большим количеством лучевых характеристик и биологическими свойствами опухоли сегодня можно провести с помощью технологий машинного обучения [4]. Машинное обучение — это способ получения математических моделей с использованием большого количества накопленных данных. Математические модели буквально «обучаются» диагностировать опухоль по медицинскому изображению на достаточном количестве клинических примеров с известными диагнозами [5]. Наиболее доступным и информативным методом первичной неинвазивной диагностики глиом является магнитно-резонансная томография (МРТ) в разных модальностях.
Настоящий систематический обзор научной литературы с метаанализом проведен с целью определить основные задачи применения методов радиомики и машинного обучения в диагностике глиальных опухолей по данным МРТ, а также оценить качество такой неинвазивной диагностики.
Материалы и методы
Исследование проведено в соответствии с международными рекомендациями PRISMA (PreferredReportingItemsforSystematicReviewsandMeta-Analyses) по подготовке систематических обзоров и метаанализов [6].
Для анализа были отобраны все научные публикации, соответствующие следующим критериям:
1) работа индексируется в базе данных PubMed;
2) статья опубликована в научном журнале;
3) статья написана на английском языке;
4) тип статьи — оригинальное научное исследование;
5) работа посвящена количественному исследованию МРТ глиальных опухолей головного мозга человека;
6) в работе проводится расчет параметров радиомики по данным МРТ;
7) техническая задача исследования — классификация МР-изображений;
8) для классификации МР-изображений используются алгоритмы машинного (в том числе «глубокого») обучения;
9) расчет метрик качества машинного обучения проведен на тестовой выборке;
10) доступность полного текста статьи.
Таким образом, в анализ не включали исследования, направленные на дифференциальную диагностику глиом и другой патологии (метастазов и других опухолей нервной системы, постлучевой псевдопрогрессии, воспалительного процесса и др.), систематические обзоры и метаанализы литературных данных, неопубликованные статьи и материалы конференций.
Стратегия поиска литературы
Поиск литературных данных был выполнен с помощью поисковой машины национальной медицинской библиотеки США PubMed (https://pubmed.ncbi.nlm.nih.gov/, дата последнего обращения — 20.08.2023 г.) по следующему запросу: «featur* AND (deep learning OR DL OR machine learning OR ML) AND (biomark* OR histol* OR mutat* OR genotyp*) AND (stratif* OR classification OR grad* OR subtyp*) AND (radiomic*[Title/Abstract] OR radiogenomic*[Title/Abstract]) AND (gliom* OR glial) AND (MRI OR magnetic resonance)».
По результатам запроса получены сведения о 71 статье на английском языке с полнотекстовым доступом к 70 статьям. Первичный скрининг найденных работ на соответствие критериям включения был независимо проведен одним из авторов настоящей статьи. Далее другим автором был независимо выполнен контрольный скрининг (см. раздел «Вклад авторов»). Разногласия между авторами по включению работ в анализ разрешали на основе консенсуса. Диаграмма с результатами отбора представлена на рис. 1.
 |
Рис. 1. Диаграмма, отражающая процесс отбора работ для систематического обзора и метаанализа в соответствии с международными рекомендациями PRISMA |
Для анализа были отобраны 42 статьи [7–48], полностью соответствующие критериям включения.
Статистический анализ данных
В работе использованы методы описательной статистики и метаанализа. Распределение непрерывных случайных величин характеризовали с помощью медианы, 25% и 75% квантилей [Q1; Q3]. Категориальные переменные представлены в процентном формате. Для оцениваемых величин рассчитывали 95% доверительные интервалы (95% ДИ).
С помощью процедур метаанализа получали обобщенную оценку точности, чувствительности и специфичности моделей машинного обучения по всей выборке публикаций и отдельным их подгруппам. Для унификации расчетов из каждой публикации мы извлекли количество истинно-положительных, истинно-отрицательных, ложноположительных и ложноотрицательных результатов прогноза в тестовых выборках (явно или с помощью вычислений на основании представленных в статье данных). Результаты статистического обобщения вышеуказанных метрик представляли с помощью графика типа «лесной график» (forestplot). Гетерогенность выборок оценивали с помощью метрики I2. В силу относительно небольших значений изучаемых эффектов для расчета обобщенного размера эффекта использовали промежуточное логит-преобразование и обобщенную линейную модель смешанных эффектов.
Анализ данных проводили с помощью языка статистического программирования R (версия 4.2.1) в интегрированной среде разработки RStudio Server. Для анализа использовали пакеты tidyverse, meta, metafor, dmetar.
Результаты
В основе исследований, изложенных в отобранных статьях, лежало получение математических моделей для предсказания разных целевых переменных с помощью машинного обучения по данным радиомики. Целевыми переменными в изученных работах были молекулярные биомаркеры: статусы мутации генов IDH (n=23), ATRX (n=4), TERT (n=3), BRAF (n=1), мутации H3K27M (n=2), амплификация EGFR (n=1), метилирование промотора MGMT (n=3), экспрессия Ki-67 (n=1), степень злокачественности глиомы по классификации ВОЗ (WHOGrade, n=2), коделеция 1p/19q (n=6) и некоторые другие характеристики молекулярного профиля глиом. В качестве предикторов был использован большой спектр лучевых биомаркеров, включавший статистики первого порядка (firstorderfeatures), а также количественные показатели, рассчитанные по матрицам совместной встречаемости уровней серого (graylevelco-occurrencematrix, GLCM), длине пробегов уровней серого (graylevelrunlengthmatrix, GLRLM), матрицам различий соседних уровней серого (neighbouringgraytonedifferencematrix, NGDTM), матрицам разноразмерных зон уровней серого (graylevelsizezonematrix, GLSZM), матрицам зависимостей уровней серого (grayleveldependencematrix, GLDM). Проанализированные работы также содержали информацию об объеме выборки, степени злокачественности глиом по классификации ВОЗ, программном обеспечении для расчета лучевых биомаркеров, напряженности поля магнитно-резонансного томографа, используемых последовательностях МР-сканирования, размере анализируемых изображений (2D/3D), способах сегментации, размерах обучающих и тестовых выборок, примененных моделях машинного обучения, а также о моделях машинного обучения с наиболее высокими показателями метрик качества на тестовых выборках. Оценку качества работы моделей машинного обучения по данным радиомики в изученных нами исследованиях проводили с использованием метрик точности, чувствительности, специфичности, F-меры, положительной и отрицательной прогностической ценности, а также площади под ROC-кривой.
Объем выборки, представленный в публикациях, варьировал от 40 до 1508 МР-исследований (155 [102; 258]). В 25 исследованиях (59,5%) МР-сканирование проводили в последовательностях T1, T1 с контрастным усилением, T2 и T2-FLAIR, а также использовали дополнительные режимы в отдельных сериях. В 16 сериях (38,1%) изображения с контрастным усилением не анализировали. В 37 (88%) работах расчеты лучевых биомаркеров проводили по «объемным» (3D) зонам интереса, из них в 3 работах дополнительно изучали «плоские» (2D) зоны. При этом в 5 работах информация о размерности зоны интереса отсутствует. Самым часто используемым программным обеспечением для извлечения признаков оказался продукт ITK-SNAP (упомянут в 14 работах). Кроме того, применяют технологию сегментации на основе алгоритма максимизации ожидания (expectation maximization, EM, algorithm), на основе которого реализован метод glioma image segmentation and registration — GLISTR (указанный в 5 публикациях), а также 3D-Slicer (приведен в 4 статьях). В 8 работах сведения о программах для сегментации зон интереса отсутствуют. В 20 исследованиях (47,6%) расчет лучевых биомаркеров был выполнен с помощью библиотеки PyRadiomics, в 5 работах использован MatLab, в 10 публикациях сведения о программах для расчета лучевых биомаркеров отсутствуют.
На рис. 2 [7–42] представлена точность моделей машинного обучения и ее обобщение по 36 работам из 42, для которых удалось оценить метрики точности. Обобщенная точность в проанализированных работах составила 0,86 с 95% ДИ [0,83; 0,89].

|
Рис. 2. Точность методов классификации МР-изображений глиом на основе радиомики и машинного обучения (n=36) |
Точность моделей классификации для предсказания отдельных молекулярных маркеров и обобщение этой точности по сериям работ демонстрирует рис. 3 [7–14, 17–21, 25, 27–29, 31, 33, 35–37, 39–42]. Наибольшую обобщенную точность имели модели предсказания статуса мутации гена IDH — на уровне 0,87 с 95% ДИ [0,84; 0,90] и ATRX — на уровне 0,85 c 95% ДИ [0,65; 0,95]. При этом модели предсказания ТЕRT и H3K27M показывали более низкие значения точности и более широкие доверительные интервалы (0,79 с 95% ДИ [0,46; 0,94] и 0,80 с 95% ДИ [0,07; 1,00] соответственно).

|
Рис. 3. Точность методов идентификации молекулярных маркеров IDH (n=18), ATRX (n=4), TERT (n=2), H3K27M (n=2) по МР-изображениям глиом |
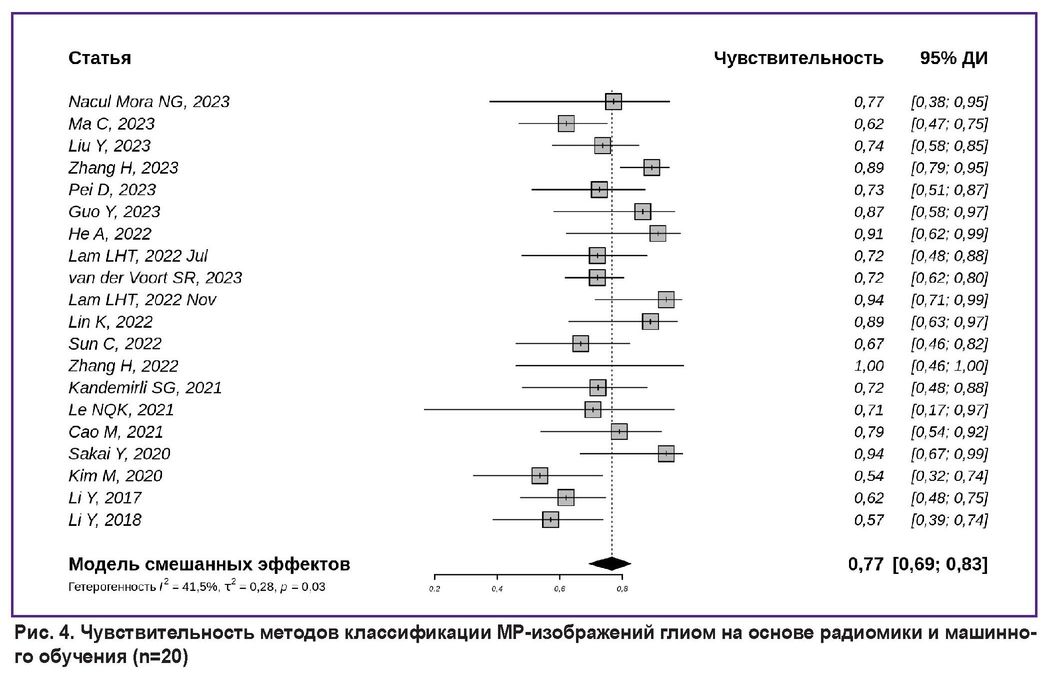
На рис. 4 [7, 8, 10–14, 16, 18, 19, 21, 22, 25, 26, 29, 38, 39, 43–45] представлена чувствительность методов неинвазивного типирования глиом на основе радиомики и машинного обучения для 20 изученных работ. Обобщенная чувствительность в изученной серии публикаций составила 0,77 с 95% ДИ [0,69; 0,83].
|
Рис. 4. Чувствительность методов классификации МР-изображений глиом на основе радиомики и машинного обучения (n=20) |
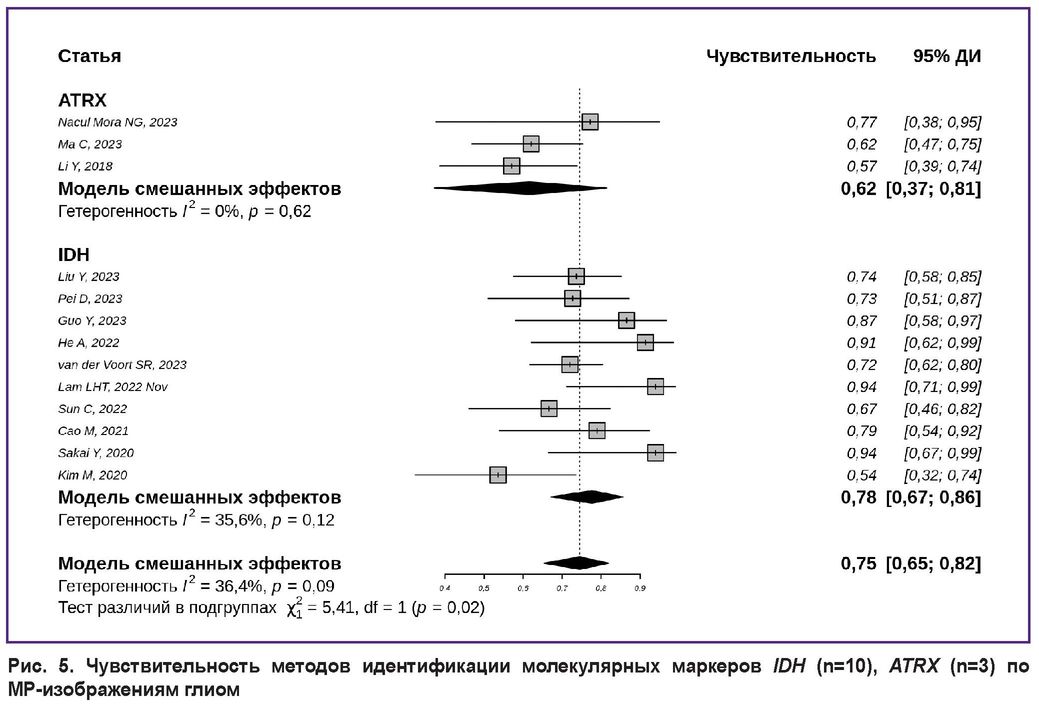
Чувствительность методов детекции статуса мутации генов IDH и ATRX показана на рис. 5 [7, 8, 10, 12–14, 18, 19, 21, 29, 39, 44, 45]. Обобщенная чувствительность моделей для обнаружения мутации гена IDH составила 0,78 с 95% ДИ [0,67; 0,86], тогда как для ATRX эта метрика была ниже — 0,62 с более широким 95% ДИ [0,37; 0,81].
Специфичность диагностики на основе радиомики и машинного обучения демонстрируют рис. 6 и 7. Обобщенная специфичность изученных методов превысила чувствительность и составила 0,85 с 95% ДИ [0,77; 0,90] (рис. 6) [7, 8, 10–14, 16, 18, 19, 21, 22, 25, 26, 29, 38, 39, 43, 44, 45].
|
Рис. 5. Чувствительность методов идентификации молекулярных маркеров IDH (n=10), ATRX (n=3) по МР-изображениям глиом |

|
Рис. 6. Специфичность методов классификации МР-изображений глиом на основе радиомики и машинного обучения (n=20) |
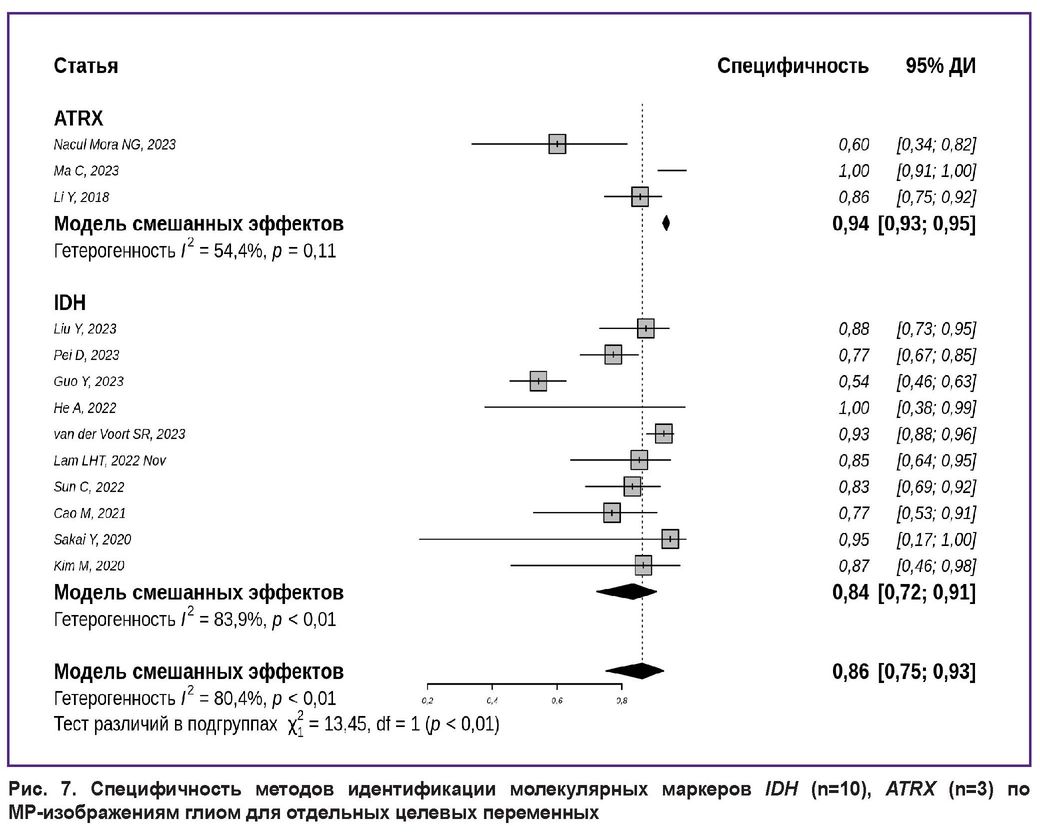
Максимальная специфичность отмечена для детекции мутаций ATRX (0,94 с 95% ДИ [0,93; 0,95]), для определения мутации в гене IDH она была несколько ниже (0,84 с 95% ДИ [0,72; 0,91]) (рис. 7) [7, 8, 10, 12–14, 18, 19, 21, 29, 39, 44, 45].
|
Рис. 7. Специфичность методов идентификации молекулярных маркеров IDH (n=10), ATRX (n=3) по МР-изображениям глиом для отдельных целевых переменных |
Обсуждение
Проведенный анализ литературы выявил довольно высокие показатели метрик качества моделей машинного обучения в решении задач неинвазивного типирования глиальных опухолей. При этом проанализированные работы демонстрируют значительное методологическое разнообразие в выборе целевых переменных для прогнозирования используемых МР-последовательностей, программных инструментов сегментации, методов вычисления лучевых биомаркеров и в других технических аспектах.
Парадоксально, но важную методологическую гетерогенность в процесс вычисления лучевых биомаркеров, с нашей точки зрения, вносит повторяющийся во всех работах принцип выделения зон интереса с помощью оконтуривания видимого сигнала опухоли. Возникающая при этом гетерогенность результатов объясняется двумя причинами: 1) несоответствием видимых границ глиом (например, зоны накопления контрастного вещества) истинной распространенности опухолевых клеток в веществе головного мозга; 2) произвольными, вариабельными размерами и формами зон интереса, в пределах которых выполняется расчет лучевых биомаркеров. Поскольку абсолютные значения лучевых биомаркеров могут зависеть от размера зоны интереса, в пределах которой они получены, последний фактор ставит под сомнение надежность такого рода измерений [49].
Одним из серьезных, на наш взгляд, ограничений современных работ в области радиомики глиальных опухолей является отсутствие оценки воспроизводимости (устойчивости) полученных биомаркеров и моделей машинного обучения.
Гетерогенность в изученных работах была свойственна не только исследовательскому процессу, но также качеству и полноте изложения результатов. В частности, в ряде работ отсутствовали указания на принципиальные аспекты технологии анализа изображения, такие как выбор зоны интереса и ее вида, применяемого программного обеспечения; напряженность поля МР-томографа и т.д. Международное сообщество, занимающееся стандартизацией процессов получения лучевых биомаркеров для количественного анализа (Image Biomarker Standardisation Initiative, IBSI), предложило рекомендации, следование которым приведет к минимизации методологической гетерогенности и повышению воспроизводимости результатов [49]. Отдельные публикации в проанализированной серии ссылались на эти рекомендации [17, 24].
Еще одно ограничение работ по радиомике глиальных опухолей — относительно небольшой объем выборки (редко превышающий 300 исследований). При существенной вариабельности визуализации этой опухолевой патологии и разнообразии ее генетических вариантов такой объем выборки представляется недостаточным для того, чтобы, с одной стороны, полноценно уловить самые разные цифровые паттерны, а с другой — проверить воспроизводимость лучевых биомаркеров. Вероятно, развитие инструментов биобанкирования с интеграцией самых разных клинических, лабораторных и инструментальных данных, а также накопление значительного банка данных (в том числе, в рамках мультицентровых исследований) позволит в будущем преодолеть этот недостаток.
Чуть более половины всех изученных нами работ были посвящены предсказанию статуса мутации гена IDH (см. рис. 3, 5, 7). В отдельных работах последних лет предложены подходы к автоматизированной сегментации глиом (на основе глубокого машинного обучения) и извлечению лучевых биомаркеров [17, 18, 46]. При этом нейросетевые модели используют для извлечения особых цифровых признаков изображений так называемые глубинные биомаркеры (deepfeatures), получаемые не по заранее известным формулам, а как промежуточный продукт в процессе машинного обучения нейронной сети. В отдельных исследованиях показано, что комбинация «классических» лучевых биомаркеров с «глубинными» позволяет улучшить точность предсказаний генетических вариантов опухоли [20].
Современная классификация опухолей нервной системы опирается на результаты молекулярных и генетических исследований, без которых сегодня невозможно назначить максимально эффективное, таргетное лечение [1]. В эпоху развития генетических технологий представления о природе опухолевых заболеваний расширяются. Однако доступ к дорогостоящим и трудоемким генетическим исследованиям в клинике ограничен. Именно поэтому поиск неинвазивных, в том числе лучевых, коррелятов генетических и молекулярных биомаркеров опухолей нервной системы видится перспективным научным направлением. В то же время, несмотря на успехи исследований с использованием технологий радиомики, предстоит большая работа по оценке их надежности, вопроизводимости и пригодности для использования в реальной клинической практике.
Заключение
Исследования в области радиомики и методов машинного обучения демонстрируют многообещающие результаты в неинвазивной диагностике глиальных опухолей. В то же время актуальными остаются вопросы надежности и воспроизводимости предлагаемых решений. В работах с использованием технологий радиомики для анализа МР-изображений глиальных опухолей и при публикации полученных результатов целесообразно придерживаться рекомендаций IBSI.
Вклад авторов. Концепция и дизайн исследования — Г.В. Данилов, С.Б. Агрба, первичный скрининг данных — С.Б. Агрба, контрольный скрининг данных — Г.В. Данилов, статистический анализ данных — Ю.В. Струнина, написание текста — Г.В. Данилов, С.Б. Агрба, Ю.В. Струнина, подготовка иллюстраций — Ю.В. Струнина, научное редактирование — И.Н. Пронин, А.М. Шевченко, Т.А. Конакова, С.В. Шугай, А.И. Баталов.
Финансирование. Работа поддержана грантом Министерства науки и высшего образования Российской Федерации 075-15-2024-561.
Конфликт интересов отсутствует.
Литература
- Ostrom Q.T., Cioffi G., Waite K., Kruchko C., Barnholtz-Sloan J.S. CBTRUS statistical report: primary brain and other central nervous system tumors diagnosed in the United States in 2014–2018. Neuro Oncol 2021; 23(12 Suppl 2): iii1–iii105, https://doi.org/10.1093/neuonc/noab200.
- Ostrom Q.T., Price M., Neff C., Cioffi G., Waite K.A., Kruchko C., Barnholtz-Sloan J.S. CBTRUS statistical report: primary brain and other central nervous system tumors diagnosed in the United States in 2015–2019. Neuro Oncol 2022; 24(Suppl 5): v1–v95, https://doi.org/10.1093/neuonc/noac202.
- Litvin A.A., Burkin D.A., Kropinov A.A., Paramzin F.N. Radiomics and digital image texture analysis in oncology (review). Sovremennye tehnologii v medicine 2021; 13(2): 97–104, https://doi.org/10.17691/stm2021.13.2.11.
- Gore S., Chougule T., Jagtap J., Saini J., Ingalhalikar M. A review of radiomics and deep predictive modeling in glioma characterization. Acad Radiol 2021; 28(11): 1599–1621, https://doi.org/10.1016/j.acra.2020.06.016.
- Jian A., Jang K., Manuguerra M., Liu S., Magnussen J., Di Ieva A. Machine learning for the prediction of molecular markers in glioma on magnetic resonance imaging: a systematic review and meta-analysis. Neurosurgery 2021; 89(1): 31–44, https://doi.org/10.1093/neuros/nyab103.
- Page M.J., McKenzie J.E., Bossuyt P.M., Boutron I., Hoffmann T.C., Mulrow C.D., Shamseer L., Tetzlaff J.M., Akl E.A., Brennan S.E., Chou R., Glanville J., Grimshaw J.M., Hróbjartsson A., Lalu M.M., Li T., Loder E.W., Mayo-Wilson E., McDonald S., McGuinness L.A., Stewart L.A., Thomas J., Tricco A.C., Welch V.A., Whiting P., Moher D. The PRISMA 2020 statement: an updated guideline for reporting systematic reviews. PLoS Med 2021; 18(3): e1003583, https://doi.org/10.1371/journal.pmed.1003583.
- Nacul Mora N.G., Akkurt B.H., Kasap D., Blömer D., Heindel W., Mannil M., Musigmann M. Comparison of MRI sequences to predict ATRX status using radiomics-based machine learning. Diagnostics (Basel) 2023; 13(13): 2216, https://doi.org/10.3390/diagnostics13132216.
- Ma C., Wang L., Song D., Gao C., Jing L., Lu Y., Liu D., Man W., Yang K., Meng Z., Zhang H., Xue P., Zhang Y., Guo F., Wang G. Multimodal-based machine learning strategy for accurate and non-invasive prediction of intramedullary glioma grade and mutation status of molecular markers: a retrospective study. BMC Med 2023; 21(1): 198, https://doi.org/10.1186/s12916-023-02898-4.
- Alom Z., Tran Q.T., Bag A.K., Lucas J.T., Orr B.A. Predicting methylation class from diffusely infiltrating adult gliomas using multimodality MRI data. Neurooncol Adv 2023; 5(1): vdad045, https://doi.org/10.1093/noajnl/vdad045.
- Liu Y., Zheng Z., Wang Z., Qian X., Yao Z., Cheng C., Zhou Z., Gao F., Dai Y. Using radiomics based on multicenter magnetic resonance images to predict isocitrate dehydrogenase mutation status of gliomas. Quant Imaging Med Surg 2023; 13(4): 2143–2155, https://doi.org/10.21037/qims-22-836.
- Zhang H., Zhang H., Zhang Y., Zhou B., Wu L., Lei Y., Huang B. Deep learning radiomics for the assessment of telomerase reverse transcriptase promoter mutation status in patients with glioblastoma using multiparametric MRI. J Magn Reson Imaging 2023; 58(5): 1441–1451, https://doi.org/10.1002/jmri.28671.
- Pei D., Guan F., Hong X., Liu Z., Wang W., Qiu Y., Duan W., Wang M., Sun C., Wang W., Wang X., Guo Y., Wang Z., Liu Z., Xing A., Guo Z., Luo L., Liu X., Cheng J., Zhang B., Zhang Z., Yan J. Radiomic features from dynamic susceptibility contrast perfusion-weighted imaging improve the three-class prediction of molecular subtypes in patients with adult diffuse gliomas. Eur Radiol 2023; 33(5): 3455–3466, https://doi.org/10.1007/s00330-023-09459-6.
- Guo Y., Ma Z., Pei D., Duan W., Guo Y., Liu Z., Guan F., Wang Z., Xing A., Guo Z., Luo L., Wang W., Yu B., Zhou J., Ji Y., Yan D., Cheng J., Liu X., Yan J., Zhang Z. Improving noninvasive classification of molecular subtypes of adult gliomas with diffusion-weighted MR imaging: an externally validated machine learning algorithm. J Magn Reson Imaging 2023; 58(4): 1234–1242, https://doi.org/10.1002/jmri.28630.
- He A., Wang P., Zhu A., Liu Y., Chen J., Liu L. Predicting IDH mutation status in low-grade gliomas based on optimal radiomic features combined with multi-sequence magnetic resonance imaging. Diagnostics (Basel) 2022; 12(12): 2995, https://doi.org/10.3390/diagnostics12122995.
- Do D.T., Yang M.R., Lam L.H.T., Le N.Q.K., Wu Y.W. Improving MGMT methylation status prediction of glioblastoma through optimizing radiomics features using genetic algorithm-based machine learning approach. Sci Rep 2022; 12(1): 13412, https://doi.org/10.1038/s41598-022-17707-w.
- Lam L.H.T., Chu N.T., Tran T.O., Do D.T., Le N.Q.K. A radiomics-based machine learning model for prediction of tumor mutational burden in lower-grade gliomas. Cancers (Basel) 2022 Jul; 14(14): 3492, https://doi.org/10.3390/cancers14143492.
- Zeng H., Xing Z., Gao F., Wu Z., Huang W., Su Y., Chen Z., Cai S., Cao D., Cai C. A multimodal domain adaptive segmentation framework for IDH genotype prediction. Int J Comput Assist Radiol Surg 2022; 17(10): 1923–1931, https://doi.org/10.1007/s11548-022-02700-5.
- van der Voort S.R., Incekara F., Wijnenga M.M.J., Kapsas G., Gahrmann R., Schouten J.W., Nandoe Tewarie R., Lycklama G.J., De Witt Hamer P.C., Eijgelaar R.S., French P.J., Dubbink H.J., Vincent A.J.P.E., Niessen W.J., van den Bent M.J., Smits M., Klein S. Combined molecular subtyping, grading, and segmentation of glioma using multi-task deep learning. Neuro Oncol 2023; 25(2): 279–289, https://doi.org/10.1093/neuonc/noac166.
- Lam L.H.T., Do D.T., Diep D.T.N., Nguyet D.L.N., Truong Q.D., Tri T.T., Thanh H.N., Le N.Q.K. Molecular subtype classification of low-grade gliomas using magnetic resonance imaging-based radiomics and machine learning. NMR Biomed 2022 Nov; 35(11): e4792, https://doi.org/10.1002/nbm.4792.
- Calabrese E., Rudie J.D., Rauschecker A.M., Villanueva-Meyer J.E., Clarke J.L., Solomon D.A., Cha S. Combining radiomics and deep convolutional neural network features from preoperative MRI for predicting clinically relevant genetic biomarkers in glioblastoma. Neurooncol Adv 2022; 4(1): vdac060, https://doi.org/10.1093/noajnl/vdac060.
- Sun C., Fan L., Wang W., Wang W., Liu L., Duan W., Pei D., Zhan Y., Zhao H., Sun T., Liu Z., Hong X., Wang X., Guo Y., Li W., Cheng J., Li Z., Liu X., Zhang Z., Yan J. Radiomics and qualitative features from multiparametric MRI predict molecular subtypes in patients with lower-grade glioma. Front Oncol 2022; 11: 756828, https://doi.org/10.3389/fonc.2021.756828.
- Zhang H., Zhang B., Pan W., Dong X., Li X., Chen J., Wang D., Ji W. Preoperative contrast-enhanced MRI in differentiating glioblastoma from low-grade gliomas in the cancer imaging archive database: a proof-of-concept study. Front Oncol 2022; 11: 761359, https://doi.org/10.3389/fonc.2021.761359.
- Kha Q.-H., Le V.-H., Hung T.N.K., Le N.Q.K. Development and validation of an efficient mri radiomics signature for improving the predictive performance of 1p/19q co-deletion in lower-grade gliomas. Cancers (Basel) 2021; 13(21): 5398, https://doi.org/10.3390/cancers13215398.
- Chen H., Lin F., Zhang J., Lv X., Zhou J., Li Z.C., Chen Y. Deep learning radiomics to predict PTEN mutation status from magnetic resonance imaging in patients with glioma. Front Oncol 2021; 11: 734433, https://doi.org/10.3389/fonc.2021.734433.
- Kandemirli S.G., Kocak B., Naganawa S., Ozturk K., Yip S.S.F., Chopra S., Rivetti L., Aldine A.S., Jones K., Cayci Z., Moritani T., Sato T.S. Machine learning-based multiparametric magnetic resonance imaging radiomics for prediction of H3K27M mutation in midline gliomas. World Neurosurg 2021; 151: e78–e85, https://doi.org/10.1016/j.wneu.2021.03.135.
- Le N.Q.K., Hung T.N.K., Do D.T., Lam L.H.T., Dang L.H., Huynh T.T. Radiomics-based machine learning model for efficiently classifying transcriptome subtypes in glioblastoma patients from MRI. Comput Biol Med 2021; 132: 104320, https://doi.org/10.1016/j.compbiomed.2021.104320.
- Fang S., Fan Z., Sun Z., Li Y., Liu X., Liang Y., Liu Y., Zhou C., Zhu Q., Zhang H., Li T., Li S., Jiang T., Wang Y., Wang L. Radiomics features predict telomerase reverse transcriptase promoter mutations in world health organization grade II gliomas via a machine-learning approach. Front Oncol 2021; 10: 606741, https://doi.org/10.3389/fonc.2020.606741.
- Zhang S., Sun H., Su X., Yang X., Wang W., Wan X., Tan Q., Chen N., Yue Q., Gong Q. Automated machine learning to predict the co-occurrence of isocitrate dehydrogenase mutations and O6-methylguanine-DNA methyltransferase promoter methylation in patients with gliomas. J Magn Reson Imaging 2021; 54(1): 197–205, https://doi.org/10.1002/jmri.27498.
- Sakai Y., Yang C., Kihira S., Tsankova N., Khan F., Hormigo A., Lai A., Cloughesy T., Nael K. MRI radiomic features to predict IDH1 mutation status in gliomas: a machine learning approach using gradient tree boosting. Int J Mol Sci 2020; 21(21): 8004, https://doi.org/10.3390/ijms21218004.
- Gao M., Huang S., Pan X., Liao X., Yang R., Liu J. Machine learning-based radiomics predicting tumor grades and expression of multiple pathologic biomarkers in gliomas. Front Oncol 2020; 10: 1676, https://doi.org/10.3389/fonc.2020.01676.
- Choi Y.S., Bae S., Chang J.H., Kang S.G., Kim S.H., Kim J., Rim T.H., Choi S.H., Jain R., Lee S.K. Fully automated hybrid approach to predict the IDH mutation status of gliomas via deep learning and radiomics. Neuro Oncol 2021; 23(2): 304–313, https://doi.org/10.1093/neuonc/noaa177.
- Kocak B., Durmaz E.S., Ates E., Sel I., Turgut Gunes S., Kaya O.K., Zeynalova A., Kilickesmez O. Radiogenomics of lower-grade gliomas: machine learning-based MRI texture analysis for predicting 1p/19q codeletion status. Eur Radiol 2020; 30(2): 877–886, https://doi.org/10.1007/s00330-019-06492-2.
- Su X., Chen N., Sun H., Liu Y., Yang X., Wang W., Zhang S., Tan Q., Su J., Gong Q., Yue Q. Automated machine learning based on radiomics features predicts H3 K27M mutation in midline gliomas of the brain. Neuro Oncol 2020; 22(3): 393–401, https://doi.org/10.1093/neuonc/noz184.
- Vamvakas A., Williams S.C., Theodorou K., Kapsalaki E., Fountas K., Kappas C., Vassiou K., Tsougos I. Imaging biomarker analysis of advanced multiparametric MRI for glioma grading. Phys Med 2019; 60: 188–198, https://doi.org/10.1016/j.ejmp.2019.03.014.
- Lee M.H., Kim J., Kim S.T., Shin H.M., You H.J., Choi J.W., Seol H.J., Nam D.H., Lee J.I., Kong D.S. Prediction of IDH1 mutation status in glioblastoma using machine learning technique based on quantitative radiomic data. World Neurosurg 2019; 125: e688–e696, https://doi.org/10.1016/j.wneu.2019.01.157.
- Wu S., Meng J., Yu Q., Li P., Fu S. Radiomics-based machine learning methods for isocitrate dehydrogenase genotype prediction of diffuse gliomas. J Cancer Res Clin Oncol 2019; 145(3): 543–550, https://doi.org/10.1007/s00432-018-2787-1.
- Liang S., Zhang R., Liang D., Song T., Ai T., Xia C., Xia L., Wang Y. Multimodal 3D DenseNet for IDH genotype prediction in gliomas. Genes (Basel) 2018; 9(8): 382, https://doi.org/10.3390/genes9080382.
- Li Y., Qian Z., Xu K., Wang K., Fan X., Li S., Jiang T., Liu X., Wang Y. MRI features predict p53 status in lower-grade gliomas via a machine-learning approach. Neuroimage Clin 2017; 17: 306–311, https://doi.org/10.1016/j.nicl.2017.10.030.
- Li Y., Liu X., Qian Z., Sun Z., Xu K., Wang K., Fan X., Zhang Z., Li S., Wang Y., Jiang T. Genotype prediction of ATRX mutation in lower-grade gliomas using an MRI radiomics signature. Eur Radiol 2018; 28(7): 2960–2968, https://doi.org/10.1007/s00330-017-5267-0.
- Shofty B., Artzi M., Ben Bashat D., Liberman G., Haim O., Kashanian A., Bokstein F., Blumenthal D.T., Ram Z., Shahar T. MRI radiomics analysis of molecular alterations in low-grade gliomas. Int J Comput Assist Radiol Surg 2018; 13(4): 563–571, https://doi.org/10.1007/s11548-017-1691-5.
- Eichinger P., Alberts E., Delbridge C., Trebeschi S., Valentinitsch A., Bette S., Huber T., Gempt J., Meyer B., Schlegel J., Zimmer C., Kirschke J.S., Menze B.H., Wiestler B. Diffusion tensor image features predict IDH genotype in newly diagnosed WHO grade II/III gliomas. Sci Rep 2017; 7(1): 13396, https://doi.org/10.1038/s41598-017-13679-4.
- Li Z., Wang Y., Yu J., Guo Y., Cao W. Deep Learning based Radiomics (DLR) and its usage in noninvasive IDH1 prediction for low grade glioma. Sci Rep 2017; 7(1): 5467, https://doi.org/10.1038/s41598-017-05848-2.
- Lin K., Cidan W., Qi Y., Wang X. Glioma grading prediction using multiparametric magnetic resonance imaging-based radiomics combined with proton magnetic resonance spectroscopy and diffusion tensor imaging. Med Phys 2022; 49(7): 4419–4429, https://doi.org/10.1002/mp.15648.
- Cao M., Suo S., Zhang X., Wang X., Xu J., Yang W., Zhou Y. Qualitative and quantitative MRI analysis in idh1 genotype prediction of lower-grade gliomas: a machine learning approach. Biomed Res Int 2021; 2021: 1235314, https://doi.org/10.1155/2021/1235314.
- Kim M., Jung S.Y., Park J.E., Jo Y., Park S.Y., Nam S.J., Kim J.H., Kim H.S. Diffusion- and perfusion-weighted MRI radiomics model may predict isocitrate dehydrogenase (IDH) mutation and tumor aggressiveness in diffuse lower grade glioma. Eur Radiol 2020; 30(4): 2142–2151, https://doi.org/10.1007/s00330-019-06548-3.
- Vafaeikia P., Wagner M.W., Hawkins C., Tabori U., Ertl-Wagner B.B., Khalvati F. MRI-based end-to-end pediatric low-grade glioma segmentation and classification. Can Assoc Radiol J 2024; 75(1): 153–160, https://doi.org/10.1177/08465371231184780.
- Park C.J., Han K., Kim H., Ahn S.S., Choi D., Park Y.W., Chang J.H., Kim S.H., Cha S., Lee S.K. MRI features may predict molecular features of glioblastoma in isocitrate dehydrogenase wild-type lower-grade gliomas. AJNR Am J Neuroradiol 2021; 42(3): 448–456, https://doi.org/10.3174/ajnr.A6983.
- Park C.J., Choi Y.S., Park Y.W., Ahn S.S., Kang S.G., Chang J.H., Kim S.H., Lee S.K. Diffusion tensor imaging radiomics in lower-grade glioma: improving subtyping of isocitrate dehydrogenase mutation status. Neuroradiology 2020; 62(3): 319–326, https://doi.org/10.1007/s00234-019-02312-y.
- IBSI Documentation Release 0.0.1dev ibsi community. 2021. URL: https://app.readthedocs.org/projects/ibsi/downloads/pdf/latest/.










