Молекулярные маркеры BЭБ- и ВГЧ6-ассоциированного мононуклеоза
Вирус Эпштейна–Барр (ВЭБ) и вирус герпеса человека тип 6 (ВГЧ6) являются возбудителями инфекционного мононуклеоза и могут вносить существенный вклад в развитие лимфопролиферативных заболеваний. Эффективные средства терапии инфекции на сегодняшний день не разработаны. В качестве потенциальной терапевтической мишени возможно использование молекулярных маркеров — ключевых транскриптов — участников патогенеза.
Цель исследования — выявление набора молекулярно-генетических маркеров ВЭБ- и ВГЧ6-ассоциированной инфекции в лейкоцитах крови пациентов с инфекционным мононуклеозом.
Материалы и методы. Исследован транскриптом лейкоцитов периферической крови детей и подростков с инфекционным мононуклеозом, вызванным вирусом Эпштейна–Барр (ВЭБ-ИМ) и вирусом герпеса человека тип 6 (ВГЧ6-ИМ), а также здоровых доноров, сопоставимых по полу и возрасту. С помощью разработанных ДНК-биочипов проанализирована экспрессия 403 генов (суммарный уровень представленности всех мРНК одного гена) и 712 транскриптов (индивидуальных сплайсированных вариантов мРНК одного гена), участвующих в регуляции активации, пролиферации и апоптоза иммунокомпетентных клеток. Анализ данных проводили с помощью комбинации методов машинного обучения и традиционной статистики. В качестве молекулярных маркеров инфекции отобраны гены и транскрипты, характеризующиеся максимальной важностью для парной классификации и статистически значимыми изменениями по сравнению с группой здоровых доноров.
Результаты. Выделены уникальные наборы кандидатных маркеров ВЭБ-ИМ и ВГЧ6-ИМ. ВЭБ-ИМ характеризовался снижением экспрессии транскриптов 5 AR и 4 ASCC1, гена CAD и мРНК FADD на фоне повышения экспрессии транскриптов 2 HLA-DPA1 и 4 RIPK1. ВГЧ6-ИМ сопровождался повышением экспрессии мРНК AVEN, транскриптов 2 CHUK, 2 CIRBP и 2 TRAF3, а также снижением экспрессии транскрипта 10 IRAK4. После выздоровления экспрессия большинства маркеров нормализовалась.
Заключение. Наборы выявленных маркеров уникальны в отношении двух изученных инфекций и могут использоваться в качестве мишеней при разработке новых специфических биотерапевтических препаратов.
Введение
Вирус Эпштейна–Барр (ВЭБ, Human gammaherpesvirus 4) и вирус герпеса человека тип 6 (ВГЧ6, Human betaherpesvirus 6) — представители семейства Herpesviridae, являющиеся возбудителями инфекционного мононуклеоза (ИМ). Оба вируса обладают тропностью к широкому спектру субпопуляций иммунокомпетентных клеток, оказывают влияние на их содержание в крови пациентов [1], а также могут вносить существенный вклад в развитие лимфопролиферативных заболеваний [2–4]. На сегодняшний день эффективные таргетные методы терапии не разработаны.
На молекулярном уровне воздействие вирусов реализуется путем модуляции экспрессии генов, участвующих в активации клеток иммунной системы, их пролиферации, дифференцировке и апоптозе. Выраженное изменение экспрессии набора генов, контролирующих указанные процессы, может служить своеобразным уникальным молекулярным паспортом ВЭБ-ИМ и ВГЧ6-ИМ, а отдельные гены — маркерами инфекции. Такие маркеры можно использовать с целью диагностики и мониторинга заболеваний, а также в качестве потенциальной терапевтической мишени [5–8].
С применением технологии ДНК-биочипов (DNA microarray) можно оценить не только экспрессию гена (суммарный уровень представленности всех его мРНК), но и состав, а также количественное соотношение его транскриптов (сплайсированных вариантов мРНК одного гена). Учитывая оптимальное соотношение стоимости исследования, производительности и точности результатов, ДНК-биочипы часто становятся технологией первого выбора при проведении поисковых работ, целью которых является обнаружение новых молекулярно-генетических маркеров широкого спектра заболеваний [9]. Анализ результатов, полученных с помощью ДНК-биочипов, так же как и с помощью методов высокопроизводительного секвенирования, требует привлечения сложного математического аппарата, сочетающего методы прикладной статистики и алгоритмы машинного обучения [10].
Целью данной работы стало выявление набора молекулярно-генетических маркеров ВЭБ- и ВГЧ6-ассоциированной инфекции в лейкоцитах крови пациентов с инфекционным мононуклеозом.
Материалы и методы
Дизайн и синтез ДНК-биочипа. Для анализа экспрессии мРНК генов — участников основных клеточных процессов — нами сконструированы ДНК-биочипы. Селекцию дискриминирующих зондов, являющихся функциональной основой биочипа, производили с использованием алгоритма «Splice variants microarray design pipeline» [11]. Всего выбрано 1115 зондов, из которых 403 позволяют оценивать экспрессию генов, а 712 детектируют уровни представленности индивидуальных транскриптов. Полный перечень генов и их транскриптов представлен в дополнительных материалах (см. Приложение 1, https://drive.google.com/file/d/1xKS_MvENloPYKwuzeC1LaqFTH3i130So/view). Также в дизайн ДНК-биочипа включены 70 зондов негативного контроля, подобранных на основе генома бактерии Rhizobium rubi. Синтез зондов на биочип осуществляли in situ с помощью оборудования B3 Synthesizer и соответствующего комплекта реагентов в соответствии с рекомендациями производителя (CustomArray Inc., США).
Отбор исследуемых групп. В исследовании приняли участие дети и подростки 7–18 лет с диагнозом «острый инфекционный мононуклеоз», а также практически здоровые добровольцы, сопоставимые по полу и возрасту. В крови и сыворотке крови участников определяли наличие специфических антител к ВЭБ, ВГЧ6 и цитомегаловирусу (ЦМВ), а также наличие ДНК этих возбудителей. Использовали коммерческие наборы для иммуноферментной диагностики: «ВектоВЭБ-VCA-IgM», «ВектоВЭБ-VCA-IgG», «ВектоHHV-6-IgG», «ВектоЦМВ-IgM», «ВектоЦМВ-IgG» («Вектор-Бест», Россия) и коммерческий набор для ПЦР в режиме реального времени «АмплиСенс EBV/CMV/HHV6-cкрин-FL» (Центральный научно-исследовательский институт эпидемиологии, Россия).
По результатам обследования были выделены группы: НОРМ — практически здоровые доноры без клинических и лабораторных признаков инфекции (n=17, средний возраст — 11 лет); ВЭБинф — пациенты с ВЭБ-опосредованным ИМ (n=6, средний возраст — 12 лет); ВГЧ6инф — пациенты с ВГЧ6-опосредованным ИМ (n=7, средний возраст — 11 лет). Клинически здоровые добровольцы с лабораторными признаками инфекции, пациенты с ЦМВ-опосредованным ИМ или микст-инфекцией из исследования были исключены.
Забор материала. В качестве материала для исследования использовали образцы периферической крови. Забор крови у пациентов с инфекцией проводили до начала лечения, а также после выздоровления и исчезновения клинических и лабораторных признаков заболевания (группы ВЭБрек и ВГЧ6рек). Средний срок повторного забора крови составил 2 мес после реконвалесценции.
Исследование проведено в соответствии с Хельсинкской декларацией (2013) и одобрено Этическим комитетом Нижегородского НИИ эпидемиологии и микробиологии им. академика И.Н. Блохиной Роспотребнадзора. Забор материала выполняли с информированного согласия родителей или опекунов пациентов.
Подготовка и гибридизация мРНК. Для выделения фракции лейкоцитов использовали раствор «Гемолитик» (Центральный научно-исследовательский институт эпидемиологии, Россия). С помощью набора «МАГНО-сорб» (Центральный научно-исследовательский институт эпидемиологии, Россия) из полученных лейкоцитов выделяли пул тотальной РНК, который дополнительно очищали и концентрировали смесью фенола и хлороформа в соотношении 1:1. Тотальная РНК (2 мкг) подвергалась обратной транскрипции и достройке второй цепи с применением набора Mint cDNA synthesis kit («Евроген», Россия), при этом 3’-олиго-Т-праймер заменяли на содержащий T7 промотор-олиго-Т-праймер («ДНК-синтез», Россия). Достройку второй цепи и амплификацию кДНК проводили в ходе 15 циклов ПЦР по программе: 95°С — 25 с; 60°С — 25 с; 72°С — 6 мин. Полученную двухцепочечную кДНК (2 мкг) транскрибировали с применением Т7 РНК-полимеразы (Thermo Scientific, Англия), добавляя биотиновую метку за счет использования биотинилированных нуклеотидов UTP («ДНК-синтез», Россия). Процедуру транскрипции повторяли трижды.
Полученные в результате синтеза пробы меченых РНК гибридизировали раздельно. Процедуру гибридизации РНК на биочип и считывание амперометрического сигнала выполняли с применением оборудования и реагентов ElectraSense (CustomArray Inc., США) в соответствии с рекомендациями производителя. Полученный сигнал рассматривали как относительный уровень экспрессии гена или транскрипта.
Обработка данных. Обработку данных и расчеты выполняли с применением языка программирования R (версия 3.5.0; R Foundation for Statistical Computing, Австрия) и среды RStudio (версия 1.1.453; RStudio, США). Данные были нормализованы с использованием квантильной нормализации на основе негативного контроля [12]. Затем строили матрицу результатов, в которой строки соответствовали факторам — уровням экспрессии генов и их транскриптов, а колонки — пробам РНК, отнесенным к той или иной группе (НОРМ, ВЭБинф, ВГЧ6инф, ВЭБрек и ВГЧ6рек).
Далее проводили попарный дискриминантный анализ всех пяти групп. Строили модель зависимости принадлежности пробы РНК к одной из двух сравниваемых групп. Использовали алгоритм градиентного бустинга над решающими деревьями. Подбор параметров модели и оценку ее эффективности осуществляли с применением метода кросс-валидации. Полученный классификатор считали эффективным при значении медианы показателя аккуратности (accuracy) и медианы показателя площади (AUC) под кривой ошибок (area under receiver operating characteristic (ROC) curve) не менее 0,75. В случае построения эффективного классификатора для улучшения качества классификации проводили повторное моделирование, снижая количество факторов. Отбирали по 20 генов или транскриптов, обладающих максимальной важностью (feature importance) для каждой модели, полученной в результате кросс-валидации, исключая дублирование [10]. Далее отобранные показатели использовали для повторной процедуры моделирования, реализуемой аналогично первой.
На следующем этапе проводили сравнение уровня экспрессии каждого гена или транскрипта в группах НОРМ и ВЭБинф, НОРМ и ВГЧ6инф, ВЭБинф и ВГЧ6инф, НОРМ и ВЭБрек, НОРМ и ВГЧ6рек. Применяли Т-тест с поправкой на ожидаемую долю ложных отклонений (false discovery rate, FDR) с расчетом значений q. При q<0,05 различия в экспрессии генов и транскриптов считали статистически значимыми.
Также рассчитывали изменение уровня экспрессии. Для этого в каждой группе определяли среднее значение уровня экспрессии для каждого гена или транскрипта, после чего для каждой пары групп рассчитывали изменение экспрессии (%):
(среднее группы 2 · 100 / среднее группы 1) – 100.
На основании полученных данных о важности генов и транскриптов для повторной классификации, об изменении уровня их экспрессии и статистической значимости этого изменения делали выводы о возможности отнесения фактора к молекулярным маркерам ВЭБ-ИМ или ВГЧ6-ИМ. Среди факторов классификации пары НОРМ–ВЭБинф отбирали гены и транскрипты, удовлетворявшие следующим требованиям: уровень экспрессии маркера в группе ВЭБинф статистически значимо изменялся по сравнению с группой НОРМ; изменение уровня экспрессии маркера и/или его важность для классификации НОРМ–ВЭБинф превышала 75-й процентиль (пороговое значение установлено эмпирически) соответствующего показателя для всех факторов; маркер входил в состав факторов классификации ВГЧ6инф–ВЭБинф; маркер отсутствовал среди факторов классификации НОРМ–ВГЧ6инф. Отобранные гены или транскрипты считали маркерами ВЭБ-инфекции. Маркеры ВГЧ6-инфекции из факторов классификации НОРМ–ВГЧ6инф отбирали аналогично.
Результаты
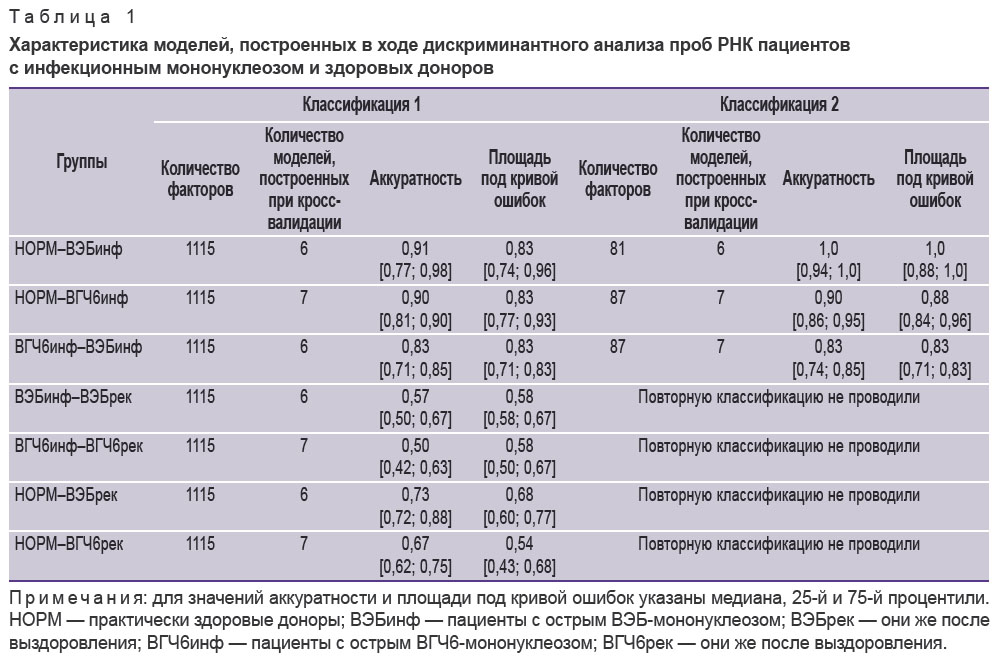
На начальном этапе были построены модели классификации проб мРНК в группах сравнения НОРМ–ВЭБинф, НОРМ–ВГЧ6инф и ВГЧ6инф–ВЭБинф. При этом сокращение количества факторов позволило улучшить качество моделей при повторной классификации групп НОРМ–ВЭБинф и НОРМ–ВГЧ6инф. Для групп сравнения ВГЧ6инф–ВЭБинф снижение количества факторов и повторное моделирование улучшило качество классификации незначительно (табл. 1). В среднем количество факторов, используемых для второй классификации, составило менее 8% от исходного набора. Полный перечень генов и транскриптов, уровень которых учитывали при построении второй модели принадлежности проб РНК к группам НОРМ–ВЭБинф, НОРМ–ВГЧ6инф и ВГЧ6инф–ВЭБинф, представлен в дополнительных материалах (см. Приложения 2–4, https://drive.google.com/file/d/1xKS_MvENloPYKwuzeC1LaqFTH3i130So/view). Для групп сравнения ВЭБинф–ВЭБрек, ВГЧ6инф–ВГЧ6рек, НОРМ–ВЭБрек и НОРМ–ВГЧ6рек не удалось построить удовлетворительных моделей принадлежности проб РНК к группам (см. табл. 1).
|
Таблица 1. Характеристика моделей, построенных в ходе дискриминантного анализа проб РНК пациентов с инфекционным мононуклеозом и здоровых доноров |
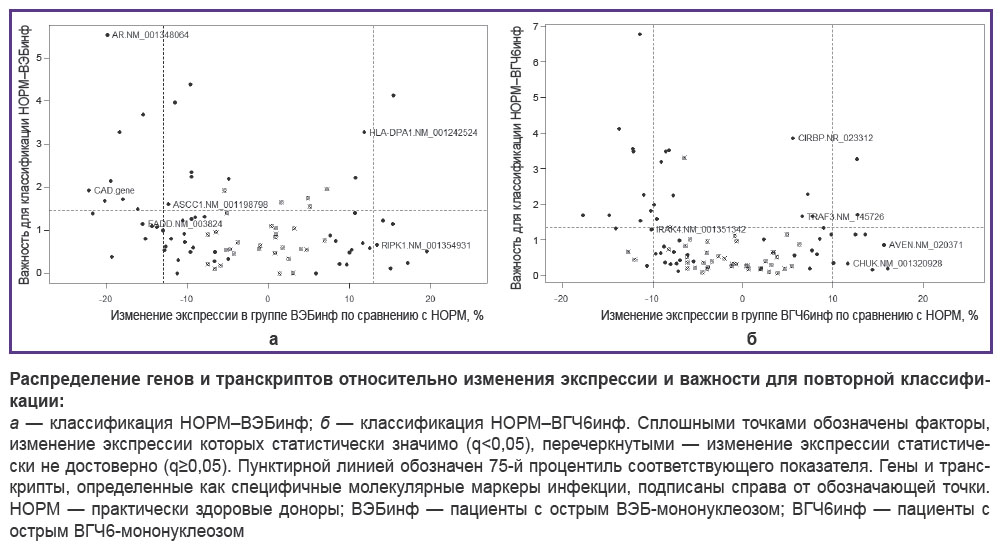
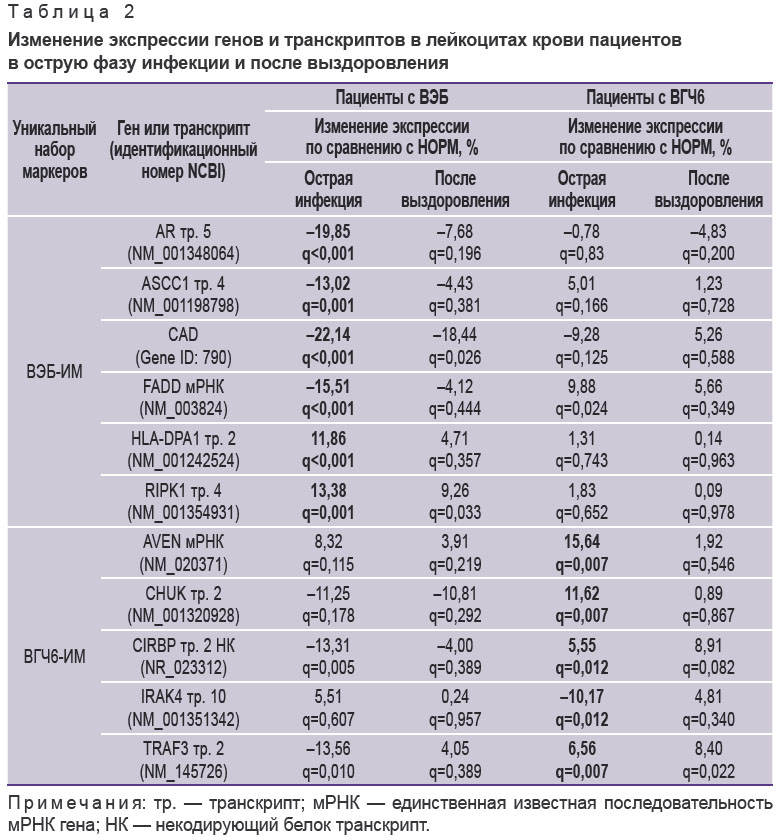
Далее согласно алгоритму были отобраны уникальные наборы молекулярных маркеров, позволяющих различить ВЭБ-ИМ и ВГЧ6-ИМ (см. рисунок). Для каждого выявленного маркера проанализированы изменения уровня его экспрессии по сравнению с нормой в острую фазу заболевания и в период реконвалесценции (табл. 2). У пациентов с острым ВЭБ-ИМ наблюдалось снижение экспрессии транскриптов 5 AR и 4 ASCC1, мРНК FADD, а также гена CAD по сравнению с нормой. Экспрессия транскриптов 2 HLA-DPA1 и 4 RIPK1, наоборот, повышалась. Реконвалесценция сопровождалась нормализацией всех показателей, за исключением уровней экспрессии гена CAD и транскрипта 4 RIPK1, которые сохранялись измененными. У пациентов с ВГЧ6-ИМ вне зависимости от фазы заболевания экспрессия маркеров ВЭБ-ИМ соответствовала таковой у здоровых доноров. Исключение составила мРНК FADD, экспрессия которой у пациентов с ВГЧ6-ИМ возрастала.
|
Таблица 2. Изменение экспрессии генов и транскриптов в лейкоцитах крови пациентов в острую фазу инфекции и после выздоровления |
В острый период ВГЧ6-ИМ обнаружено повышение экспрессии мРНК AVEN, транскриптов 2 CHUK, 2 CIRBP и 2 TRAF3, а также снижение экспрессии транскрипта 10 IRAK4. После выздоровления наблюдали нормализацию всех параметров за исключением уровня экспрессии транскрипта 2 TRAF3, который оставался повышенным. У пациентов с ВЭБ-ИМ экспрессия вышеперечисленных маркеров не изменялась, за исключением транскриптов 2 CIRBP и 10 IRAK4, уровни представленности которых соответственно снижались и повышались в острую фазу инфекции.
Обсуждение
Предложенный нами алгоритм обработки данных гибридизации проб РНК на ДНК-биочип позволяет выявить уникальный набор молекулярных маркеров ВЭБ-ИМ и ВГЧ6-ИМ. Он основан на комбинации методов машинного обучения и традиционной статистики. Такой комбинированный подход позволил решить несколько задач: определить принципиальную возможность отнесения пациента к той или иной группе на основании экспрессии набора генов и индивидуальных транскриптов (задача классификации); сократить количество анализируемых факторов за счет удаления незначимых для анализа элементов (выделение важных для классификации параметров); определить статистически значимые изменения экспрессии каждого из множества генов у пациентов с ВЭБ-ИМ и ВГЧ6-ИМ (задача статистики); выделить уникальный для каждого заболевания набор маркерных генов и транскриптов (логическое исключение).
В рамках настоящего исследования построены классификаторы, позволяющие дифференцировать практически здоровых лиц, пациентов с ВЭБ-ИМ и с ВГЧ6-ИМ на основании экспрессии изученных генов и транскриптов. Данный факт свидетельствует о существовании различий в молекулярных механизмах патогенеза двух вирусных инфекций. С другой стороны, модели отличий паттернов экспрессии генов и транскриптов группы пациентов после выздоровления от здоровых доноров и детей с острым ИМ построить не удалось. Полученный результат позволяет предположить, что экспрессия набора изученных нами генов и транскриптов после клинического выздоровления не нормализуется, по крайней мере в течение двух месяцев, сохраняя «молекулярный след» инфекции. Данный вывод подтверждается фактом изменения относительно нормы экспрессии некоторых маркеров ВЭБ-ИМ и ВГЧ6-ИМ и после выздоровления (см. табл. 2). Таким образом, ВЭБ и ВГЧ6 способны оказывать пролонгированное влияние на иммунную систему пациента, регулируя экспрессию генов и транскриптов, участвующих в апоптозе, пролиферации и других фундаментальных процессах, которые реализуются в иммунокомпетентных клетках.
С использованием предложенного алгоритма нами определены уникальные наборы молекулярных маркеров ВЭБ- и ВГЧ6-ассоциированного мононуклеоза. Преобладающее большинство кандидатных маркеров являются сплайсированными вариантами мРНК генов, принадлежащих к разным функциональным группам. Часть транскриптов представляют собой единственный известный либо «основной» вариант мРНК одного гена (мРНК FADD, мРНК AVEN). Как правило, функция белков, кодируемых такими транскриптами, в литературе описывается как свойство самого гена. Другие транскрипты кодируют структурно-функциональные формы белка, отличные от «основного» варианта, но проявляющие схожие свойства (транскрипты 5 AR, 2 CHUK и 10 IRAK4). Третью группу формируют сплайсированные варианты мРНК гена, отличающиеся от «основной» формы нуклеотидными последовательностями в 5’-НТР (транскрипты 2 HLA-DPA1 и 4 RIPK1). Белковый продукт таких транскриптов аналогичен продукту «основного» сплайсированного варианта мРНК. При этом стабильность самих транскриптов и активность их трансляции могут отличаться вследствие различий нуклеотидного состава в регуляторных областях мРНК. Особый интерес представляют транскрипты, продукты трансляции которых имеют функцию, отличающуюся от таковой у «основного» варианта мРНК или прямо противоположную ему. Среди выявленных нами маркеров к этой группе относится транскрипт 4 ASCC1, кодирующий белок, который, в отличие от «основной» формы, не способен ингибировать экспрессию NF-kB и NF-kB-таргетных генов [13]. Другой пример — транскрипт 2 ингибитора NF-kB-киназ TRAF3, продукт которого индуцирует экспрессию NF-kB в активированных Т-клетках [14].
Помимо белок-кодирующих транскриптов в качестве маркера ВГЧ6-инфекции нами определен некодирующий транскрипт 2 регулятора клеточного цикла и индуктора апоптоза CIRBP. Функциональное значение не транслирующих белок транскриптов хорошо объяснено в рамках концепции непродуктивного сплайсинга — альтернативного сплайсинга, приводящего к образованию некодирующих транскриптов таргетного гена и их последующей деградации. Изменение соотношения кодирующих и некодирующих транскриптов определяет уровень экспрессии белкового продукта целевого гена [15]. Описанный механизм регуляции генной экспрессии за счет альтернативного сплайсинга может быть расширен и на кодирующие белок варианты мРНК: увеличение доли одного транскрипта приводит к уменьшению доли других транскриптов одного гена и наоборот.
Таким образом, использование транскриптов в качестве маркеров инфекционного заболевания не только оправдано, но и перспективно с точки зрения расширения диагностических и прогностических возможностей биомедицины. Некоторые транскрипты одного гена могут иметь различную клиническую информативность. Так, по нашим данным, снижение экспрессии транскрипта 4 ASCC1 специфично только для ВЭБ-ИМ, в то время как изменение экспрессии транскрипта 1 ASCC1 обнаружено как при ВЭБ-ИМ, так и при ВГЧ6-ИМ (см. Приложения 2, 3, https://drive.google.com/file/d/1xKS_MvENloPYKwuzeC1LaqFTH3i130So/view). При этом большинство используемых в настоящее время диагностических тест-систем определяют экспрессию гена или содержание белка, но не позволяют оценивать соотношение индивидуальных транскриптов.
Полученные нами результаты говорят об активации различных молекулярных механизмов регуляции пролиферации и апоптоза клеток в лейкоцитах крови пациентов с ВЭБ-ИМ и ВГЧ6-ИМ. Развитие ВЭБ-инфекции сопровождалось снижением экспрессии промоторов пролиферации транскрипта 5 рецептора стероидных гормонов AR, транскрипта 4 ASCC1 и гена CAD. С другой стороны, при ВЭБ-ИМ также наблюдалось снижение экспрессии мРНК адаптерной молекулы FADD, играющей ключевую роль в цитотоксическом иммунном ответе и апоптозе, и повышение экспрессии транскрипта 4 RIPK1. Киназа RIPK1 является полифункциональным белком, входящим в состав сигнальных путей некроптоза, воспаления и активации NF-kB. Интересно отметить, что реализация некроптоза требует формирования крупного сигнального комплекса, в состав которого в эквимолярных количествах входят RIPK1, FADD, каспаза-8 и другие белки [16]. Таким образом, выявленное усиление экспрессии транскрипта 4 RIPK1 при ВЭБ-инфекции на фоне снижения экспрессии FADD может ингибировать апоптоз клетки и способствовать ее выживанию и пролиферации. Кроме того, обнаружено повышение экспрессии транскрипта 2 HLA-DPA1, который не только играет важную роль в презентации антигена, но и служит корецептором, облегчая проникновение ВЭБ в В-лимфоциты [17].
Функциональная роль маркеров ВГЧ6-инфекции преимущественно сводится к активации транскрипции и подавлению апоптоза в клетках. Так, обнаружено повышение экспрессии активаторов NF-kB транскрипта 2 TRAF3 и транскрипта 2 CHUK на фоне роста уровня экспрессии мРНК ингибитора митохондриального пути апоптоза AVEN. Экспрессия некодирующего транскрипта 2 проапоптотического фактора CIRBP повышалась, что также свидетельствует о снижении апоптотической активности в клетках. Кроме того, отметим снижение экспрессии важного медиатора врожденного иммунного ответа транскрипта 10 IRAK4.
Выявленные уникальные наборы молекулярных маркеров ВЭБ- и ВГЧ6-инфекционного мононуклеоза могут быть использованы в качестве терапевтических мишеней при разработке средств современной биотерапии. Важно отметить, что предложенный нами алгоритм может применяться для поиска уникальных маркеров патогенеза других инфекционных заболеваний.
Заключение
В лейкоцитах крови детей и подростков с ВЭБ и ВГЧ6, ассоциированными с мононуклеозом, выявлено изменение экспрессии некоторых генов и транскриптов, регулирующих активацию, пролиферацию и апоптоз иммунокомпетентных клеток. По сравнению с нормой в лейкоцитах пациентов с ВЭБ-инфекцией выявлено снижение экспрессии транскриптов 5 AR и 4 ASCC1, гена CAD и мРНК FADD на фоне повышения экспрессии транскриптов 2 HLA-DPA1 и 4 RIPK1. У пациентов с ВГЧ6-ИМ обнаружено повышение экспрессии мРНК AVEN, транскриптов 2 CHUK, 2 CIRBP и 2 TRAF3, а также снижение экспрессии транскрипта 10 IRAK4. Выявленные маркеры являются важными участниками патогенеза ВЭБ-ИМ и ВГЧ6-ИМ и отражают специфические черты его молекулярных механизмов, а также иммунного ответа на инфекцию. Наборы выявленных маркеров уникальны для двух исследованных инфекций.
Финансирование исследования. Работа выполнена по государственному заданию №АААА-А16-116040810135-4.
Конфликт интересов. Авторы заявляют об отсутствии конфликта интересов.
Литература
- Филатова Е.Н., Солнцев Л.А., Преснякова Н.Б., Кулова Е.А., Уткин О.В. Определение некоторых иммунологических особенностей ВГЧ-6-опосредованного инфекционного мононуклеоза у детей методом дискриминантного анализа. Инфекция и иммунитет 2018; 8(2): 223–229, https://doi.org/10.15789/2220-7619-2018-2-223-229.
- Dojcinov S., Fend F., Quintanilla-Martinez L. EBV-positive lymphoproliferations of B- T- and NK-cell derivation in non-immunocompromised hosts. Pathogens 2018; 7(1): 28, https://doi.org/10.3390/pathogens7010028.
- Nakayama-Ichiyama S., Yokote T., Oka S., Iwaki K., Kobayashi K., Hirata Y., Hiraoka N., Takayama A., Akioka T., Miyoshi T., Takubo T., Tsuji M., Hanafusa T. Diffuse large B-cell lymphoma, not otherwise specified, associated with coinfection of human herpesvirus 6 and 8. J Clin Oncol 2011; 29(21): e636–e637, https://doi.org/10.1200/jco.2011.35.1254.
- Razzaque A. Oncogenic potential of human herpesvirus-6 DNA. Oncogene 1990; 5(9): 1365–1370.
- Li B., Zeng Q. Personalized identification of differentially expressed pathways in pediatric sepsis. Mol Med Rep 2017; 16(4): 5085–5090, https://doi.org/10.3892/mmr.2017.7217.
- Omar M., Klawonn F., Brand S., Stiesch M., Krettek C., Eberhard J. Transcriptome-wide high-density microarray analysis reveals differential gene transcription in periprosthetic tissue from hips with chronic periprosthetic joint infection vs aseptic loosening. J Arthroplasty 2017; 32(1): 2342–2340, https://doi.org/10.1016/j.arth.2016.06.036.
- Sano D., Tazawa M., Inaba M., Kadoya S., Watanabe R., Miura T., Kitajima M., Okabe S. Selection of cellular genetic markers for the detection of infectious poliovirus. J Appl Microbiol 2018; 124(4): 1001–107, https://doi.org/10.1111/jam.13621.
- Scicluna B.P., van Vught L.A., Zwinderman A.H., Wiewel M.A., Davenport E.E., Burnham K.L., Nürnberg P., Schultz M.J., Horn J., Cremer O.L., Bonten M.J., Hinds C.J., Wong H.R., Knight J.C., van der Poll T.; MARS consortium. Classification of patients with sepsis according to blood genomic endotype: a prospective cohort study. Lancet Respir Med 2017; 5(10): 816–826, https://doi.org/10.1016/s2213-2600(17)30294-1.
- Knyazev D.I., Starikova V.D., Utkin О.V., Solntsev L.А., Sakharnov N.А., Efimov E.I. Splicing-sensitive DNA-microarrays: peculiarities and applicationin biomedical research (review). Sovremennye tehnologii v medicine 2015; 7(4): 162–173, https://doi.org/10.17691/stm2015.7.4.23.
- Pirooznia M., Yang J.Y., Yang M.Q., Deng Y. A comparative study of different machine learning methods on microarray gene expression data. BMC Genomics 2008; 9(Suppl 1): S13, https://doi.org/10.1186/1471-2164-9-s1-s13.
- Solntsev L.A., Starikova V.D., Sakharnov N.A., Knyazev D.I., Utkin O.V. Strategy of probe selection for studying mRNAs that participate in receptor-mediated apoptosis signaling. Mol Biol 2015; 49(3): 457–465, https://doi.org/10.1134/s0026893315030164.
- Wu Z., Aryee M.J. Subset quantile normalization using negative control features. J Comput Biol 2010; 17(10): 1385–1395, https://doi.org/10.1089/cmb.2010.0049.
- Torices S., Alvarez-Rodríguez L., Grande L., Varela I., Muñoz P., Pascual D., Balsa A., López-Hoyos M., Martinez-Taboada V., Fernández-Luna J.L. A truncated variant of ASCC1, a novel inhibitor of NF-B, is associated with disease severity in patients with rheumatoid arthritis. J Immunol 2015; 195(11): 5415–5420, https://doi.org/10.4049/jimmunol.1501532.
- Michel M., Wilhelmi I., Schultz A.-S., Preussner M., Heyd F. Activation-induced tumor necrosis factor receptor-associated factor 3 (Traf3) alternative splicing controls the noncanonical nuclear factor κB pathway and chemokine expression in human T cells. J Biol Chem 2014; 289(19): 13651–13660, https://doi.org/10.1074/jbc.m113.526269.
- Filatova E.N., Utkin O.V. The role of noncoding MRNA isoforms in the regulation of gene expression. Russ J Genet 2018; 54(8): 879–887, https://doi.org/10.1134/s1022795418080057.
- Feoktistova M., Leverkus M. Programmed necrosis and necroptosis signalling. FEBS J 2014; 282(1): 19–31, https://doi.org/10.1111/febs.13120.
- Haan K.M., Kwok W.W., Longnecker R., Speck P. Epstein-Barr virus entry utilizing HLA-DP or HLA-DQ as a coreceptor. J Virol 2000; 74(5): 2451–2454, https://doi.org/10.1128/jvi.74.5.2451-2454.2000.