Спектр вариантов генов PRSS1, SPINK1, CTRC, CFTR и CPA1 у пациентов с хроническим панкреатитом в России
Цель исследования — определить спектр генетических факторов риска развития хронического панкреатита (ХП) у пациентов, проживающих в европейской части России.
Материалы и методы. В группу обследуемых вошло 105 пациентов с ХП с возрастом манифестации заболевания до 40 лет (средний возраст манифестации — 26,9 года). Контрольную группу составили 76 человек без признаков панкреатита. Диагноз «хронический панкреатит» установлен на основании клинических признаков и результатов лабораторно-инструментального обследования. Генетическое обследование пациентов проведено с использованием технологии секвенирования нового поколения (NGS) и включало в себя таргетное секвенирование всех экзонов и экзон-интронных участков генов PRSS1, SPINK1, CTRC, CFTR и CPA1. Также определяли генотип по локусу rs61734659 гена PRSS2.
Результаты. Генетические факторы риска развития ХП выявлены у 61% пациентов. Патогенные и вероятно-патогенные варианты, ассоциированные с риском развития ХП, обнаружены в следующих генах: CTRC (37,1% больных), CFTR (18,1%), SPINK1 (8,6%), PRSS1 (8,6%) и CPA1 (6,7%). Определены повторяющиеся варианты в генах среди российских больных: ген CTRC — c.180C>T (rs497078), c.760C>T (rs121909293), c.738_761del24 (rs746224507); кумулятивный показатель отношения шансов (OR) для всех рисковых аллелей составил 1,848 (95% CI: 1,054–3,243); ген CFTR — c.3485G>T (rs1800120), c.1521_1523delCTT (p.Phe508del, rs113993960) и c.650A>G (rs121909046); OR=2,432 (95% CI: 1,066–5,553). В генах SPINK1, PRSS1 и CPA1 патогенные варианты обнаружены только в группе больных. К частым вариантам гена SPINK1 можно отнести c.101A>G (p.Asn34Ser, rs17107315) и c.194+2T>C (rs148954387); гена PRSS1 — c.86A>T (p.Asn29Ile, rs111033566); гена CPA1 — c.586-30C>T (rs782335525) и c.696+23_696+24delGG. OR развития ХП для генотипа c.180TT (rs497078) CTRC по рецессивной модели (TT vs. CT+CC) составил 7,05 (95% CI: 0,86–263, p=0.011). В гене CTRC вариант с.493+49G>C (rs6679763) оказался доброкачественным; вариант c.493+51C>A (rs10803384) был частым как среди больных, так и среди здоровых людей и не продемонстрировал протективного действия. Протективный фактор c.571G>A (p.Gly191Arg, rs61734659) гена PRSS2 выявлен только в группе здоровых лиц и подтвердил свое протективное значение. У 12,4% больных ХП обнаружены рисковые факторы в 2–3 генах.
Заключение. Секвенирование кодирующих участков генов PRSS1, SPINK1, CTRC, CFTR и CPA1 позволило выявить генетические факторы риска развития ХП в 61% случаев. Определение генетической причины заболевания даст возможность спрогнозировать его течение, провести профилактику у родственников пробанда и в будущем — приблизиться к персонализированному лечению пациента.
Введение
Хронический панкреатит (ХП) характеризуется хроническим воспалительным процессом в ткани поджелудочной железы, которая по мере прогрессирования заболевания замещается фиброзной тканью. Частота встречаемости ХП среди взрослых людей достигает 1 на 2632 человека, среди детей — 1 на 7692 [1, 2]. Однако в силу разных причин может присутствовать определенная доля нераспознанных случаев данного заболевания.
В целом факторы риска, способствующие развитию ХП, хорошо известны. К ним относятся курение, чрезмерный прием алкоголя, наличие желчнокаменной болезни, воздействие стрессовых факторов, гипертриглицеридемия, гиперкальциемия, травмы и пороки развития поджелудочной железы (ПЖ), прием некоторых лекарственных средств и др. [3]. Однако в клинической практике зачастую возникают ситуации, когда установить точную причину заболевания не удается. В таких случаях ставится диагноз «идиопатический панкреатит». Именно для таких пациентов наиболее важно проводить генетическое обследование, поскольку примерно в 20% случаев удается выявить наследственно-обусловленную форму ХП [2, 4].
Согласно последней версии классификации основных причин ХП (TIGAR-O Version 2), существенная роль в патогенезе заболевания отводится генетическим факторам [5].
Продукты генов PRSS1 (катионный трипсиноген 1-го типа), SPINK1 (секреторный панкреатический ингибитор трипсина 1-го типа), CTRC (химотрипсин С), CFTR (муковисцидозный трансмембранный регулятор проводимости) и CPA1 (карбоксипептидаза А1) имеют существенное значение для функционирования ПЖ [4].
Цельисследования — определить структуру генетических причин хронического панкреатита у пациентов, проживающих в европейской части России, путем секвенирования всей кодирующей последовательности генов PRSS1, SPINK1, CTRC, CFTR и CPA1.
Материалы и методы
В группу обследуемых вошло 105 больных ХП с возрастом манифестации заболевания до 40 лет (средний возраст на момент забора крови — 35,1 года; средний возраст манифестации ХП — 26,9 года). Из них было 65 пациентов мужского пола (средний возраст на момент забора крови — 36,3 года; средний возраст манифестации ХП — 28,7 года) и 40 пациентов женского пола (средний возраст на момент забора крови — 33,2 года; средний возраст манифестации ХП — 23,9 года). Контрольную группу составили 76 человек соответствующего пола и возраста без признаков панкреатита (средний возраст — 31,6 года).
Все обследуемые дали добровольное информированное согласие на участие в исследовании. Работа одобрена локальным этическим комитетом Московского клинического научно-практического центра им. А.С. Логинова Департамента здравоохранения города Москвы (протокол №8 от 2015 г.) и соответствует требованиям Хельсинкской декларации (2013).
Диагноз «хронический панкреатит» устанавливали на основании клинических признаков и результатов лабораторно-инструментального обследования. Генетическое исследование проводили с использованием секвенирования нового поколения (NGS, от англ. next generation sequencing), которое включало в себя таргетное секвенирование всех экзонов и экзон-интронных участков генов PRSS1 (5 экзонов), SPINK1 (4 экзона), CTRC (8 экзонов), CFTR (27 экзонов) и CPA1 (10 экзонов). Кроме того, в своем исследовании мы решили проверить роль протективного фактора rs61734659 гена PRSS2 в отношении развития ХП у российских пациентов, поэтому включили данную область гена в разработанную таргетную генетическую панель.
Для генетического анализа из образцов цельной венозной крови обследуемых выделяли ДНК с использованием набора реагентов DNeasy Blood & Tissue Kit (QIAGEN, Германия) на автоматизированной системе выделения ДНК, РНК и белков QIAcube (QIAGEN, Германия) по стандартному протоколу производителя.
Пробоподготовка образцов к секвенированию включала ПЦР с использованием праймерной панели из 67 пар праймеров (с общим покрытием 22 693 нуклеотида), разработанной специально для данного исследования с помощью программы Ion AmpliSeq Designer (Thermo Fisher Scientific, США). Нуклеотидные последовательности анализируемых генов получены из базы данных NCBI (https://www.ncbi.nlm.nih.gov/). Целевую амплификацию проводили с помощью реагентов для ПЦР («АмплиСенс»; ЦНИИ эпидемиологии Роспотребнадзора, Россия) на приборе QuantStudio 5 Real-Time PCR Systems (Thermo Fisher Scientific, США).
Приготовление NGS-библиотек осуществляли с использованием реагентов T4 Polynucleotide Kinase (New England Biolabs, США) и T4 DNA Ligase (New England Biolabs, США) согласно инструкциям производителей с незначительными модификациями.
Очистку продукта ПЦР, а также NGS-библиотек на всех стадиях от компонентов реакционных смесей выполняли с помощью карбоксилированных магнитных частиц Sera-Mag SpeedBeads (Sigma, США). Концентрацию ДНК выделенных образцов, продукта ПЦР и финальных библиотек измеряли флуорометрическим методом с использованием набора реагентов Qubit dsDNA HS Assay Kit (Thermo Fisher Scientific, США) на приборе Qubit 2.0 Fluorometer (Thermo Fisher Scientific, США). Оценку качества готовых библиотек проводили на чипе методом капиллярного электрофореза на приборе Agilent 2100 Bioanalyzer (Agilent Technologies, США) с помощью набора реагентов Agilent High Sensitivity DNA Kit (Agilent Technologies, США).
Секвенирование на платформе Ion S5 (Thermo Fisher Scientific, США) выполняли с использованием набора реагентов Ion 520 & Ion 530 Kit-Chef (Thermo Fisher Scientific, США) и полупроводникового чипа Ion 530™ Chip Kit (Thermo Fisher Scientific, США).
Биоинформатическая обработка данных NGS-секвенирования включала в себя удаление низкокачественных прочтений с помощью программы PRINSEQ-lite [6]; картирование на референсный геном человека (GRCh38.p7, PRJNA31257) с использованием Burrows-Wheeler Aligner (BWA-MEM, v_0.7.13) [6]; поиск вариантов нуклеотидной последовательности пакетом программ Genome Analysis Toolkit (GATK version 4.0.11.0) [7]. Пакеты SAMtools v_1.3.1 [8] и Picard toolkit v_2.18.17 применяли для операций с файлами sam/bam; программу VEP [9] с использованием кэша 94_GRCh38 — для первичной аннотации вариантов.
Валидацию выявленных автоматическим анализом вариантов нуклеотидной последовательности осуществляли вручную на графическом пользовательском интерфейсе для просмотра сборок и выравниваний Tablet [10].
Клиническую интерпретацию обнаруженных вариантов проводили с использованием следующих электронных ресурсов и баз данных: dbSNP (https://www.ncbi.nlm.nih.gov/snp/), ClinVar (https://www.ncbi.nlm.nih.gov/clinvar/), Cystic Fibrosis Mutation Database (http://www.genet.sickkids.on.ca), CFTR2 (https://cftr2.org), CFTR-France (https://cftr.iurc.montp.inserm.fr), OMIM (https://www.omim.org/), pancreasgenetics.org.
Статистический анализ выполняли с помощью стандартного пакета программ R [11]. Для определения наличия ассоциации между полиморфизмами анализируемых генов и развитием ХП рассчитывали отношение шансов (OR, от англ. odds ratio) и относительный риск (RR, от англ. relative risk) по стандартным формулам с расчетом 95% доверительного интервала (CI). Для определения уровня значимости различий значений применяли t-критерий Стьюдента с расчетом χ2 и уровня значимости р. За уровень значимости различий значений между выборками принимали p≤0,05. Стандартное отклонение процентной доли рассчитывали по формуле:
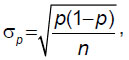
где р — процентная доля, σр — стандартное отклонение процентной доли, n — количество наблюдений.
Результаты
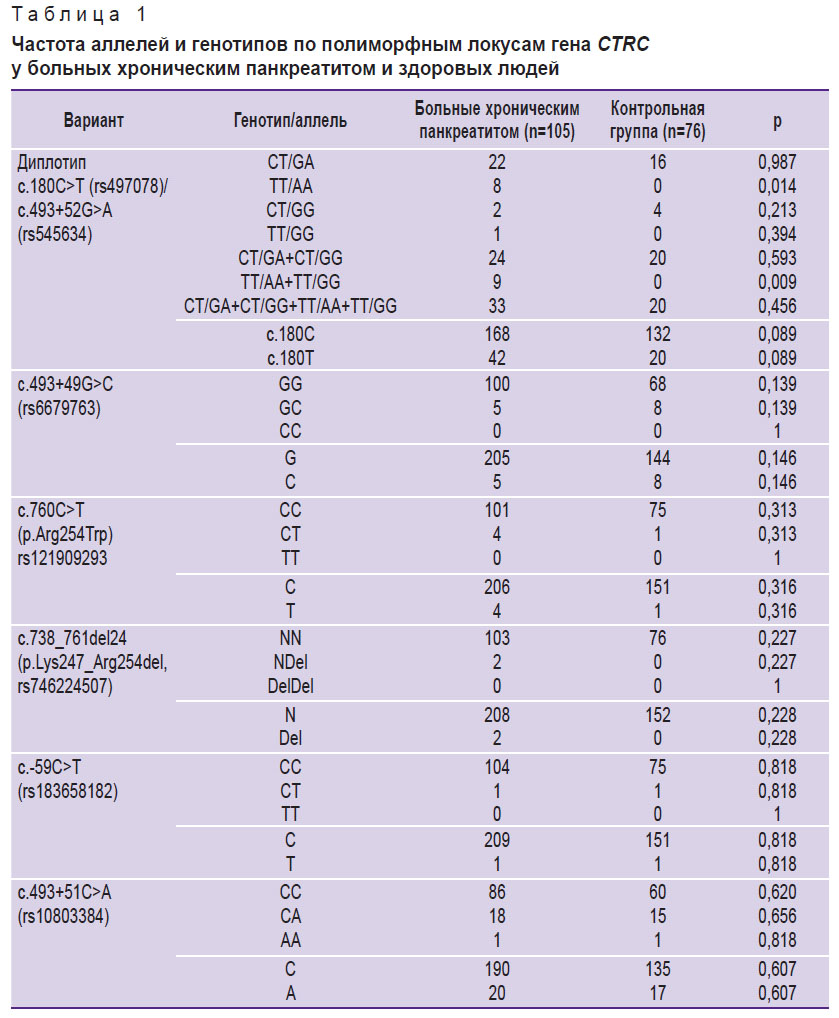
Варианты гена CTRC. Наиболее часто генетические факторы риска развития заболевания обнаруживали в гене CTRC. В большинстве случаев пациенты являлись гетерозиготными (n=22) или гомозиготными (n=8) носителями так называемого рискового диплотипа c.180C>T (rs497078)/c.493+52G>A (rs545634), который был выявлен у 30 человек. Еще у трех больных ХП был обнаружен вариант c.180C>T (rs497078) без сочетания с c.493+52G>A (rs545634): в двух случаях в гетерозиготной и в одном случае — в гомозиготной форме. Таким образом, 33 человека из 105 (31,4%) обладали вариантом c.180C>T (rs497078) гена CTRC. Второй частой находкой в гене CTRC у больных ХП стал вариант неизвестного клинического значения — с.493+49G>C (rs6679763), который встретился у пяти человек в гетерозиготной форме (4,8%). Еще у четырех пациентов (3,8%) был выявлен патогенный вариант c.760C>T (p.Arg254Trp, rs121909293) в гетерозиготной форме и у двух больных (1,9%) — патогенный вариант c.738_761del24 (p.Lys247_Arg254del, rs746224507), тоже в гетерозиготной форме. У одного больного обнаружен вариант неизвестного клинического значения — c.-59C>T (rs183658182) в гетерозиготной форме (табл. 1).
|
Таблица 1. Частота аллелей и генотипов по полиморфным локусам гена CTRC у больных хроническим панкреатитом и здоровых людей |
В группе контроля выявлен 21 человек (27,6%) с изменениями гена CTRC в гетерозиготной форме. Из них 16 человек оказались гетерозиготными носителями варианта c.180C>T (rs497078) в виде диплотипа c.180C>T (rs497078)/c.493+52G>A (rs545634), четыре человека — гетерозиготными носителями варианта c.180C>T (rs497078) без его сочетания с вариантом c.493+52G>A (rs545634), один человек обладал вариантом c.760C>T (p.Arg254Trp, rs121909293) (см. табл. 1).
При сравнении частоты аллеля c.180T (rs497078) в группе больных ХП (20%) и группе контроля (13,2%) различия были статистически незначимыми (χ2=2,908; p=0,089). Однако гомозиготность по диплотипу c.180C>T (rs497078)/c.493+52G>A (rs545634) наблюдалась исключительно у пациентов, страдающих ХП (см. табл. 1). Последнее говорит о существенном влиянии гомозиготного генотипа по варианту c.180C>T (rs497078) гена CTRC на развитие воспалительных изменений в ПЖ. Так, OR для генотипа c.180TT по рецессивной модели (TT vs. CT+CC) составил 7,05 (95% CI: 0,86–263, p=0,011).
Интересно, что вариант с.493+49G>C (rs6679763) был выявлен в гетерозиготной форме у 8 из 76 здоровых людей (10,5%). При сравнении частот различных генотипов и аллелей по полиморфному локусу rs6679763 между группой больных и группой здоровых лиц не обнаружено статистически значимых отличий (p=0,139 и p=0,146 соответственно). Это позволяет сделать заключение об отсутствии патогенного эффекта данного варианта в отношении риска развития ХП.
Вариант неизвестного клинического значения c.-59C>T (rs183658182) в гене CTRC выявлен в гетерозиготной форме у одного человека контрольной группы, что косвенно может свидетельствовать в пользу его малой значимости в отношении развития ХП. Вариант c.760C>T (p.Arg254Trp, rs121909293) обнаружен у одного представителя группы контроля (женщины 23 лет без признаков ХП на момент сдачи крови в исследование). Вариант c.738_761del24 (p.Lys247_Arg254del, rs746224507/rs515726210) не выявлен ни у одного из обследуемых контрольной группы.
Помимо указанных выше находок как в группе больных ХП, так и среди контрольных образцов в гене CTRC обнаружен частый полиморфизм c.493+51C>A (rs10803384), который по информации из базы данных pancreasgenetics.org имеет протективное значение в отношении развития заболевания. В нашем исследовании существенных отличий в частоте генотипов и аллелей по данному полиморфному локусу CTRC между двумя группами не выявлено (см. табл. 1).
Таким образом, патогенные варианты c.760C>T (p.Arg254Trp, rs121909293) и c.738_761del24 (p.Lys247_Arg254del, rs746224507) и генетические факторы риска c.180C>T (rs497078)/c.493+52G>A (rs545634) гена CTRC выявлены у 39 из 105 обследованных пациентов с ХП (37,1%).
К наиболее частым и значимым вариантам гена CTRC, обнаруживаемым у пациентов с ХП, относятся диплотип c.180C>T (rs497078)/c.493+52G>A (rs545634) и вариант c.180C>T (rs497078) — 84,6% всех находок в данном гене; c.760C>T (p.Arg254Trp, rs121909293) — 10,3% всех находок в данном гене; c.738_761del24 (p.Lys247_Arg254del, rs746224507) — 5,1% всех находок в данном гене.
Частота обнаружения какого-либо из упомянутых выше рисковых аллелей гена CTRC в группе больных ХП оказалась равной 22,9% (48 аллелей из 210), в то время как тот же показатель для контрольной группы обследуемых составил 13,8% (21 аллель из 152) (χ2=4,672; p=0,031). OR развития ХП при наличии рискового аллеля гена CTRC составило 1,848 (95% CI: 1,054–3,243; стандартная ошибка — 0,287).
Варианты гена CFTR. Из 105 пациентов с ХП у 19 человек (18,1%) выявлены патогенные или вероятно-патогенные варианты гена CFTR в разных комбинациях: c.2620-6T>C, c.224G>A (p.Arg75Gln), c.451C>A (p.Gln151Lys), c.650A>G (p.Glu217Gly), c.1079C>T (p.Thr360Ile), c.2012delT (p.Leu671Ter), c.1516A>G (p.Ile506Val), c.1521_1523delCTT (p.Phe508del), c.1399C>T (p.Leu467Phe), c.1584G>A (p.Glu528=), c.3485G>T (p.Arg1162Leu), c.2991G>C (p.Leu997Phe), c.1969A>G (p.Arg657Gly), c.2619+86delT. Три патогенных варианта, а именно c.3485G>T (p.Arg1162Leu), c.1521_1523delCTT (p.Phe508del) и c.650A>G (p.Glu217Gly), оказались повторяющимися и были обнаружены у семи больных (6,7%). Один из вариантов гена CFTR (c.2619+86delT) выявлен впервые в нашем исследовании и присутствовал у пяти пациентов (во всех случаях в гомозиготной форме). Более подробную характеристику пациентов и обнаруженных у них вариантов в гене CFTR можно найти в опубликованной нами ранее работе [12].
В контрольной группе также были обнаружены патогенные или вероятно-патогенные варианты гена CFTR. Так, 8 из 76 (10,5%) человек без признаков панкреатита обладали вариантами c.650A>G (p.Glu217Gly), c.3854C>T (p.Ala1285Val), c.443T>C (p.Ile148Thr), c.1584G>A (p.Glu528=), c.224G>A (p.Arg75Gln), c.3532_3535dup (p.Thr1179IlefsTer17) в гетерозиготной форме.
Частота обнаружения патологически измененного аллеля гена CFTR в группе больных ХП составила 11,9% (25 аллелей из 210), в то время как тот же показатель для контрольной группы обследуемых составил 5,3% (8 аллелей из 152) (χ2=4,695; p=0,031). OR развития ХП при наличии патологического аллеля гена CFTR оказалось равным 2,432 (95% CI: 1,066–5,553; стандартная ошибка — 0,421).
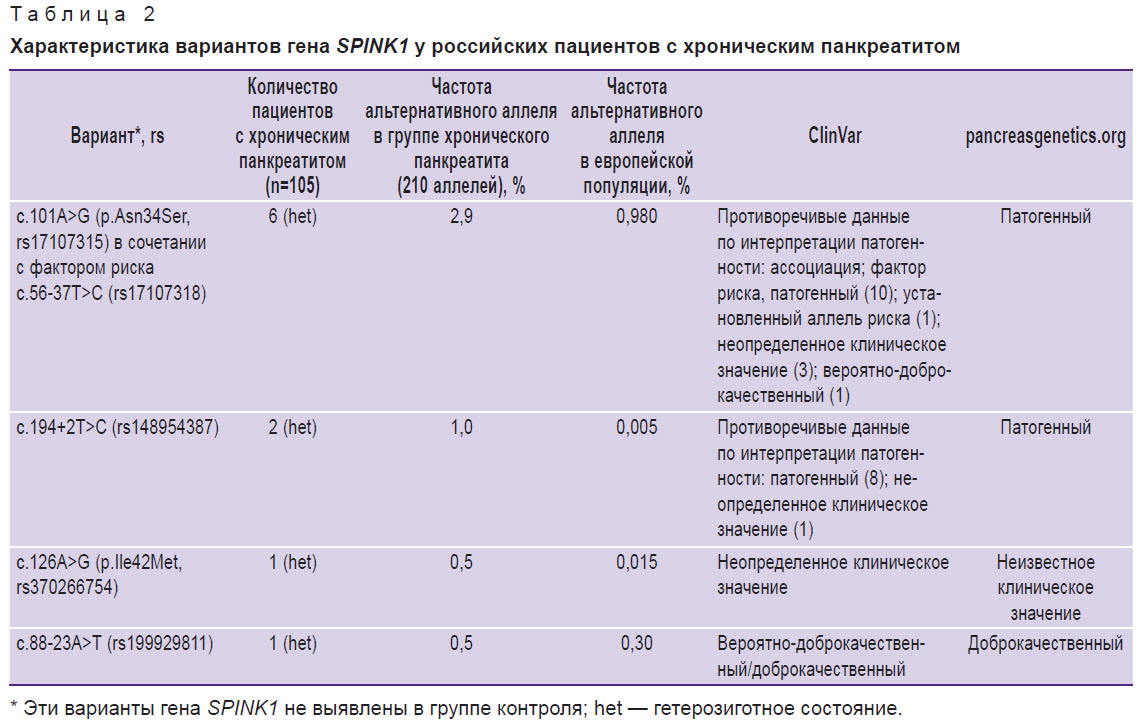
Варианты гена SPINK1. Патогенные и вероятно-патогенные варианты гена SPINK1 выявлены лишь в группе больных ХП (все в гетерозиготной форме). Восемь пациентов (7,6%) оказались носителями известных патогенных вариантов гена SPINK1: 6 больных имели патогенный вариант c.101A>G (p.Asn34Ser, rs17107315) в сочетании с фактором риска c.56-37T>C (rs17107318), 2 пациента — вариант c.194+2T>C (rs148954387) (табл. 2).
|
Таблица 2. Характеристика вариантов гена SPINK1 у российских пациентов с хроническим панкреатитом |
Кроме того, в одном случае выявлен вариант неизвестного клинического значения c.126A>G (p.Ile42Met, rs370266754) и еще у двух пациентов обнаружен вариант c.88-23A>T (rs199929811), который по информации базы данных pancreasgenetics.org является доброкачественным и не приводит к повышению риска развития ХП. Стоит отметить, что этот вариант (rs199929811) не обнаружен ни у одного из обследуемых контрольной группы, что поднимает вопрос о пересмотре данных о клинической интерпретации этого варианта.
Таким образом, без учета доброкачественного варианта rs199929811 гена SPINK1, 9 из 105 больных ХП (8,6%) оказались носителями патогенных изменений. К наиболее частым находкам можно отнести варианты c.101A>G (p.Asn34Ser, rs17107315) в сочетании с фактором риска c.56-37T>C (rs17107318) — 66,7% всех находок в данном гене и c.194+2T>C (rs148954387) — 22,2% всех находок в данном гене.
Варианты гена PRSS1. Так же, как и в случае гена SPINK1, патогенные и вероятно-патогенные варианты гена PRSS1 были выявлены только в группе больных ХП (все в гетерозиготной форме).
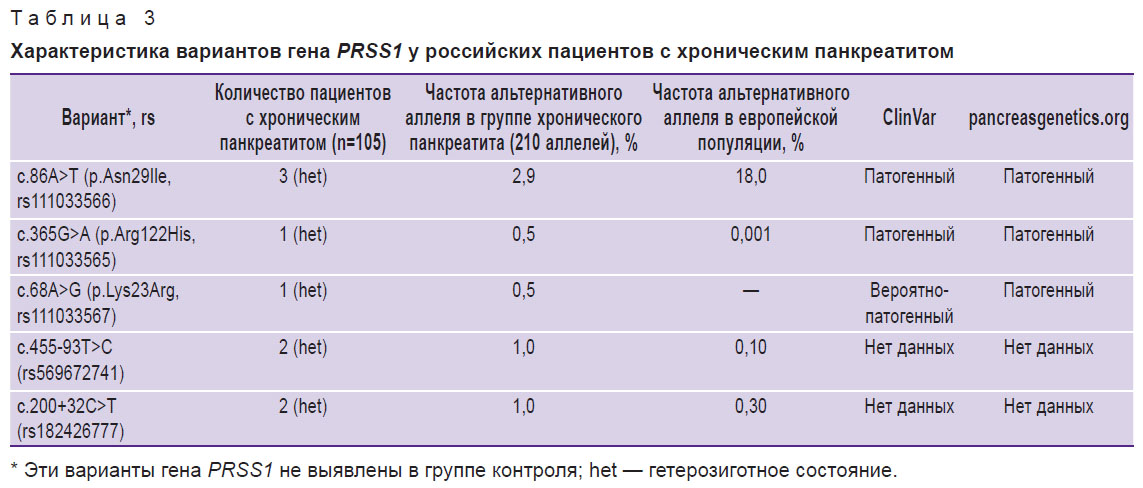
Из 105 больных ХП у 5 (4,8%) обнаружены известные патогенные варианты гена PRSS1: 3 пациента оказались носителями варианта c.86A>T (p.Asn29Ile, rs111033566), 1 пациент — варианта c.365G>A (p.Arg122His, rs111033565) и еще 1 пациент — варианта c.68A>G (p.Lys23Arg, rs111033567) (табл. 3).
|
Таблица 3. Характеристика вариантов гена PRSS1 у российских пациентов с хроническим панкреатитом |
Кроме того, в ходе секвенирования гена PRSS1 в группе больных ХП выявлены дополнительные находки. Вариант c.455-93T>C (rs569672741) обнаружен у двух пациентов в гетерозиготной форме. По информации из базы dbSNP, частота аллеля c.455-93C в европейской популяции составляет 0,1%. В базе ClinVar и в специализированной базе pancreasgenetics.org данный вариант не аннотирован. Следует отметить, что в нашем исследовании в группе контроля не выявлена эта замена гена PRSS1.
Аналогичная ситуация наблюдалась с интронным вариантом c.200+32C>T (rs182426777), который был обнаружен в гетерозиготной форме у двух больных ХП. Этот вариант не аннотирован в упомянутых выше базах и встречается в европейской популяции с частотой около 0,3%.
Таким образом, к повторяющимся патогенным вариантам PRSS1 у российских пациентов с ХП можно отнести c.86A>T (p.Asn29Ile, rs111033566), который встретился у трех больных.
Совокупная частота рисковых аллелей гена PRSS1 в группе больных ХП составила 4,3% (9 аллелей), в группе здоровых лиц она была равна нулю (χ2=6,680; p=0,01).
Варианты гена CPA1. При секвенировании всей кодирующей последовательности гена CPA1 выявлено несколько вариантов, которые требуют дальнейшего изучения.
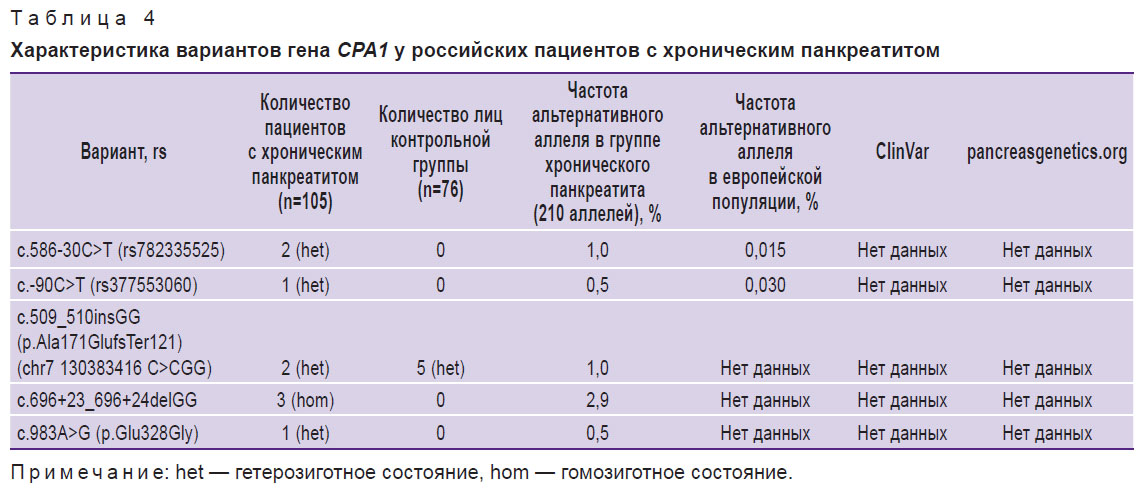
Из 105 пациентов с ХП 2 человека (1,9%) оказались гетерозиготными носителями варианта c.586-30C>T (rs782335525), который не представлен в базах ClinVar и pancreasgenetics.org и не обнаружен в контрольных образцах. Этот вариант встречается в европейской популяции с частотой 0,015%.
У одного пациента выявлен вариант c.-90C>T (rs377553060) в гетерозиготной форме, который также не описан в упомянутых базах данных и не найден в группе контроля. Данный вариант встречается в европейской популяции с частотой 0,03%.
У трех неродственных больных (2,8%) ХП выявлен вариант c.696+23_696+24delGG (chr7 130383816 TGG>TG,T) в гомозиготной форме. Вариант ранее не описан в литературе, не представлен в геномных базах данных, частота его не установлена. В контрольной выборке настоящего исследования он не зарегистрирован. Сегрегационный генетический анализ в семьях пациентов не проводился.
Один больной ХП оказался гетерозиготным носителем миссенс-варианта c.983A>G (p.Glu328Gly), который так же, как и предыдущий вариант, выявлен в нашей когорте больных ХП впервые.
У двух пациентов с ХП (1,9%) обнаружен вариант c.509_510insGG (p.Ala171GlufsTer121) (chr7 130383416 C>CGG ) в гетерозиготной форме. Данный вариант гена CPA1 не аннотирован в мировых геномных базах данных. Стоит отметить, что в этой области гена ранее описаны патогенные варианты типа миссенс с неопределенным клиническим значением. Однако в нашем исследовании вариант c.509_510insGG также выявлен в гетерозиготной форме у 5 из 76 здоровых людей контрольной группы (6,6%), что говорит об отсутствии ассоциации между его наличием и повышенным риском развития панкреатита (табл. 4).
|
Таблица 4. Характеристика вариантов гена CPA1 у российских пациентов с хроническим панкреатитом |
Таким образом, совокупная частота предположительно рисковых аллелей гена CPA1 в группе больных ХП составила 4,8% (10 аллелей), в группе здоровых лиц — 0 (χ2=7,444; p=0,007).
Протективный фактор гена PRSS2. Протективный фактор c.571G>A (p.Gly191Arg, rs61734659) гена PRSS2 выявлен только в группе здоровых лиц, семь из которых оказались гетерозиготными носителями данного полиморфизма. Таким образом, частота аллеля c.571A в группе больных ХП составила 0%, в то время как в группе здоровых лиц она была равна 4,6% (χ2=9,862; p=0,002), т.е. протективный эффект аллеля c.571A по полиморфному локусу rs61734659 подтвердился.
Сводные данные по выявленным генетическим факторам риска. Семейный анамнез был отягощен ХП у 26,5% пациентов.
В результате проведенного генетического исследования у 64 из 105 пациентов (61%) с возрастом манифестации ХП до 40 лет выявлены те или иные значимые находки в разных комбинациях в генах PRSS1, SPINK1, CTRC, CFTR и CPA1.
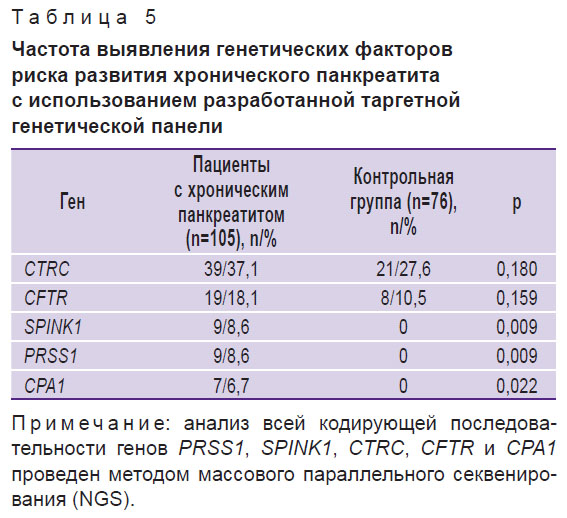
Сводные данные по результатам секвенирования с использованием разработанной нами таргетной генетической панели генов представлены в табл. 5.
|
Таблица 5. Частота выявления генетических факторов риска развития хронического панкреатита с использованием разработанной таргетной генетической панели |
Наиболее часто у пациентов с ХП встречаются генные варианты CTRC (37,1% больных). Далее по убыванию — патогенные и вероятно-патогенные варианты генов CFTR (18,1%), SPINK1 (8,6%), PRSS1 (8,6%) и CPA1 (6,7%).
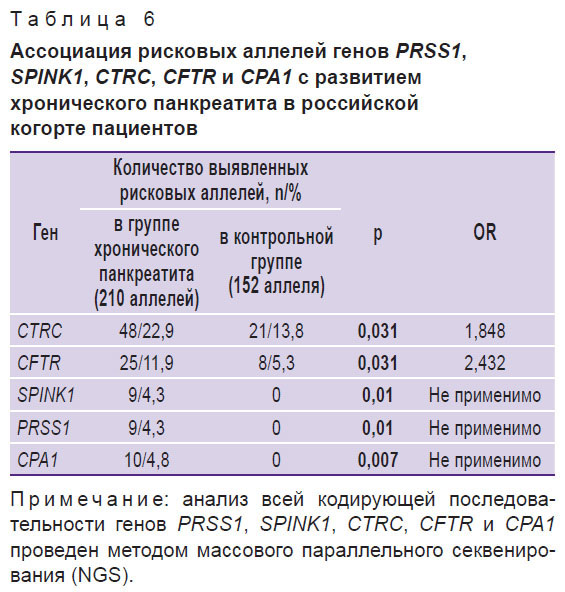
Вклад вариантов генов PRSS1, SPINK1, CTRC, CFTR и CPA1 в вероятность развития ХП в когорте российских пациентов представлен в табл. 6, из которой видно, что OR развития ХП для вариантов генов CTRC и CFTR составляет 1,848 и 2,432 соответственно. Максимальные риски развития хронического воспаления в ПЖ наблюдаются у носителей патогенных вариантов в генах PRSS1, SPINK1 и CPA1, что согласуется с принятой на сегодняшний день аутосомно-доминантной теорией наследования PRSS1- и SPINK1-ассоциированного ХП (OMIM #167800).
|
Таблица 6. Ассоциация рисковых аллелей генов PRSS1, SPINK1, CTRC, CFTR и CPA1 с развитием хронического панкреатита в российской когорте пациентов |
У части больных ХП наблюдалось сочетание патогенных вариантов и рисковых факторов сразу в нескольких генах. Так, 11 пациентов оказались носителями двух генетических факторов риска, два пациента имели повреждения сразу трех генов одновременно (табл. 7).

|
Таблица 7. Сочетание генетических факторов риска у пациентов с хроническим панкреатитом |
Обсуждение
В настоящем исследовании, которое включало 105 больных ХП, наблюдалось некоторое преобладание лиц мужского пола, что в целом согласуется с данными мировой литературы, согласно которым соотношение мужчин и женщин среди больных ХП равно 1,05 [13].
Исходя из предположения, что наследственно-обусловленные формы ХП манифестируют в более раннем возрасте, в группу обследуемых были включены больные с возрастом манифестации заболевания до 40 лет. В мировой литературе имеются работы, в которых применялся критерий отбора пациентов с ХП по этому принципу [14–16]. Так, в исследовании W.B. Zou с соавт. [17] показано, что средний возраст манифестации панкреатита у пациентов с молекулярными находками в генах SPINK1, PRSS1, CTRC и CFTR составлял 29,70±14,84 года, в то время как у больных без мутаций в этих генах первые клинические признаки заболевания возникали в возрасте 43,01±15,97 года [17].
Реализация этого подхода позволила выявить генетические предикторы развития ХП у большей части (61%) больных, что в целом согласуется с данными литературы. В частности, в исследовании W.B. Zou с соавт. [17] при секвенировании генов SPINK1, PRSS1, CTRC и CFTR в обширной группе пациентов, страдающих ХП, патогенные варианты выявлены в 50,42% случаев. При этом в группе контроля мутации перечисленных генов авторами обнаружены лишь в 5,94% случаев (OR=16,12; p<0,001) [17].
Состав разработанной нами генетической панели продиктован результатами оригинальных исследований и данными геномных баз, согласно которым гены PRSS1, SPINK1, CTRC, CFTR и CPA1 играют существенную роль в молекулярном патогенезе заболевания [18]. Ген PRSS1 ответственен за выработку катионного трипсиногена 1-го типа, ген SPINK1 — секреторного панкреатического ингибитора трипсина 1-го типа, ген CTRC — химотрипсина С, ген CFTR — муковисцидозного трансмембранного регулятора проводимости, ген CPA1 связан с неправильным свертыванием белка карбоксипептидазы А1 и стрессом эндоплазматического ретикулума в клетках ПЖ [4]. Стоит отметить, что для наследственных форм ХП, связанных с мутациями в генах PRSS1 (MIM# 276000) и SPINK1 (MIM# 167790), характерен аутосомно-доминантный тип наследования.
Интересными представляются результаты анализа всей кодирующей последовательности упомянутых генов с помощью NGS для выявления повторяющихся патогенных вариантов и верификации факторов риска, характерных для российской популяции. До сих пор больные ХП в РФ в таком объеме не обследовались. На сегодняшний день в большинстве случаев диагностика генетических причин ХП в нашей стране сводится к поиску нескольких частых мутаций генов PRSS1 и SPINK1. В отдельных случаях пациентам проводят секвенирование кодирующей последовательности этих генов, что, согласно полученным в настоящем исследовании данным, является недостаточным и может пропускать генетические факторы риска ХП у пациентов в большом проценте случаев.
Кроме того, примененный в настоящем исследовании подход позволил сформировать перечень генетических маркеров, которые могут быть использованы в качестве скрининга на наиболее частые генетические факторы риска ХП у российских пациентов.
Самой частой молекулярной находкой среди пациентов с ХП оказались варианты гена CTRC, которые выявлены у 37,1% человек и, по-видимому, в данной популяции могут рассматриваться в качестве факторов риска (см. табл. 5). OR развития патологии при наличии рисковых генетических факторов данного гена составило 1,848 (p=0,031). При этом разные варианты этого гена вносят неравный вклад в риск развития ХП.
Диплотип c.180C>T (rs497078)/c.493+52G>A (rs545634) гена CTRC широко обсуждается в мировой литературе и является известным генетическим фактором риска развития ХП. В частности, в исследовании J. LaRusch с соавт. [19] продемонстрирована ассоциация аллеля c.180T (rs497078) с риском развития ХП в европейской популяции (показатель OR для генотипа CT составил 1,36; для генотипа TT — 3,98). В нашем исследовании выявлена ассоциация полиморфизма c.180C>T (rs497078) с повышенным риском развития ХП только в случае его наличия у пациента в гомозиготной форме (OR=7,05; p=0,011). Вероятно, отсутствие статистически значимой ассоциации гетерозиготного носительства rs497078 с повышенной вероятностью развития патологии в настоящем исследовании связано с особенностями изучаемой нами выборки, а именно с ранним возрастом манифестации ХП у пациентов, так как для лиц с патогенными вариантами этого гена характерна манифестация заболевания в более позднем возрасте.
Другие изменения гена CTRC у больных ХП оказывают гораздо большее влияние на риск развития данного заболевания. Так, патогенный вариант c.760C>T (p.Arg254Trp, rs121909293) в гетерозиготной форме встретился у четырех (3,8%) больных ХП и только у одного (1,3%) человека из контрольной группы. К тому же этот контрольный образец был получен от женщины 23 лет, у которой в силу возраста ХП мог еще просто не проявиться. Наши находки соотносятся с данными зарубежных коллег, в частности с результатами исследования J. Rosendahl с соавт. [20], в котором частота этого миссенс-варианта в выборке немецких больных с идиопатическим и наследственным ХП составила 2,1%, в то время как в контрольной группе он встретился лишь у 0,6% индивидов.
Аналогичная ситуация наблюдалась для патогенного варианта c.738_761del24 (p.Lys247_Arg254del, rs746224507/rs515726210), который выявлен в нашем исследовании лишь в группе больных ХП.
Таким образом, максимальный вклад в риск развития ХП в изучаемой выборке оказывают относительно редкие, но повторяющиеся варианты c.760C>T (p.Arg254Trp, rs121909293) и c.738_761del24 (p.Lys247_Arg254del, rs515726210).
Кроме того, показано, что вариант с.493+49G>C (rs6679763) гена CTRC, аннотированный в специализированной базе данных по генетических находкам при ХП (pancreasgenetics.org) как вариант неизвестного значения, не увеличивает риск развития заболевания, а значит, является доброкачественным (benign) вариантом.
Другой частый вариант c.493+51C>A (rs10803384) гена CTRC, аннотированный в базе pancreasgenetics.org как протективный в отношении развития ХП, не показал протективного действия на наших выборках обследуемых.
Еще один вариант гена CTRC неизвестного клинического значения — c.-59C>T (rs183658182) — требует дальнейшего изучения и по результатам нашей работы не может быть отнесен к рисковым факторам.
Находки в гене CFTR в группе больных ХП были на втором месте по распространенности. 18,1% больных оказались носителями разнообразных вариантов данного гена. Среди всех вариантов повторяющимися у российских больных ХП являются следующие: c.3485G>T (p.Arg1162Leu), c.1521_1523delCTT (p.Phe508del) и c.650A>G (p.Glu217Gly). Один из вариантов гена CFTR (c.2619+86delT), обнаруженный впервые в настоящем исследовании у пяти пациентов с ХП в гомозиготной форме, требует дальнейшего изучения с точки зрения как распространенности, так и функционального значения. Более подробную информацию о вариантах гена CFTR можно найти в работе М.М. Литвиновой с соавт. [12]. Кумулятивное OR развития ХП при наличии патологического аллеля CFTR составило 2,432, что в целом согласуется с исследованием W.B. Zou с соавт. [17], где аналогичный показатель составил 3,71.
Патогенные изменения в генах SPINK1 и PRSS1 выявлены у 8,6% больных ХП. Наши данные отличаются от уровня выявления мутаций в этих двух генах в исследовании A. Masamune с соавт. [21], где 41% больных оказались носителями патогенных вариантов в гене PRSS1 и 36% больных имели мутации гена SPINK1. Вероятно, такая разница связана с тем, что в настоящем исследовании мы не концентрировали внимание на отборе пациентов с отягощением семейного анамнеза панкреатитом, в то время как в упомянутой работе, проведенной в Японии (231 образец), наличие отягощения семейного анамнеза было одним из критериев отбора пациентов в группу обследуемых.
Патогенные варианты генов SPINK1 и PRSS1 вносят максимальный вклад в риск развития ХП у пациентов разных стран, поскольку продукты этих генов являются ключевыми участниками трипсин-зависимого патогенетического пути развития панкреатита. Очень часто у пациентов с мутациями генов SPINK1 и PRSS1 наблюдается отягощение семейного анамнеза, более того, возраст манифестации патологии обычно гораздо более ранний по сравнению с CFTR- или CTCR-ассоциированным ХП [17]. При сопоставлении результатов проведенного исследования с похожими работами зарубежных коллег обнаружено довольно большое сходство спектра генетических находок в генах SPINK1 и PRSS1 в российской и европейской популяции пациентов. К наиболее частым патогенным вариантам гена SPINK1 в российской выборке можно отнести миссенс-вариант c.101A>G (p.Asn34Ser, rs17107315) и мутацию сайта сплайсинга c.194+2T>C (rs148954387); к повторяющимся патогенным вариантам PRSS1 — c.86A>T (p.Asn29Ile, rs111033566), который, по данным зарубежных коллег, встречается у 5–21% пациентов с данным заболеванием [4].
Отмечены и отдельные варианты в генах SPINK1 и PRSS1, требующие дальнейшего изучения (SPINK1: rs370266754, rs199929811; PRSS1: rs569672741, rs182426777). В частности, вариант c.88-23A>T (rs199929811) гена SPINK1 аннотирован в базах данных как доброкачественный, однако результаты нашей работы поднимают вопрос о пересмотре клинической интерпретации этого варианта, так как он был выявлен лишь в когорте больных и не встретился ни у одного человека контрольной группы. Аналогичная ситуация наблюдалась с вариантами rs569672741 и rs182426777 гена PRSS1.
К настоящему времени ген CPA1 наименее изучен среди генов предрасположенности к ХП. Вместе с тем данные функционального анализа различных изменений CPA1 на клеточных линиях и модельных животных свидетельствует о значимом вкладе некоторых вариантов этого гена в патогенез заболевания [22]. По данным работы [4], наличие вариантов гена CPA1 у пациентов приводит к более ранней манифестации болезни. По результатам настоящего исследования, наибольшего внимания заслуживают следующие варианты гена CPA1: c.586-30C>T (rs782335525), c.-90C>T (rs377553060), c.983A>G (p.Glu328Gly) и c.696+23_696+24delGG (chr7 130383816 TGG>TG,T). Запланировано дальнейшее изучение данных вариантов в российской выборке больных ХП.
Таким образом, наиболее значимый вклад с точки зрения генетических причин ХП у российских пациентов продемонстрирован (в порядке убывания) для вариантов генов CTRC (выявлен в 37,1% случаев), CFTR (18,1%), SPINK1 (8,6%), PRSS1 (8,6%) и CPA1 (6,7%). Наши данные вполне согласуются с результатами, полученными при исследовании европейской когорты пациентов с ХП [23]. В то же время в крупном исследовании, проведенном в Китае, выявлены особенности, продемонстрировавшие максимальный вклад в риск развития идиопатического панкреатита вариантов генов SPINK1 (выявлен у 61,5% человек) и PRSS1 (13,5%). При этом на долю изменений генов CTRC и CFTR пришлось всего 1,5 и 4,9% соответственно [17]. Это может свидетельствовать о том, что при поиске генетических причин ХП нужно учитывать популяционные особенности и обращать на это внимание при разработке генетических тестов для обследования пациентов с данным заболеванием.
У части исследуемых нами больных ХП генетические причины заболевания не были идентифицированы (39%), что в целом согласуется с данными зарубежных коллег [24]. У больных без выявленных генетических факторов риска вполне могут присутствовать дополнительные генетические причины заболевания, связанные как с полигенным характером развития патологии, так и с моногенными заболеваниями, клинические признаки которых сопровождаются развитием воспалительных явлений в ткани ПЖ. В частности, известно, что некоторые моногенные нарушения обмена (метилмалоновая ацидурия, нарушение окисления жирных кислот и др.) характеризуются клиническими признаками ХП в детском и юношеском возрасте [25]. Между тем выявление точной генетической причины заболевания имеет крайне важное значение.
На основании генетической характеристики больных ХП уже сегодня можно сделать вывод об особенностях течения болезни, ее прогнозе и вероятности развития тех или иных осложнений, к которым прежде всего относятся экзокринная недостаточность ПЖ, сахарный диабет и рак ПЖ [26]. А с учетом данных об активном внедрении в клиническую практику ген-терапевтических препаратов и таргетной терапии, направленной на конкретные звенья молекулярного патогенеза заболевания, можно утверждать, что в будущем будут появляться новые возможности лечения больных ХП на основании особенностей их генетического профиля. Так, показано, что потенциаторы белка CFTR способны уменьшать количество приступов панкреатита у пациентов с CFTR-ассоциированным панкреатитом [27].
Заключение
С помощью разработанной генетической панели (гены PRSS1, SPINK1, CTRC, CFTR и CPA1) для выявления генетических факторов риска развития хронического панкреатита с использованием технологии массового параллельного секвенирования генетические предикторы развития заболевания выявлены у 61% пациентов. Патогенные и вероятно-патогенные варианты, ассоциированные с риском развития данного заболевания, обнаружены в генах CTRC (37,1% больных), CFTR (18,1%), SPINK1 (8,6%), PRSS1 (8,6%) и CPA1 (6,7%). Определены повторяющиеся варианты генов PRSS1, SPINK1, CTRC, CFTR и CPA1, вносящие определенный вклад в предрасположенность к развитию хронического панкреатита в выборке российских больных. Предполагается, что результаты настоящей работы найдут практическое применение в генетической диагностике и интерпретации результатов молекулярно-генетических исследований хронического панкреатита в Российской Федерации, а также послужат повышению эффективности генетического консультирования семей с хроническими заболеваниями желудочно-кишечного тракта и, в частности, с заболеваниями поджелудочной железы.
Финансирование. Исследование не финансировалось никакими источниками.
Конфликт интересов. Авторы статьи подтвердили отсутствие конфликта интересов, о котором необходимо сообщить.
Литература
- Meyer A., Coffey M.J., Oliver M.R., Ooi C.Y. Contrasts and comparisons between childhood and adult onset acute pancreatitis. Pancreatology 2013; 13(4): 429–435, https://doi.org/10.1016/j.pan.2013.06.005.
- Machicado J.D., Yadav D. Epidemiology of recurrent acute and chronic pancreatitis: similarities and differences. Dig Dis Sci 2017; 62(7): 1683–1691, https://doi.org/10.1007/s10620-017-4510-5.
- Beyer G., Habtezion A., Werner J., Lerch M.M., Mayerle J. Chronic pancreatitis. Lancet 2020; 396(10249): 499–512, https://doi.org/10.1016/s0140-6736(20)31318-0.
- Литвинова М.М., Хафизов К.Ф., Шипулин Г.А., Айгинин А.А., Винокурова Л.В., Никольская К.А., Дубцова Е.А., Бордин Д.С., Асанов А.Ю. Генетические факторы развития хронического панкреатита. Вопросы практической педиатрии 2018; 13(3): 29–40, https://doi.org/10.20953/1817-7646-2018-3-29-40.
- Whitcomb D.C.; North American Pancreatitis Study Group. Pancreatitis: TIGAR-O Version 2 Risk/Etiology Checklist with topic reviews, updates, and use primers. Clin Transl Gastroenterol 2019; 10(6): e00027, https://doi.org/10.14309/ctg.0000000000000027.
- Schmieder R., Edwards R. Quality control and preprocessing of metagenomic datasets. Bioinformatics 2011; 27(6): 863–864, https://doi.org/10.1093/bioinformatics/btr026.
- McKenna A., Hanna M., Banks E., Sivachenko A., Cibulskis K., Kernytsky A., Garimella K., Altshuler D., Gabriel S., Daly M., DePristo M.A. The genome analysis toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res 2010; 20(9): 1297–1303, https://doi.org/10.1101/gr.107524.110.
- Li H., Handsaker B., Wysoker A., Fennell T., Ruan J., Homer N., Marth G., Abecasis G., Durbin R.; 1000 Genome Project Data Processing Subgroup. The sequence alignment/map format and SAMtools. Bioinformatics 2009; 25(16): 2078–2079, https://doi.org/10.1093/bioinformatics/btp352.
- McLaren W., Gil L., Hunt S.E., Riat H.S., Ritchie G.R.S., Thormann A., Flicek P., Cunningham F. The Ensembl variant effect predictor. Genome Biol 2016; 17(1): 122, https://doi.org/10.1186/s13059-016-0974-4.
- Milne I., Stephen G., Bayer M., Cock P.J., Pritchard L., Cardle L., Shaw P.D., Marshall D. Using Tablet for visual exploration of second-generation sequencing data. Brief Bioinform 2013; 14(2): 193–202, https://doi.org/10.1093/bib/bbs012.
- R Foundation. The R project for statistical computing. Vienna; 2019. URL: https://www.R-project.org/.
- Литвинова М.М., Хафизов К.Ф., Сперанская А.С., Мацвай А.Д., Никольская К.А., Винокурова Л.В., Дубцова Е.А., Мухина Т.Ф., Хавкин А.И., Бордин Д.С. Спектр мутаций гена CFTR у больных хроническим панкреатитом в России. Вопросы детской диетологии 2020; 18(3): 5–18, https://doi.org/10.20953/1727-5784-2020-3-5-18.
- Machicado J.D., Dudekula A., Tang G., Xu H., Wu B.U., Forsmark C.E., Yadav D. Period prevalence of chronic pancreatitis diagnosis from 2001–2013 in the commercially insured population of the United States. Pancreatology 2019; 19(6): 813–818, https://doi.org/10.1016/j.pan.2019.07.003.
- Saito N., Suzuki M., Sakurai Y., Nakano S., Naritaka N., Minowa K., Sai J.K., Shimizu T. Genetic analysis of Japanese children with acute recurrent and chronic pancreatitis. J Pediatr Gastroenterol Nutr 2016; 63(4): 431–436, https://doi.org/10.1097/mpg.0000000000001320.
- Xiao Y., Yuan W., Yu B., Guo Y., Xu X., Wang X., Yu Y., Yu Y., Gong B., Xu C. Targeted gene next-generation sequencing in Chinese children with chronic pancreatitis and acute recurrent pancreatitis. J Pediatr 2017; 191: 158–163.e3, https://doi.org/10.1016/j.jpeds.2017.08.063.
- Párniczky A., Mosztbacher D., Zsoldos F., Tóth A., Lásztity N., Hegyi P.; Hungarian Pancreatic Study Group and the International Association of Pancreatology. Analysis of pediatric pancreatitis (APPLE trial): pre-study protocol of a multinational prospective clinical trial. Digestion 2016; 93(2): 105–110, https://doi.org/10.1159/000441353.
- Zou W.B., Tang X.Y., Zhou D.Z., Qian Y.Y., Hu L.H., Yu F.F., Yu D., Wu H., Deng S.J., Lin J.H., Zhao A.J., Zhao Z.H., Wu H.Y., Zhu J.H., Qian W., Wang L., Xin L., Wang M.J., Wang L.J., Fang X., He L., Masson E., Cooper D.N., Férec C., Li Z.S., Chen J.M., Liao Z. SPINK1, PRSS1, CTRC, and CFTR genotypes influence disease onset and clinical outcomes in chronic pancreatitis. Clin Transl Gastroenterol 2018; 9(11): 204, https://doi.org/10.1038/s41424-018-0069-5.
- Mayerle J., Sendler M., Hegyi E., Beyer G., Lerch M.M., Sahin-Tóth M. Genetics, cell biology, and pathophysiology of pancreatitis. Gastroenterology 2019; 156(7): 1951–1968.e1, https://doi.org/10.1053/j.gastro.2018.11.081.
- LaRusch J., Lozano-Leon A., Stello K., Moore A., Muddana V., O’Connell M., Diergaarde B., Yadav D., Whitcomb D.C. The common chymotrypsinogen C (CTRC) variant G60G (C.180T) increases risk of chronic pancreatitis but not recurrent acute pancreatitis in a North American population. Clin Transl Gastroenterol 2015; 6(1): e68, https://doi.org/10.1038/ctg.2014.13.
- Rosendahl J., Witt H., Szmola R., Bhatia E., Ozsvári B., Landt O., Schulz H.U., Gress T.M., Pfützer R., Löhr M., Kovacs P., Blüher M., Stumvoll M., Choudhuri G., Hegyi P., te Morsche R.H.M., Drenth J.P., Truninger K., Macek M. Jr., Puhl G., Witt U., Schmidt H., Büning C., Ockenga J., Kage A., Groneberg D.A., Nickel R., Berg T., Wiedenmann B., Bödeker H., Keim V., Mössner J., Teich N., Sahin-Tóth M. Chymotrypsin C (CTRC) variants that diminish activity or secretion are associated with chronic pancreatitis. Nat Genet 2008; 40(1): 78–82, https://doi.org/10.1038/ng.2007.44.
- Masamune A., Kikuta K., Hamada S., Nakano E., Kume K., Inui A., Shimizu T., Takeyama Y., Nio M., Shimosegawa T. Nationwide survey of hereditary pancreatitis in Japan. J Gastroenterol 2018; 53(1): 152–160, https://doi.org/10.1007/s00535-017-1388-0.
- Németh B.C., Demcsák A., Geisz A., Sahin-Tóth M. Misfolding-induced chronic pancreatitis in CPA1 N256K mutant mice is unaffected by global deletion of Ddit3/Chop. Sci Rep 2022; 12(1): 6357, https://doi.org/10.1038/s41598-022-09595-x.
- Masson E., Chen J.M., Audrézet M.P., Cooper D.N., Férec C. A conservative assessment of the major genetic causes of idiopathic chronic pancreatitis: data from a comprehensive analysis of PRSS1, SPINK1, CTRC and CFTR genes in 253 young French patients. PLoS One 2013; 8(8): e73522, https://doi.org/10.1371/journal.pone.0073522.
- Masamune A. Genetics of pancreatitis: the 2014 update. Tohoku J Exp Med 2014; 232(2): 69–77, https://doi.org/10.1620/tjem.232.69.
- Hwang W.J., Lim H.H., Kim Y.M., Chang M.Y., Kil H.R., Kim J.Y., Song W.J., Levy H.L., Kim S.Z. Pancreatic involvement in patients with inborn errors of metabolism. Orphanet J Rare Dis 2021; 16(1): 37, https://doi.org/10.1186/s13023-021-01685-9.
- Suzuki M., Minowa K., Nakano S., Isayama H., Shimizu T. Genetic abnormalities in pancreatitis: an update on diagnosis, clinical features, and treatment. Diagnostics (Basel) 2020; 11(1): 31, https://doi.org/10.3390/diagnostics11010031.
- Carrion A., Borowitz D.S., Freedman S.D., Siracusa C.M., Goralski J.L., Hadjiliadis D., Srinivasan S., Stokes D.C. Reduction of recurrence risk of pancreatitis in cystic fibrosis with ivacaftor: case series. J Pediatr Gastroenterol Nutr 2018; 66(3): 451–454, https://doi.org/10.1097/mpg.0000000000001788.










