Новый кофилин-зависимый механизм регуляции биогенеза и деградации митохондрий головного мозга
Цель исследования — изучение роли посттрансляционных модификаций кофилина в регуляции дыхания и аутофагии митохондрий головного мозга мышей.
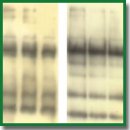
Материалы и методы. Эксперименты выполнены на мышах линии C57BL/6. Для получения цитоплазматической и митохондриальной фракций мозга использовали метод дифференциального центрифугирования. Анализ уровня экспрессии кофилина, фосфокофилина, К48- и К63-ассоциированных цепей убиквитина, маркера аутофагии LC3B выполняли с помощью методов электрофореза, иммунопреципитации и вестерн-блота. Для исследования процессов убиквитилирования использовали ингибитор деубиквитилирующих ферментов PR619. Активность дыхания митохондрий головного мозга оценивали с помощью метода высокоразрешающей флуороспирометрии.
Результаты. Показана модификация кофилина неканоническими К63-мультиубиквитиновыми цепями в цитоплазме и митохондриях головного мозга. Выявлен разный уровень экспрессии фосфокофилина, кофилина и его убиквитилированных протеоформ. Ингибитор деубиквитилирующих ферментов PR619 влияет на уровень фосфорилированных и убиквитилированных форм кофилина в митохондриях и цитоплазме, при этом установлено изменение активности тканевого дыхания и митофагии.
Заключение. Чувствительность кофилина к ингибитору деубиквитилирующих ферментов свидетельствует о существовании в клетках нового некатаболического механизма регуляции данного белка, который может быть вовлечен в регуляцию функций митохондрий, что проявляется в изменении активности дыхания и аутофагии митохондрий головного мозга. Полученные данные вносят вклад в понимание молекулярных механизмов функционирования митохондрий в норме и при патологии для разработки эффективных методов терапии заболеваний нервной системы.
- Ding W.X., Yin X.M. Mitophagy: mechanisms, pathophysiological roles, and analysis. J Biol Chem 2012; 393(7): 547–564, https://doi.org/10.1515/hsz-2012-0119.
- Kubli D.A., Gustafsson A.B. Mitochondria and mitophagy: the yin and yang of cell death control. Circ Res 2012; 111(9): 1208–1221, https://doi.org/10.1161/circresaha.112.265819.
- Suzuki S.W., Onodera J., Ohsumi Y. Starvation induced cell death in autophagy-defective yeast mutants is caused by mitochondria dysfunction. PLoS One 2011; 6(2): e17412, https://doi.org/10.1371/journal.pone.0017412.
- Al-Hakim A., Escribano-Diaz C., Landry M.C., O’Donnell L., Panier S., Szilard R.K., Durocher D. The ubiquitous role of ubiquitin in the DNA damage response. DNA Repair (Amst) 2010; 9(12): 1229–1240, https://doi.org/10.1016/j.dnarep.2010.09.011.
- Popovic D., Vucic D., Dikic I. Ubiquitination in disease pathogenesis and treatment. Nat Med 2014; 20(11): 1242–1253, https://doi.org/10.1038/nm.3739.
- Bragoszewski P., Turek M., Chacinska A. Control of mitochondrial biogenesis and function by the ubiquitin-proteasome system. Open Biol 2017; 7(4): 170007, https://doi.org/10.1098/rsob.170007.
- Ashrafi G., Schlehe J.S., LaVoie M.J., Schwarz T.L. Mitophagy of damaged mitochondria occurs locally in distal neuronal axons and requires PINK1 and Parkin. J Cell Biol 2014; 206(5): 655–670, https://doi.org/10.1083/jcb.201401070.
- Kim K.H., Son J.H. PINK1 gene knockdown leads to increased binding of parkin with actin filament. Neurosci Lett 2010; 468(3): 272–276, https://doi.org/10.1016/j.neulet.2009.11.011.
- Kanellos G., Frame M.C. Cellular functions of the ADF/cofilin family at a glance. J Cell Sci 2016; 129: 3211–3218, https://doi.org/10.1242/jcs.187849.
- Kovaleva T.F., Maksimova N.S., Zhukov I.Y., Pershin V.I., Mukhina I.V., Gainullin M.R. Cofilin: molecular and cellular functions and its role in the functioning of the nervous system. Neurochem J 2019; 13 (1): 11–19, https://doi.org/10.1134/s1819712419010124.
- Kotiadis V.N., Leadsham J.E., Bastow E.L., Gheeraert A., Whybrew J.M., Bard M., Lappalainen P., Gourlay C.W. Identification of new surfaces of cofilin that link mitochondrial function to the control of multi-drug resistance. J Cell Sci 2012; 125(Pt 9): 2288–2299, https://doi.org/10.1242/jcs.099390.
- Palikaras K., Lionaki E., Tavernarakis N. Balancing mitochondrial biogenesis and mitophagy to maintain energy metabolism homeostasis. Cell Death Differ 2015; 22: 1399–1401, https://doi.org/10.1038/cdd.2015.86.
- Li G.B., Zhang H.W., Fu R.Q., Hu X.Y., Liu L., Li Y.N., Liu Y.X., Liu X., Hu J.J., Deng Q., Luo Q.S., Zhang R., Gao N. Mitochondrial fission and mitophagy depend on cofilin-mediated actin depolymerization activity at the mitochondrial fission site. Oncogene 2018; 37(11): 1485–1502, https://doi.org/10.1038/s41388-017-0064-4.
- Madineni A. Role of cofilin, an actin cytoskeletal protein, in ischemic conditions: potential therapeutic target for ischemic stroke [dissertation]. University of Toledo Health Science Campus; 2013.
- Rehklau K., Gurniak C.B., Conrad M., Friauf E., Ott M., Rust M.B. ADF/cofilin proteins translocate to mitochondria during apoptosis but are not generally required for cell death signaling. Cell Death Differ 2012; 19(6): 958–967, https://doi.org/10.1038/cdd.2011.180.
- Gainullin M.R., Zhukov I.Y., Zhou X., Mo Y., Astakhova L., Ernberg I., Matskova L. Degradation of cofilin is regulated by Cbl, AIP4 and Syk resulting in increased migration of LMP2A positive nasopharyngeal carcinoma cells. Sci Rep 2017; 7(1): 9012, https://doi.org/10.1038/s41598-017-09540-3.
- Myeku N., Figueiredo-Pereira M.E. Dynamics of the degradation of ubiquitinated proteins by proteasomes and autophagy: association with sequestosome 1/p62. J Biol Chem 2011; 286(25): 22426–22440, https://doi.org/10.1074/jbc.m110.149252.
- Komander D., Clague M.J., Urbé S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol 2009; 10(8): 550–563, https://doi.org/10.1038/nrm2731.
- Swatek K.N., Komander D. Ubiquitin modifications. Cell Res 2016; 26(4): 399–422, https://doi.org/10.1038/cr.2016.39.
- Seiberlich V., Borchert J., Zhukareva V., Richter-Landsberg C. Inhibition of protein deubiquitination by PR-619 activates the autophagic pathway in OLN-t40 oligodendroglial cells. Cell Biochem Biophys 2013; 67(1): 149–160, https://doi.org/10.1007/s12013-013-9622-8.
- Pershin V., Maksimova N., Mukhina I., Gainullin M., Yudintsev A., Sergeeva T. Effect of the ubiquitin-proteasome system inhibitors on the energy metabolism of nerve cells. FEBS Open Bio 2018; 8 (Suppl S1): 291.










